Похожие презентации:
Биотехнология. Вектора. Метод Максама—Гилберта
1. Биотехнология 1
2.
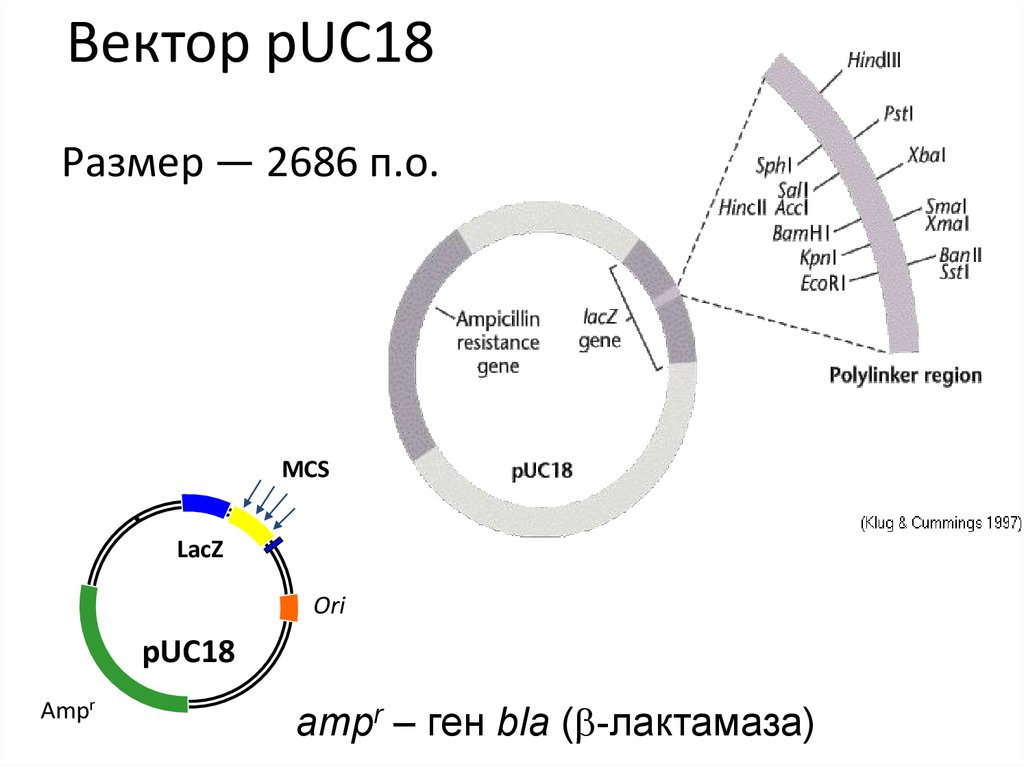
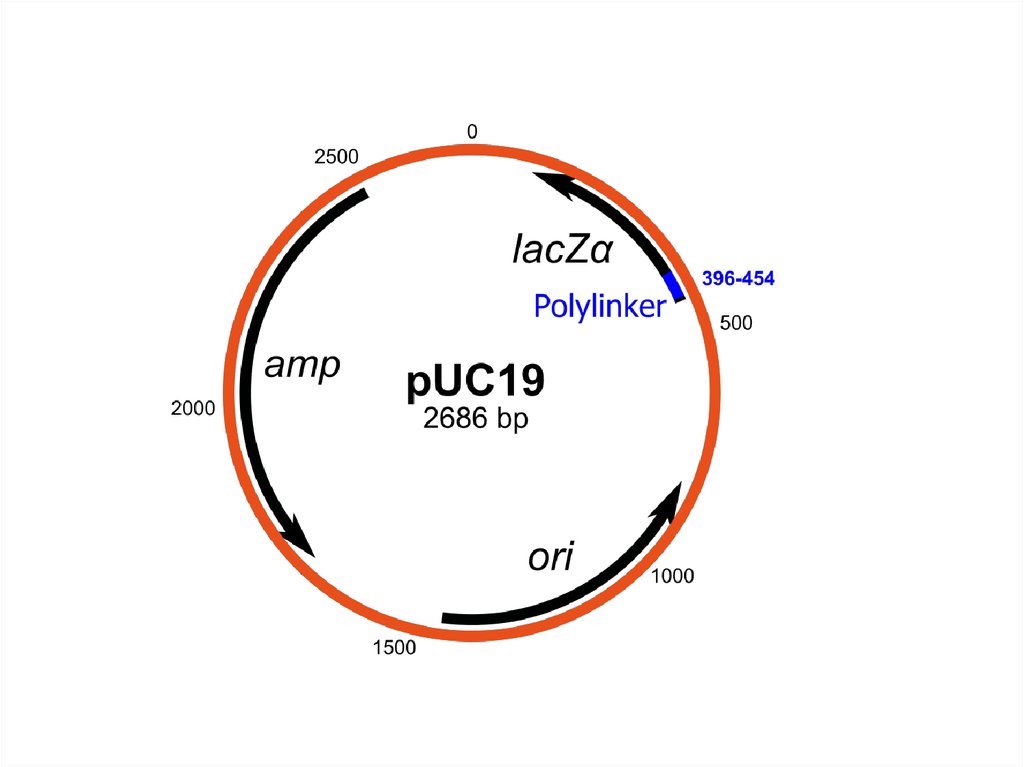
Вектор pUC18Размер — 2686 п.о.
MCS
LacZ
Ori
pUC18
Ampr
ampr – ген bla (b-лактамаза)
3.
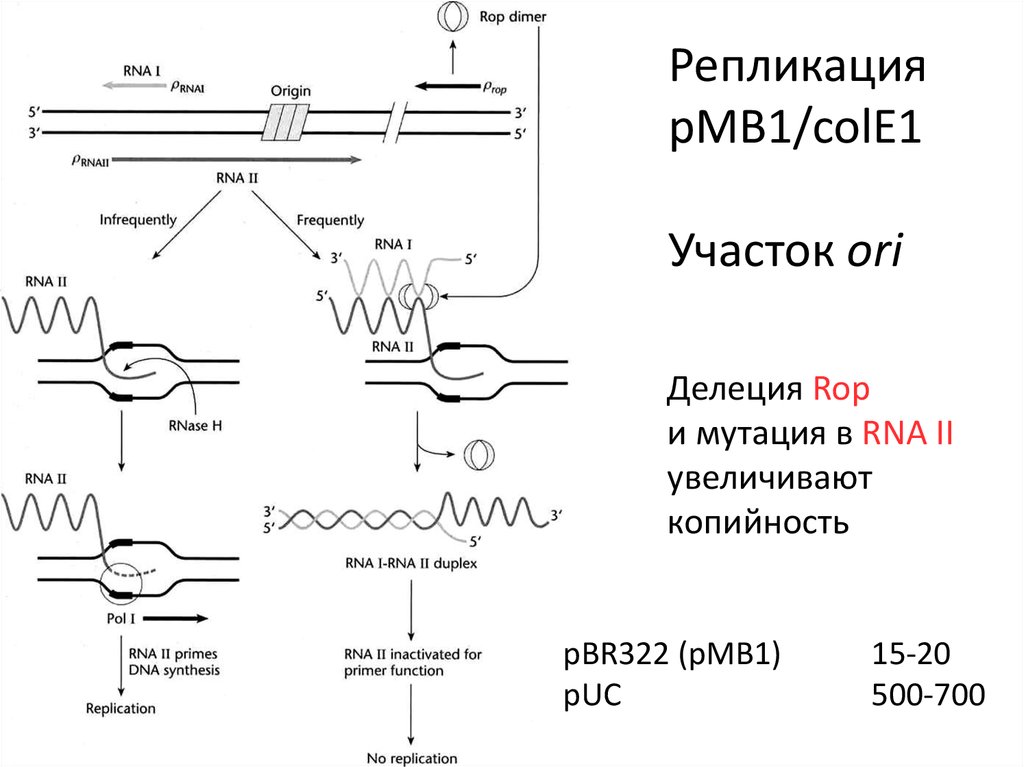
РепликацияpMB1/colE1
Участок ori
Делеция Rop
и мутация в RNA II
увеличивают
копийность
pBR322 (pMB1)
pUC
15-20
500-700
4.
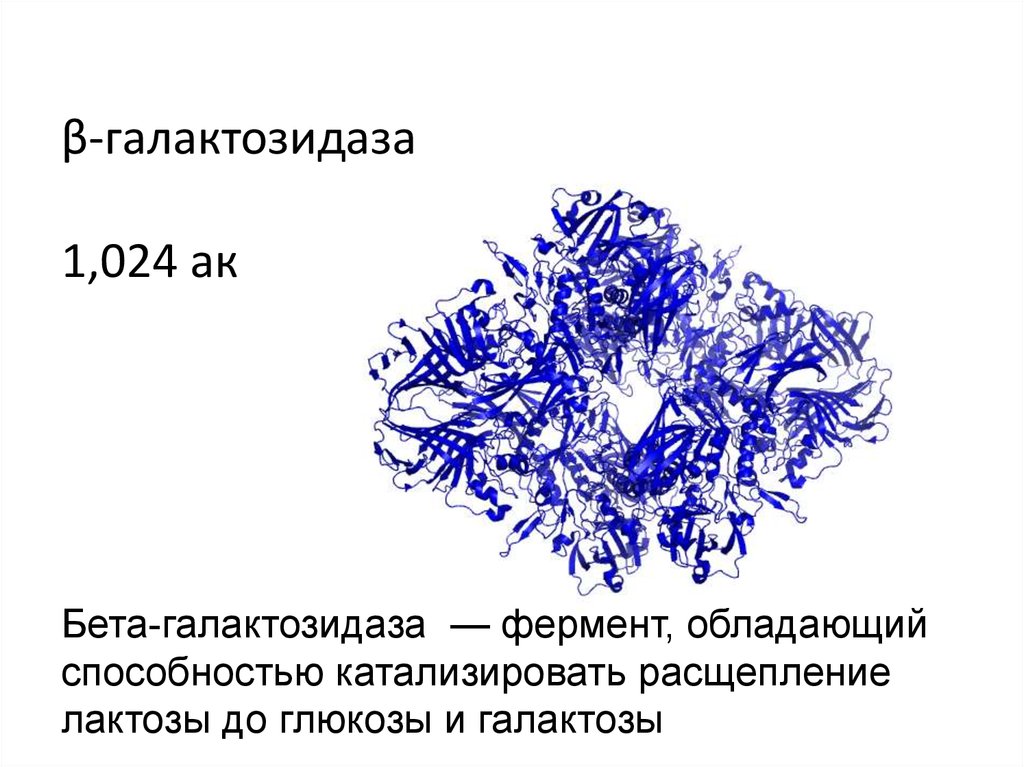
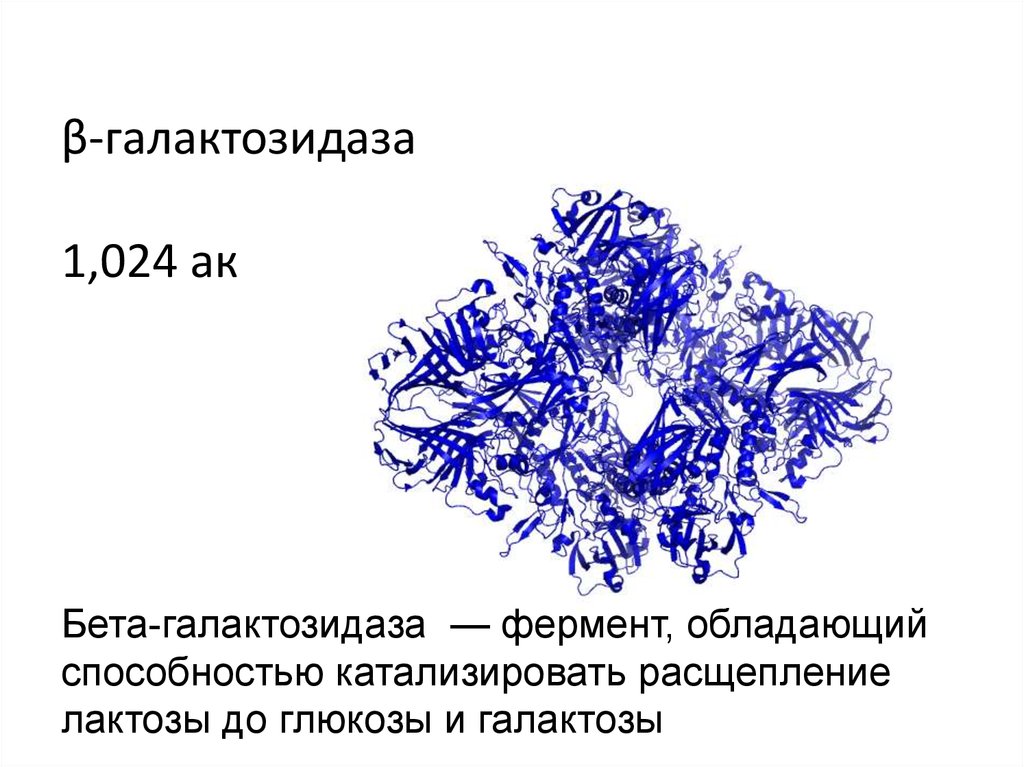
β-галактозидаза1,024 ак
Бета-галактозидаза — фермент, обладающий
способностью катализировать расщепление
лактозы до глюкозы и галактозы
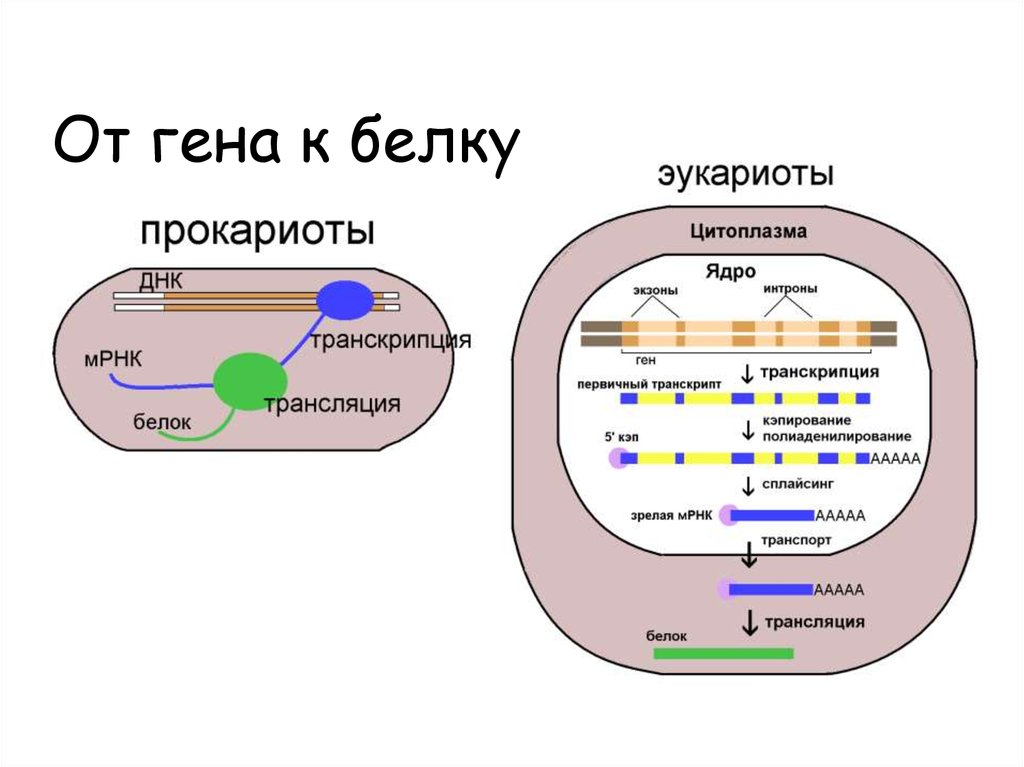
5. От гена к белку
6. РНК считывается с одной цепи ДНК
Считываемая цепь ДНК называется матричной илинекодирующей.
РНК
3'
5'
5’ AUGCUAU
TACGATA
РНК
некодирующая цепь
UAGCUGU
ATCGACA
TAGCTGT
ATGCTAT
кодирующая цепь
Противоположная цепь называется кодирующей
Для каждого гена одна из цепей ДНК кодирующая.
Разные гены могут считываться с противоположных
цепей ДНК
5'
3'
7. Синтез РНК РНК-полимеразой
5’-3’ направление синтеза
затравка не нужна
РНК-полимераза сама расплетает ДНК
Синтез РНК происходит процессивно
Во время синтеза только часть молекулы
РНК спарена с матрицей
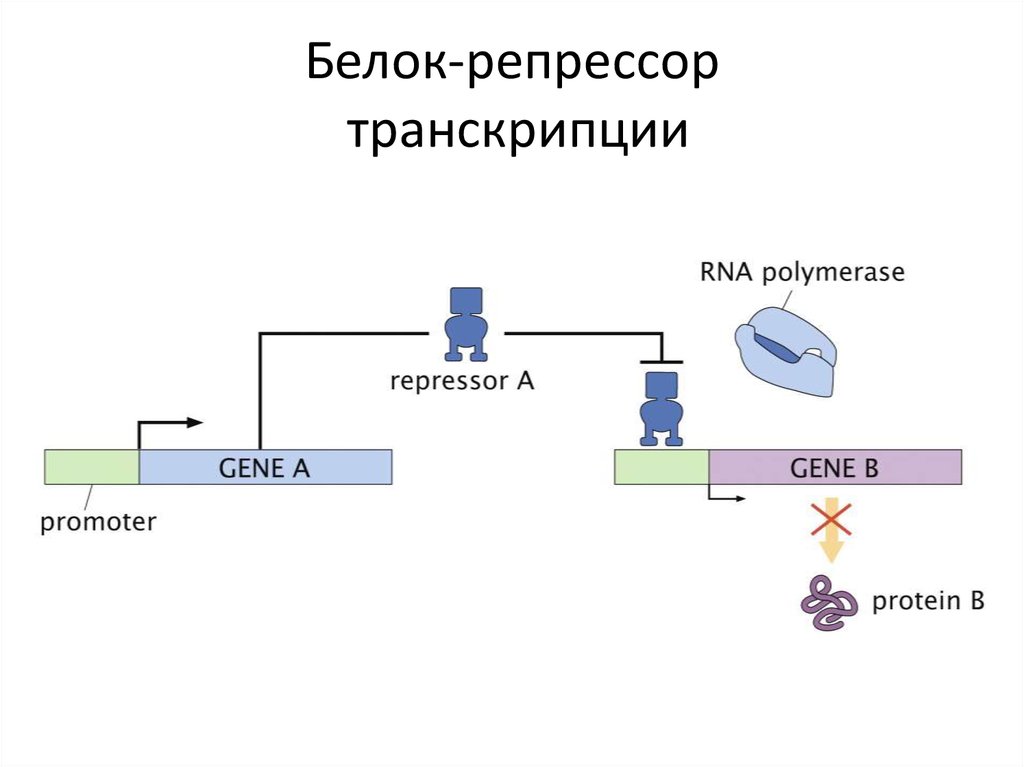
8. Белок-репрессор транскрипции
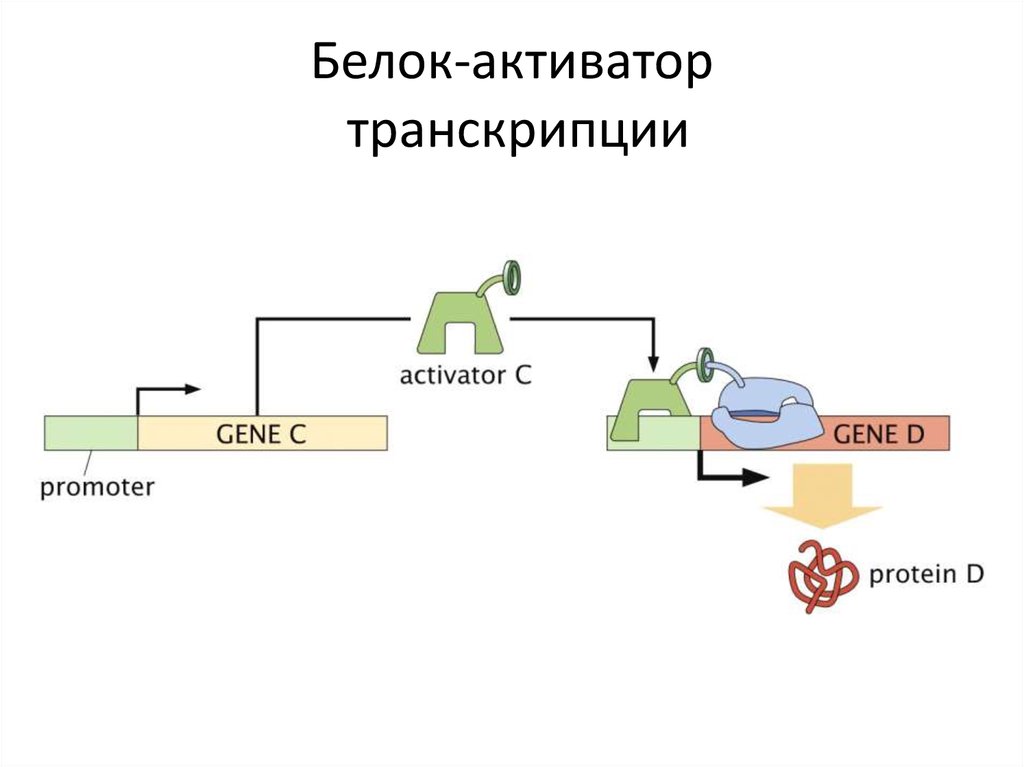
9. Белок-активатор транскрипции
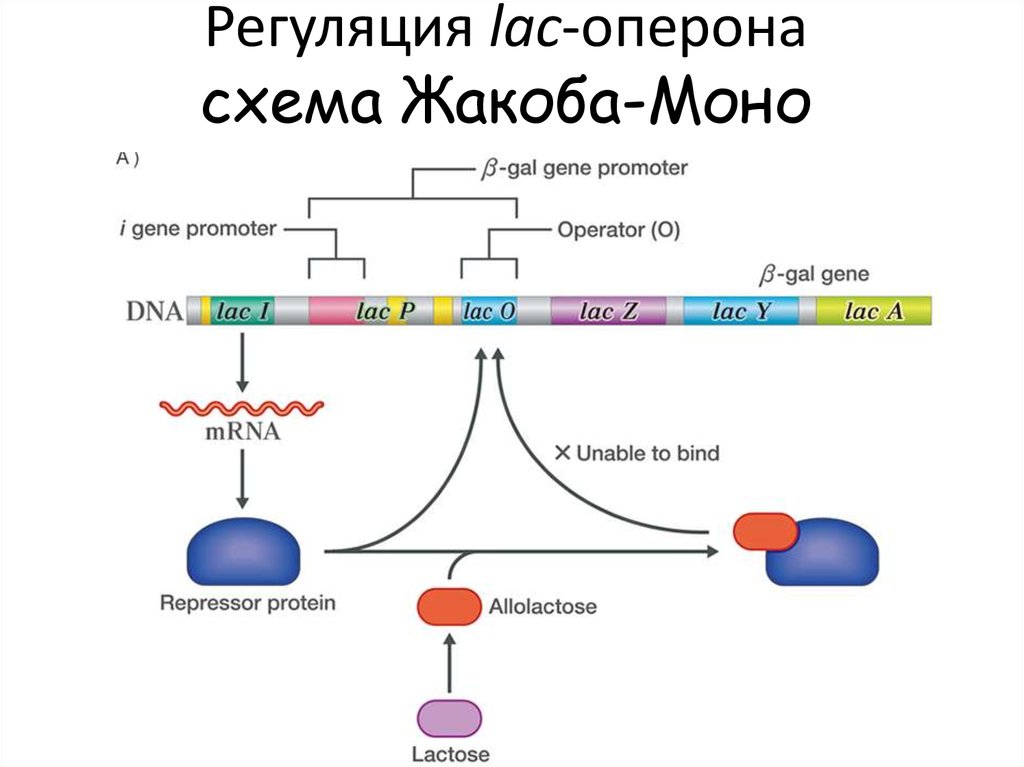
10. Гены lac-оперона
lacZ кодирует фермент β-галактозидазу, котораярасщепляет дисахарид лактозу
на глюкозу и галактозу
lacY кодирует β-галактозид пермеазу, мембранный
транспортный белок, который переносит лактозу
внутрь клетки
lacA кодирует β-галактозид трансацетилазу, фермент,
переносящий ацетильную группу от ацетилКoA на бета-галактозиды
Для катаболизма лактозы необходимы только продукты
генов lacZ и lacY, роль продукта гена lacA не ясна.
11.
Регуляция lac-оперонасхема Жакоба-Моно
12.
Регуляция lac-оперона13.
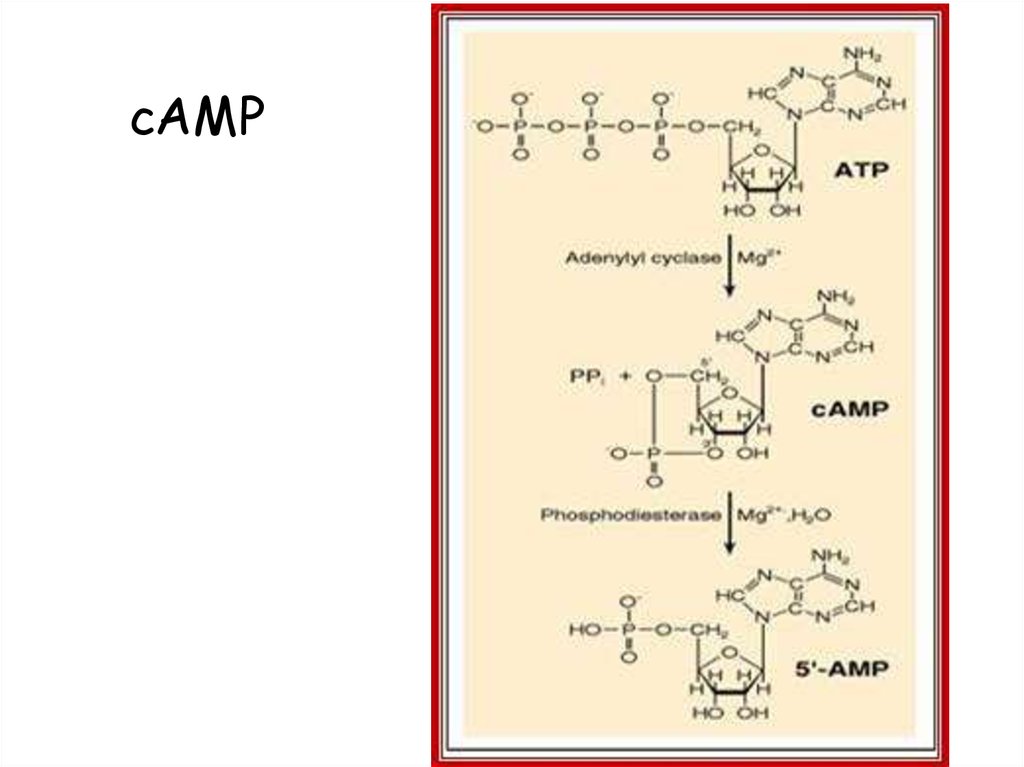
Регуляция lac-оперона14. cAMP
15. Lac-оперон
ЛактозаГлюкоза
-
+
-
CAP-cAMP
+
-
+
+
репрессор
фф
16.
β-галактозидаза1,024 ак
Бета-галактозидаза — фермент, обладающий
способностью катализировать расщепление
лактозы до глюкозы и галактозы
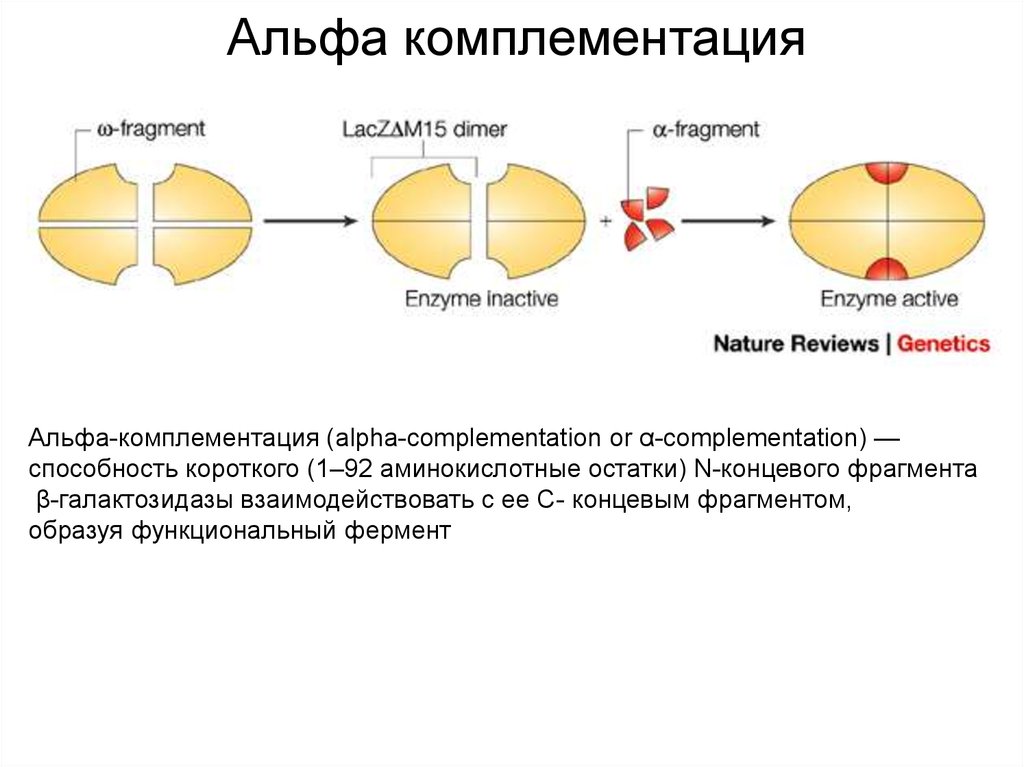
17. Альфа комплементация
Альфа-комплементация (аlpha-complementation or α-complementation) —способность короткого (1–92 аминокислотные остатки) N-концевого фрагмента
β-галактозидазы взаимодействовать с ее С- концевым фрагментом,
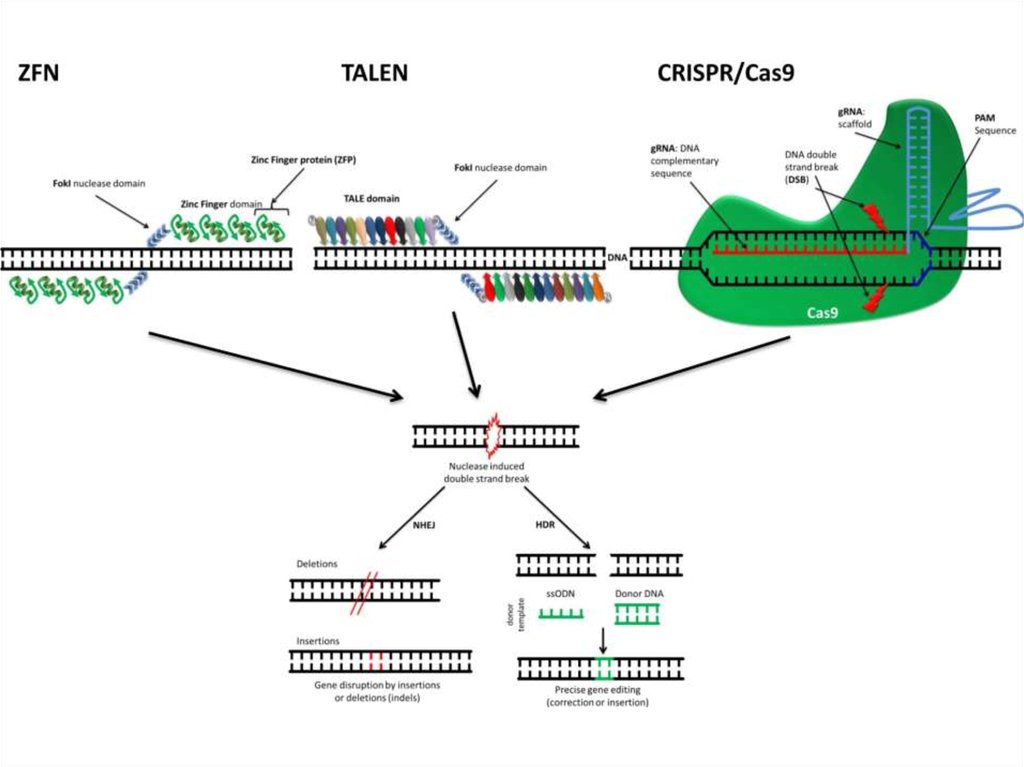
образуя функциональный фермент
18.
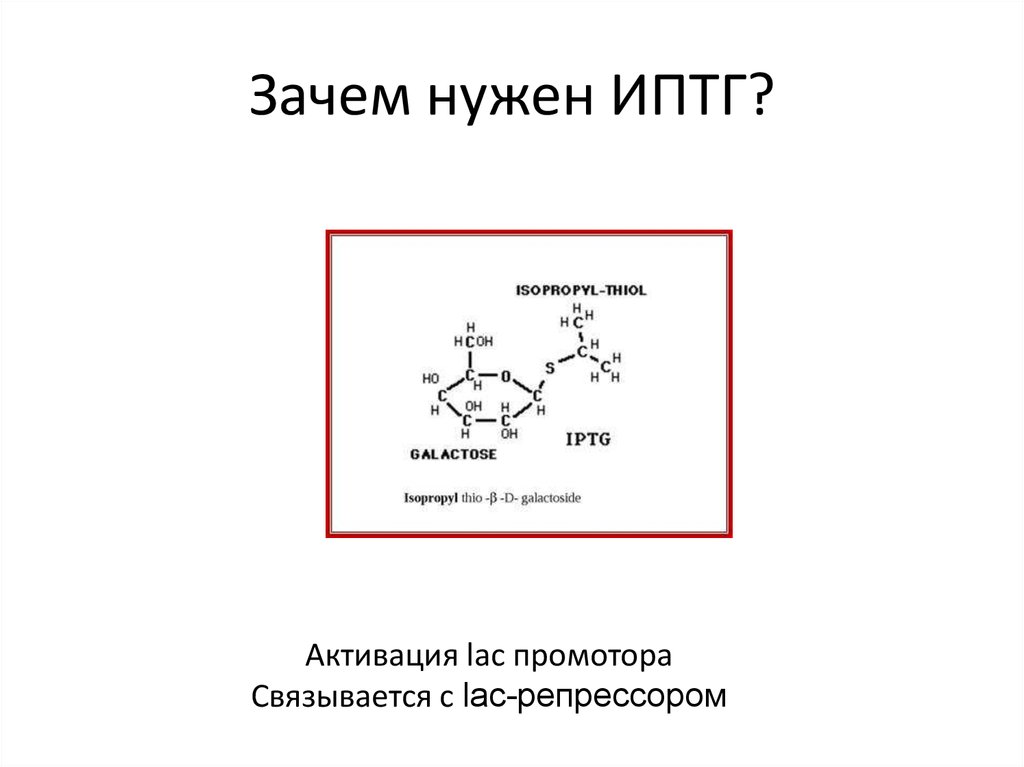
Субстрат Xgal (5-бром-4-хлор-3-индолил-бета-D-галактопиранозид)19. Зачем нужен ИПТГ?
Активация lac промотораСвязывается с lac-репрессором
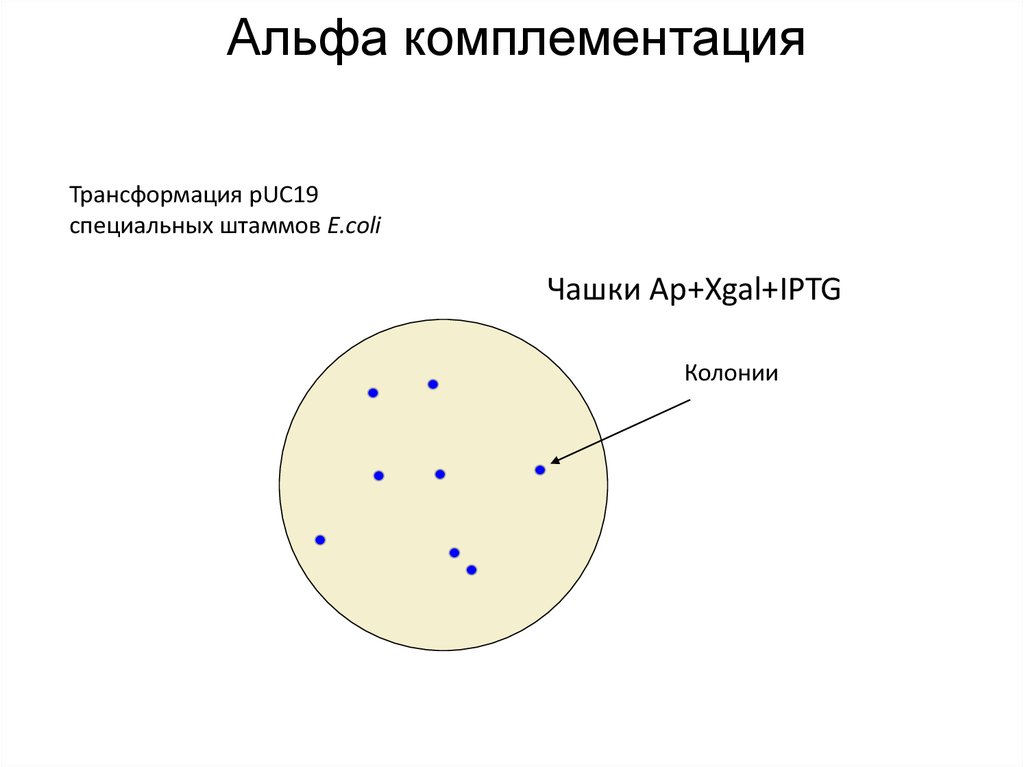
20. Альфа комплементация
Трансформация pUC19специальных штаммов E.coli
Чашки Ap+Xgal+IPTG
Колонии
21.
22.
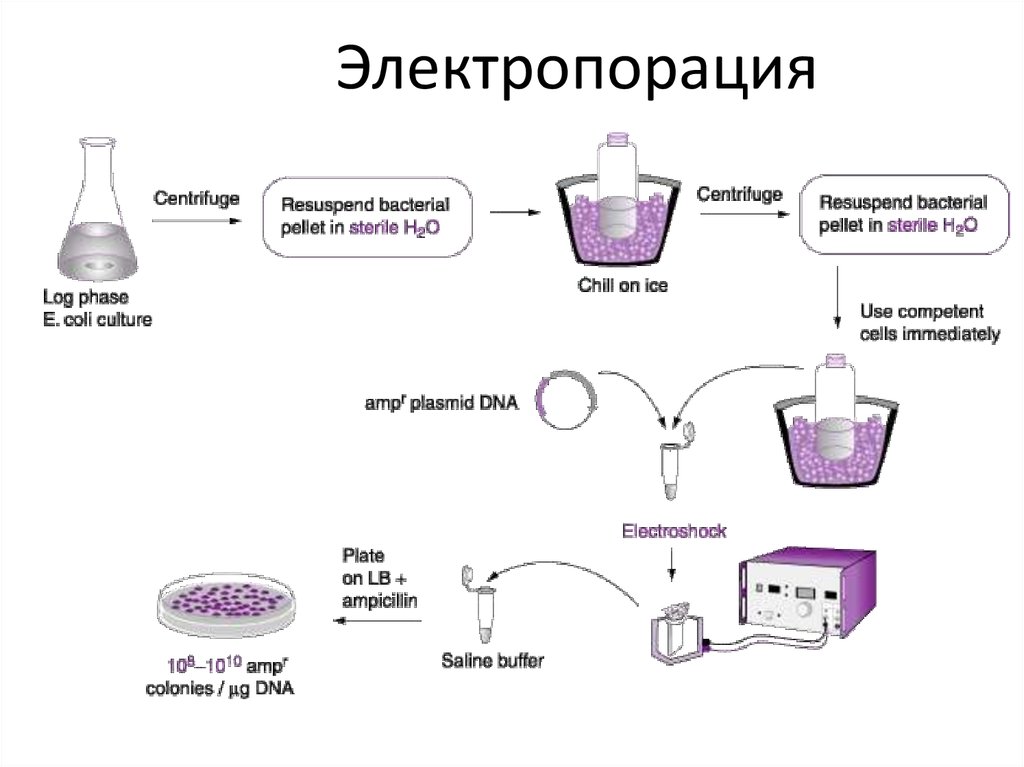
Трансформация23.
Электропорация24.
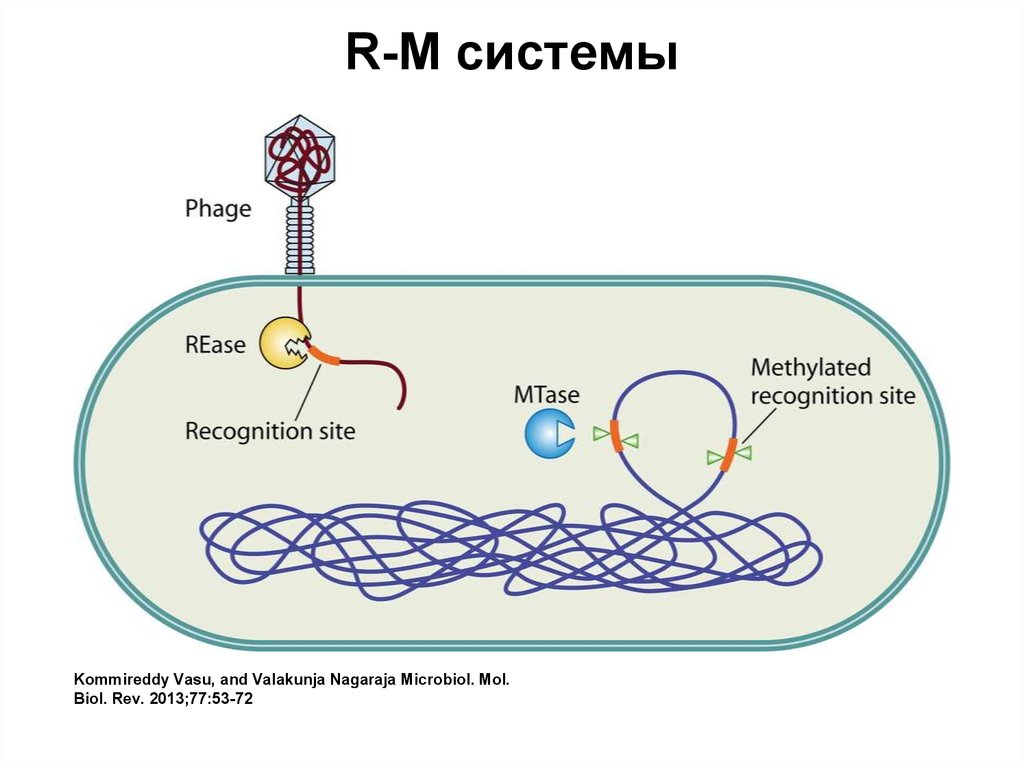
R-M системыKommireddy Vasu, and Valakunja Nagaraja Microbiol. Mol.
Biol. Rev. 2013;77:53-72
25.
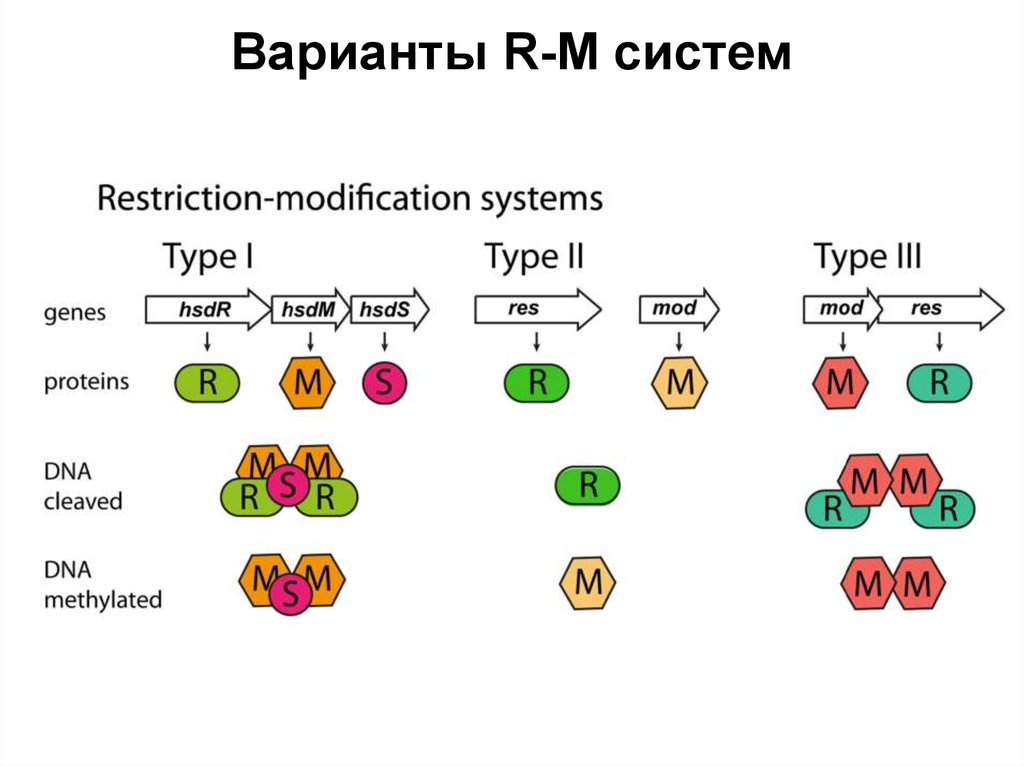
Варианты R-M систем26. Рестриктазы
Размер сайтаЧастота
4 bp
44 =256 bp
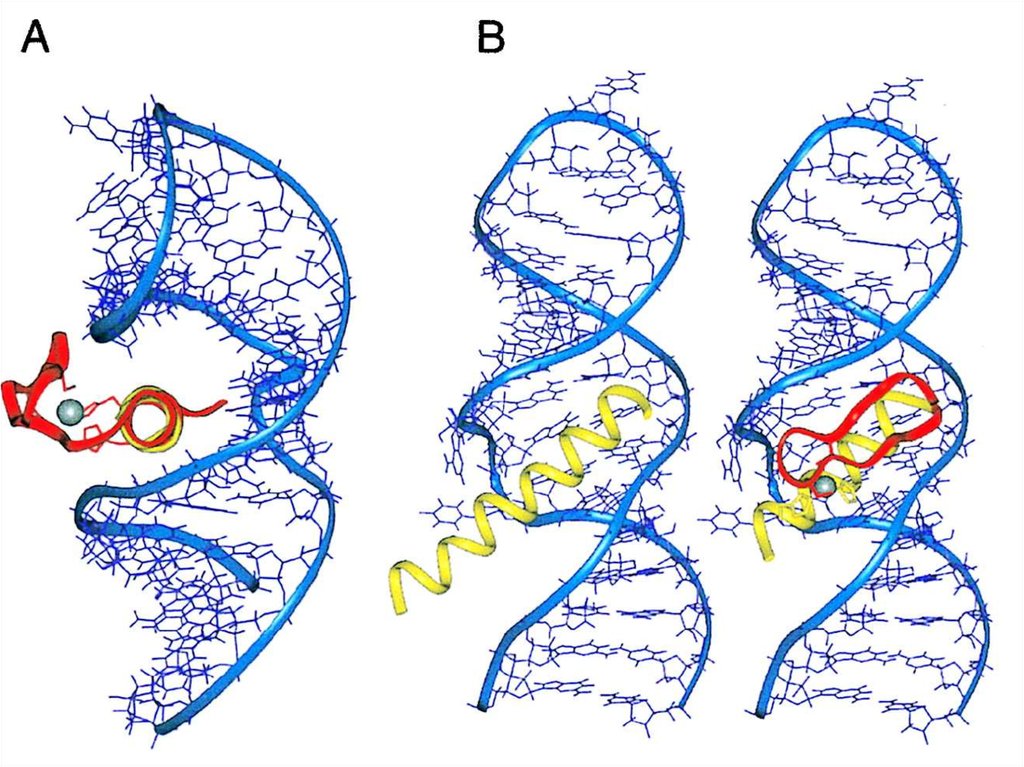
6 bp
46=4096 bp
8 bp
48=65 536 bp
27. Рестриктаза EcoRI
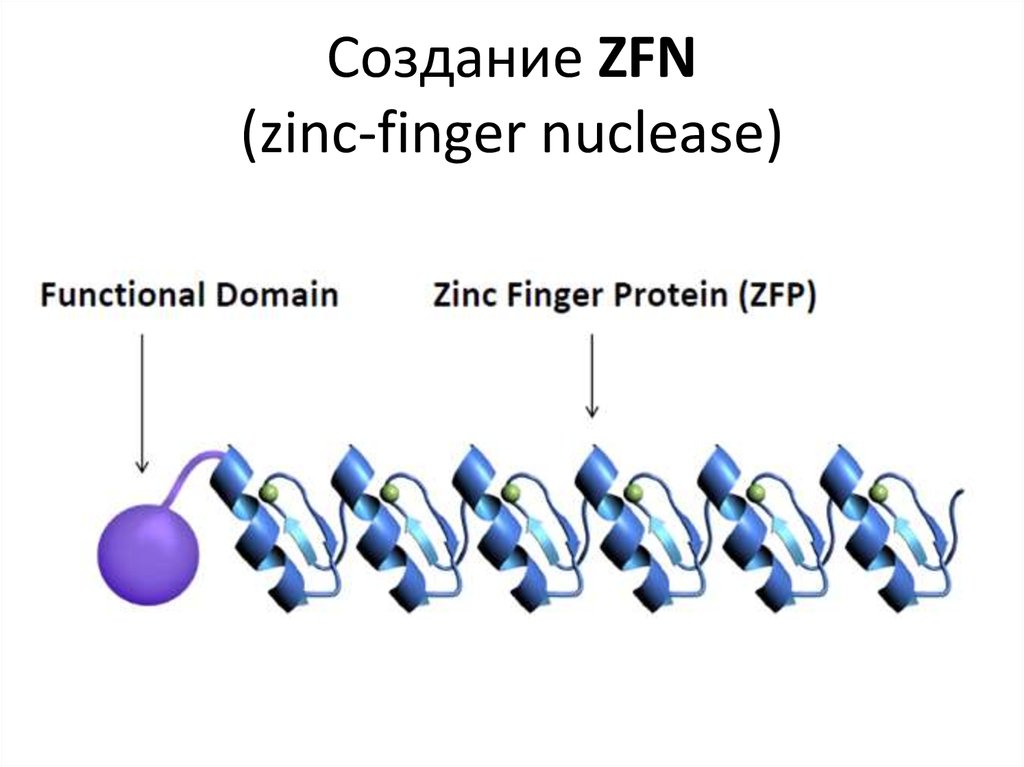
EcoRI5´ ... G^A A T T C ... 3´
3´ ... C T T A A^G ... 5´
EcoRI
5´ ... G^ 3’
5’ A A T T C ... 3´
3´ ... C T T A A 5’
3’ ^G ... 5´
28. X. Смит и Д. Натане в 1973 г. предложили номенклатуру рестриктаз Genus+SPecies (strain)
EcoRI –
BamHI –
DpnI –
HindIII –
BglII –
PstI –
Sau3AI –
KpnI –
Escherichia coli strain R, 1st enzyme
Bacillus amyloliquefaciens strain H, 1st enzyme
Diplococcus pneumoniae, 1st enzyme
Haemophilus influenzae, strain D, 3rd enzyme
Bacillus globigii, 2nd enzyme
Providencia stuartii 164, 1st enzyme
Staphylococcus aureus strain 3A, 1st enzyme
Klebsiella pneumoniae, 1st enzyme
29.
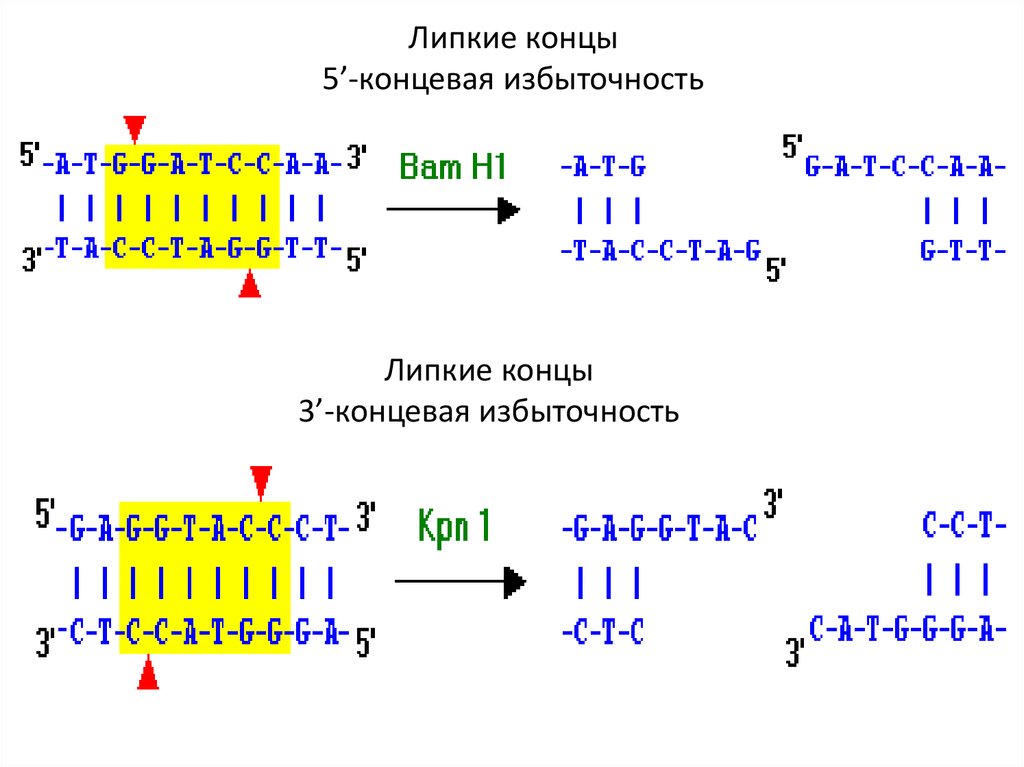
Липкие концы5’-концевая избыточность
Липкие концы
3’-концевая избыточность
30.
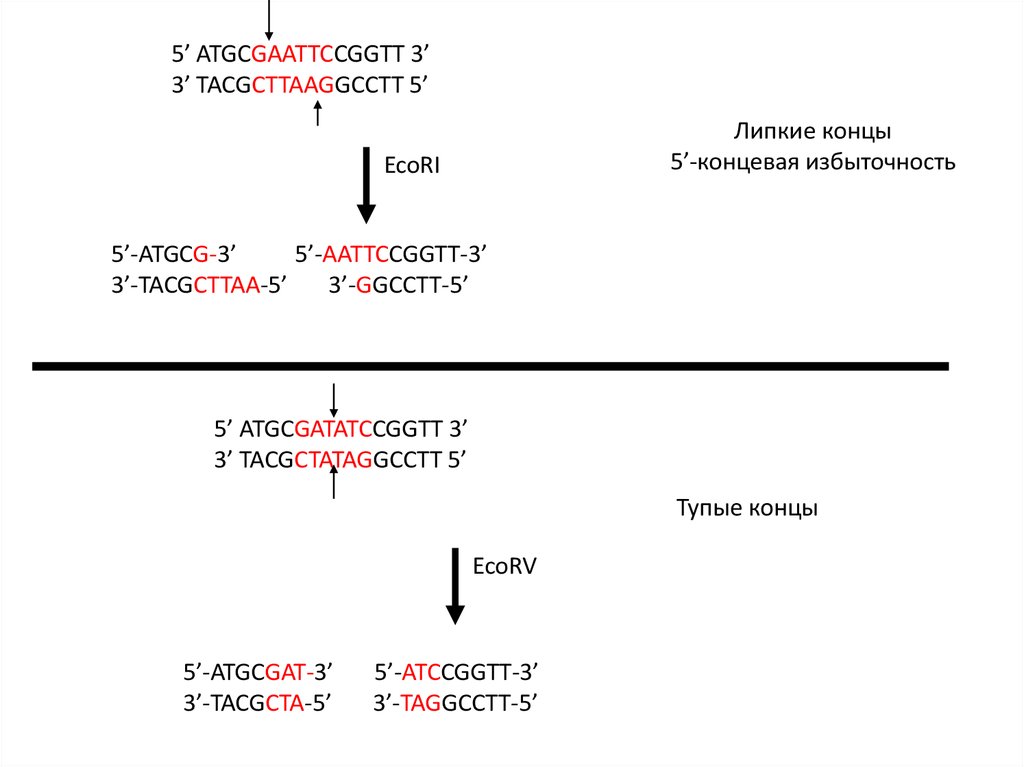
5’ ATGCGAATTCCGGTT 3’3’ TACGCTTAAGGCCTT 5’
Липкие концы
5’-концевая избыточность
EcoRI
5’-ATGCG-3’
5’-AATTCCGGTT-3’
3’-TACGCTTAA-5’
3’-GGCCTT-5’
5’ ATGCGATATCCGGTT 3’
3’ TACGCTATAGGCCTT 5’
Тупые концы
EcoRV
5’-ATGCGAT-3’
3’-TACGCTA-5’
5’-ATCCGGTT-3’
3’-TAGGCCTT-5’
31. Рестриктазы
4:AluI
5´ ... AG^CT ... 3´
тупые
MspI
5´ ... C^CGG ... 3´
5’ концевая избыточность (2 bp)
PvuII
5´ ... CAG^CTG ... 3´
тупые
KpnI
5´ ... GGTAC^C ... 3´
3’ концевая избыточность (4 bp)
NotI
5´ ... GC^GGCCGC ... 3´
5’ концевая избыточность (4 bp)
6
8
Необычные сайты
MwoI 5´ ...
GCNNNNN^NNGC
... 3´
3’ концевая избыточность
3´ ...
CGNN^NNNNNCG
... 5´
(3 bp)
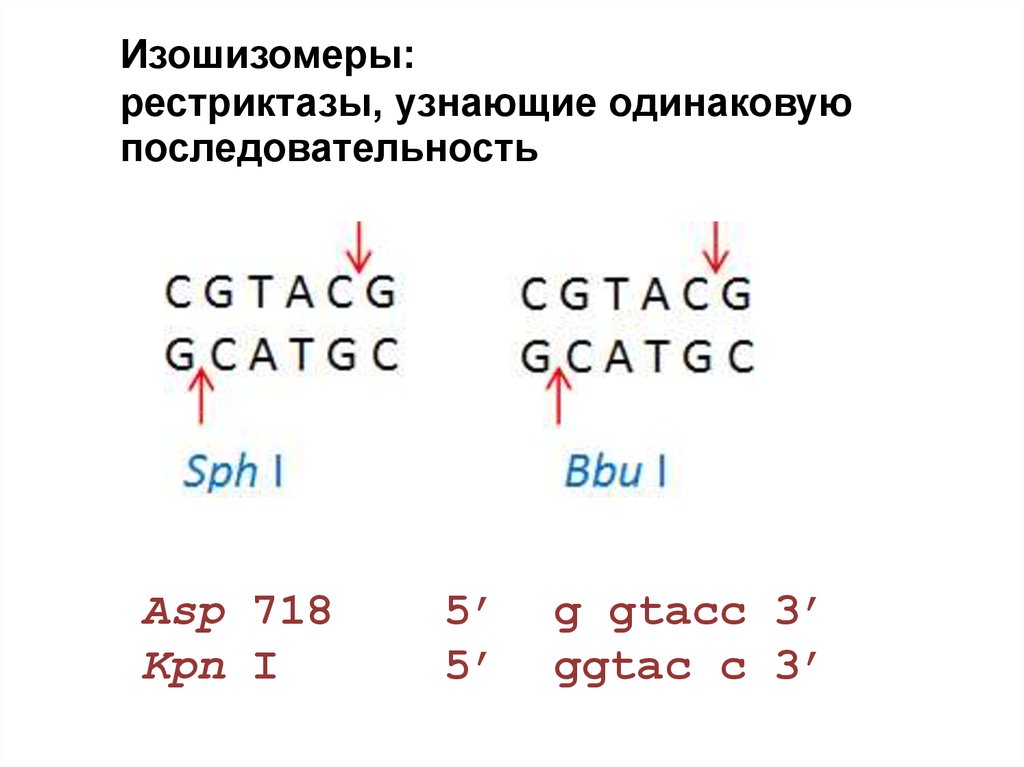
32.
Изошизомеры:рестриктазы, узнающие одинаковую
последовательность
Asp 718
Kpn I
5’
5’
g gtacc 3’
ggtac c 3’
33. Картирование 1
34. Картирование 2
35. Картирование 3
36.
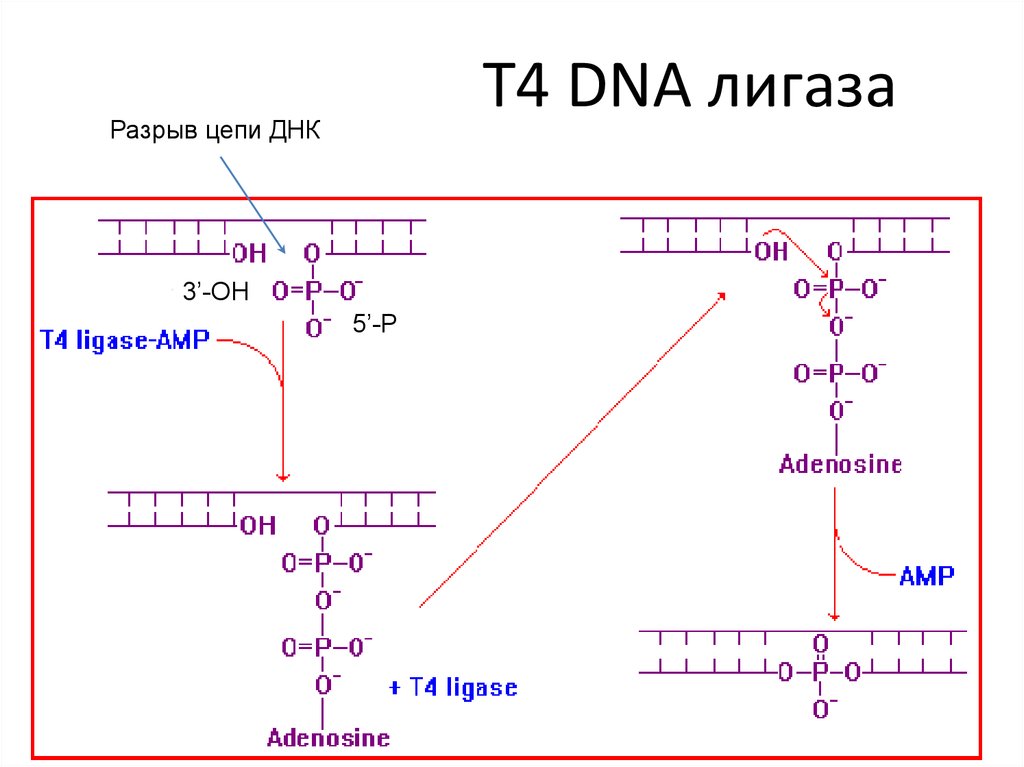
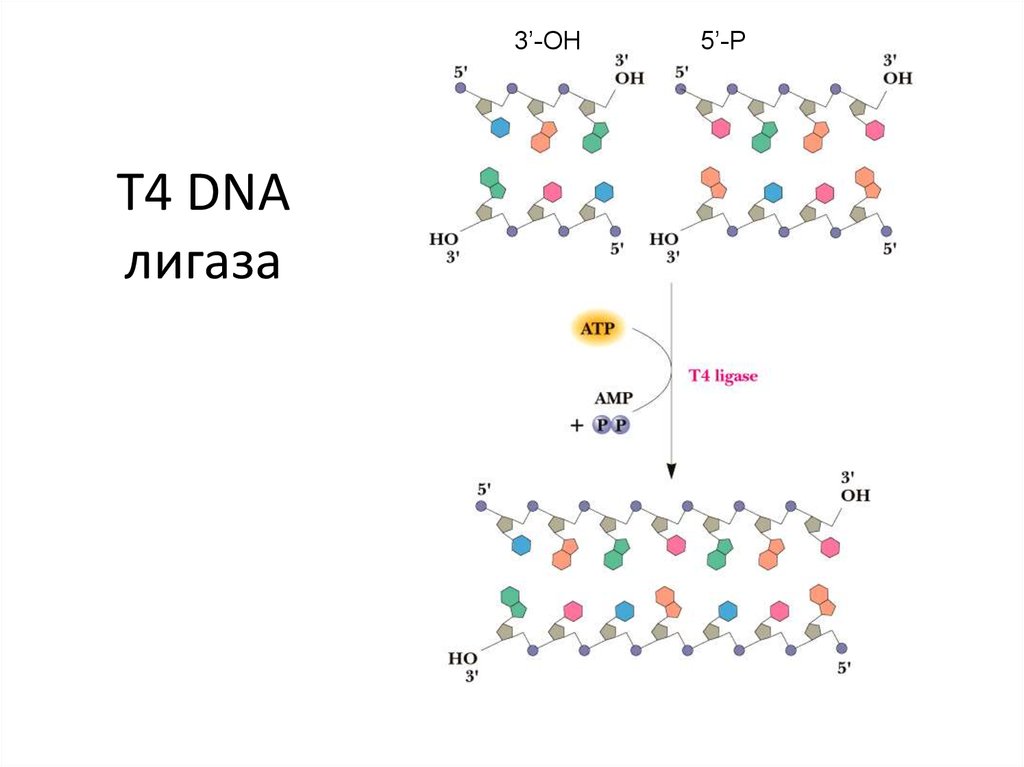
37. T4 DNA лигаза
Разрыв цепи ДНК3’-OH
5’-P
38.
5’-P3’-OH
39. T4 DNA лигаза
3’-OHT4 DNA
лигаза
5’-P
40.
+ДНК-лигаза
41.
X-galBright blue
Бело-голубая
селекция клонов
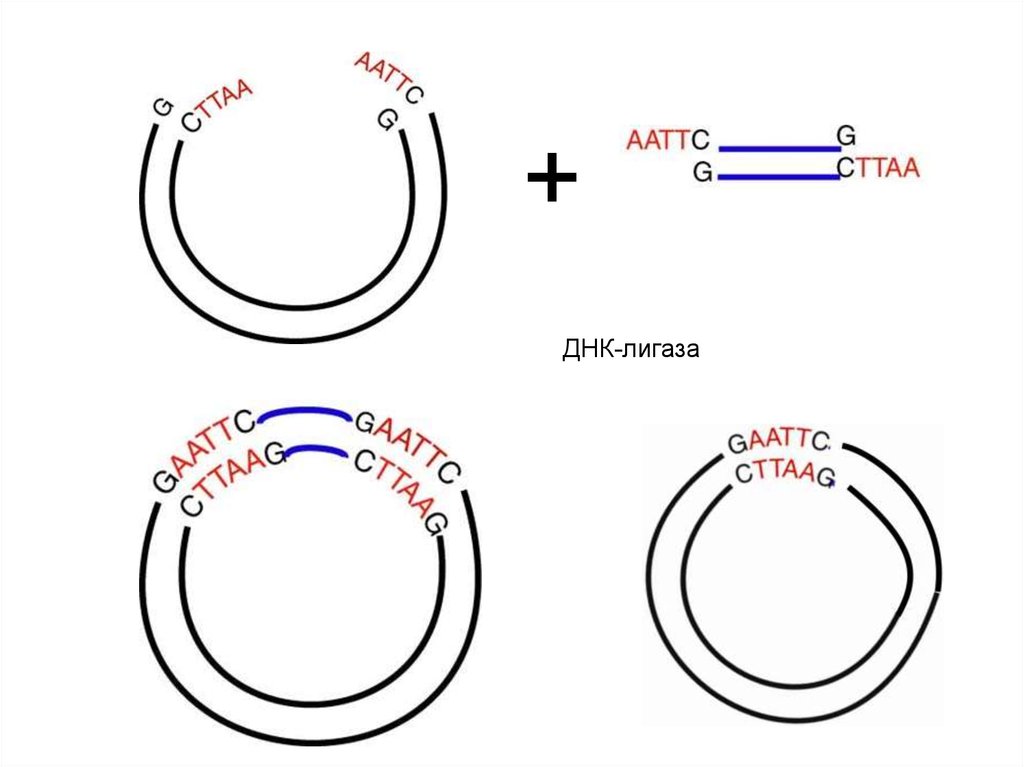
42. Соединение фрагментов с совместимыми липкими концами
G gatcCA gatcT
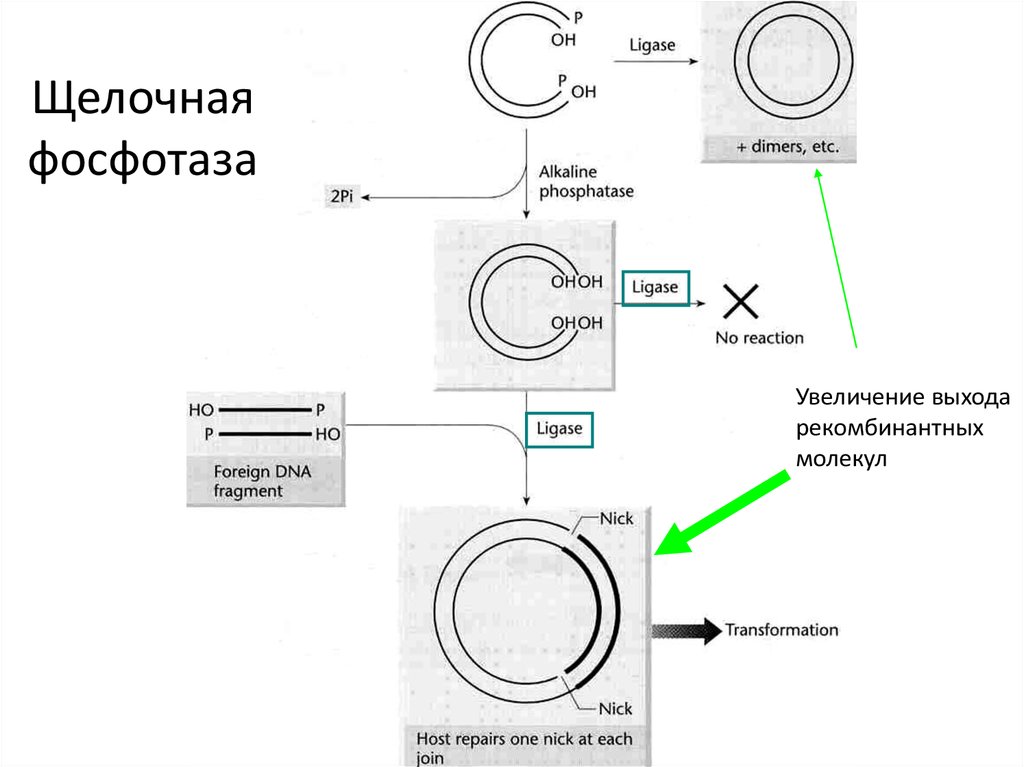
43. Щелочная фосфотаза
Увеличение выходарекомбинантных
молекул
44.
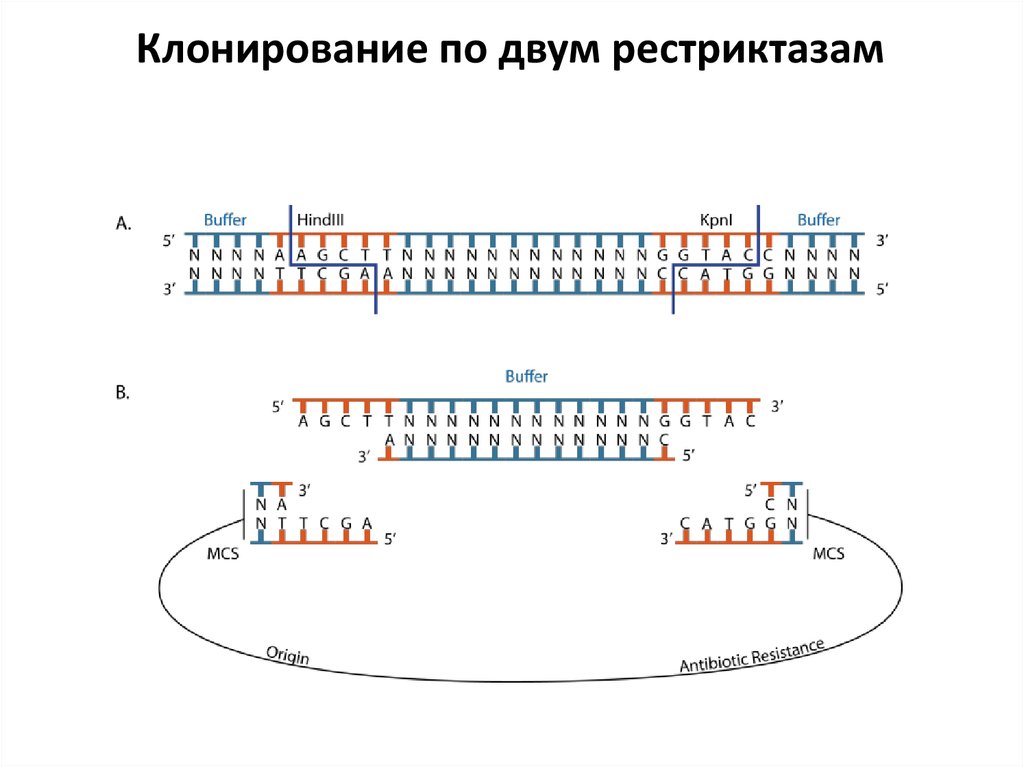
Клонирование по двум рестриктазам45. PCR
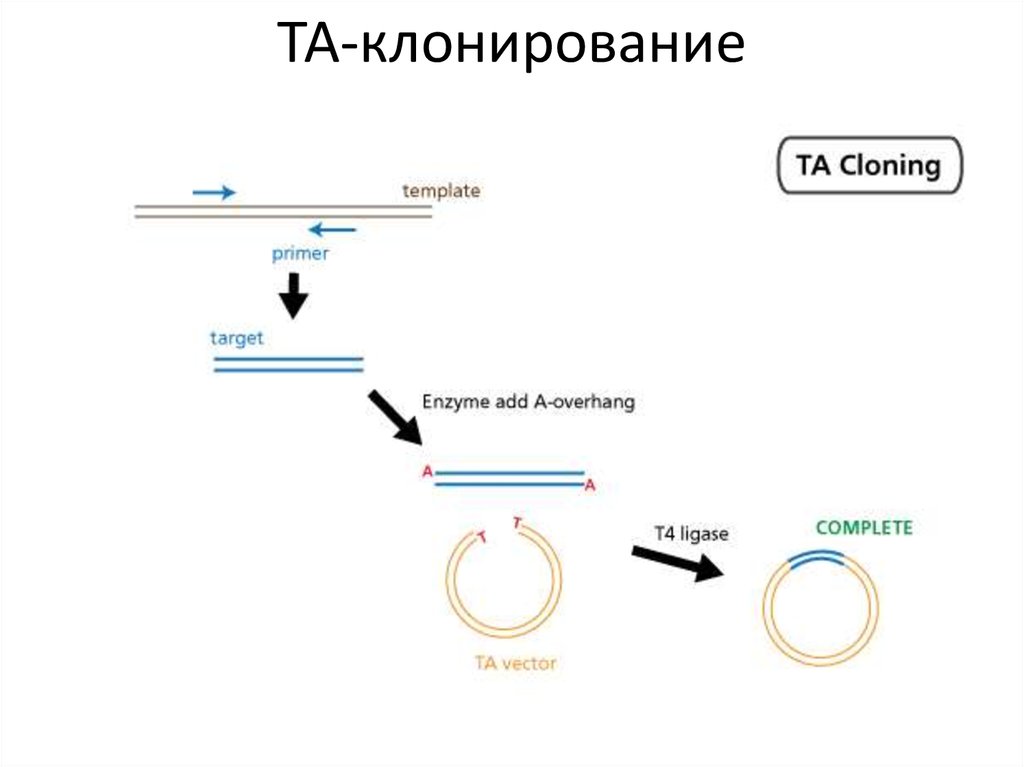
46.
ТА-клонирование47. Нематричный синтез ДНК ДНК-полимеразами
Нематричныйсинтез ДНК
ДНКполимеразами
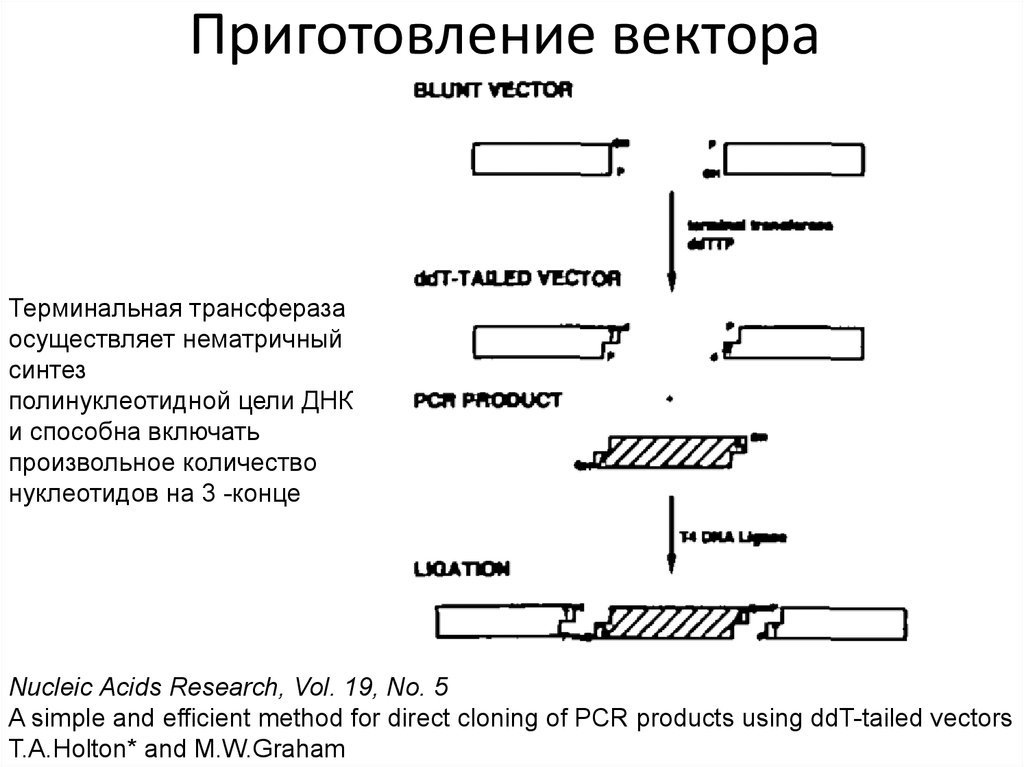
48. Приготовление вектора
Терминальная трансферазаосуществляет нематричный
синтез
полинуклеотидной цели ДНК
и способна включать
произвольное количество
нуклеотидов на 3 -конце
Nucleic Acids Research, Vol. 19, No. 5
A simple and efficient method for direct cloning of PCR products using ddT-tailed vectors
T.A.Holton* and M.W.Graham
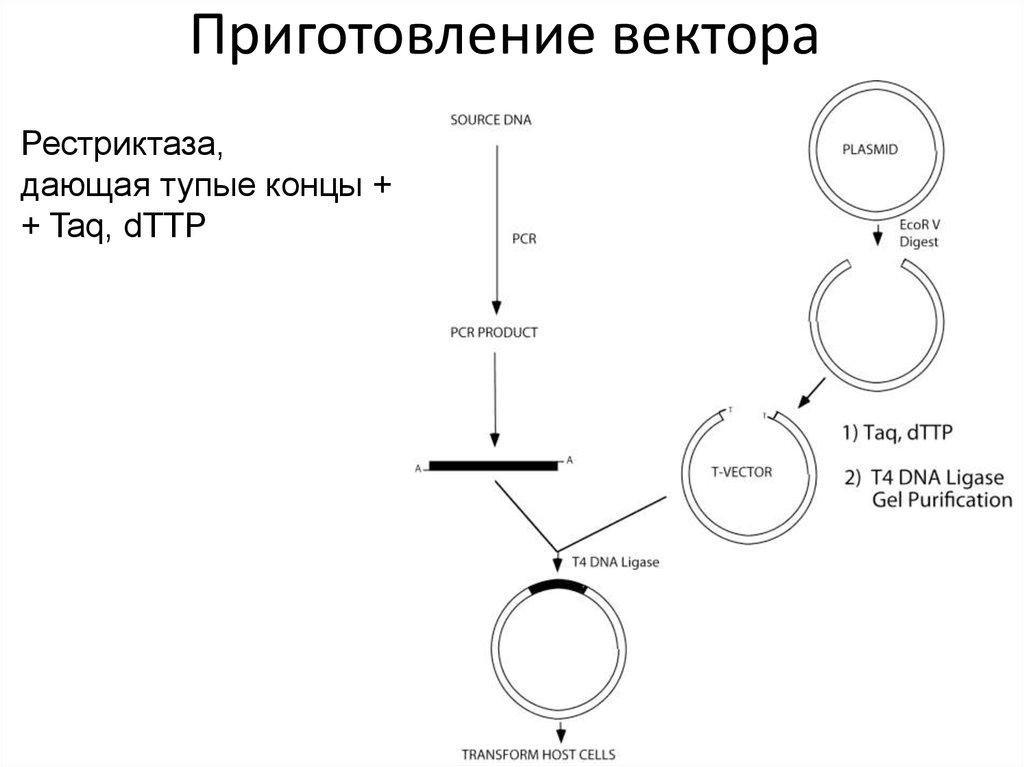
49. Приготовление вектора
Рестриктаза,дающая тупые концы +
+ Taq, dTTP
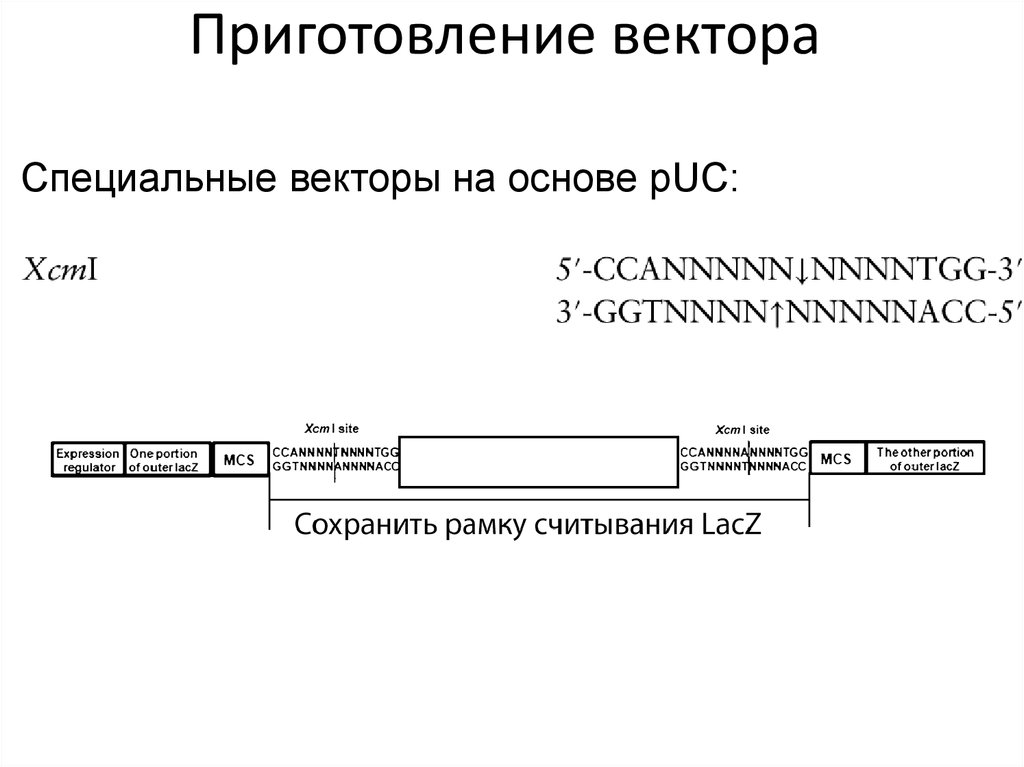
50. Приготовление вектора
Специальные векторы на основе pUC:51.
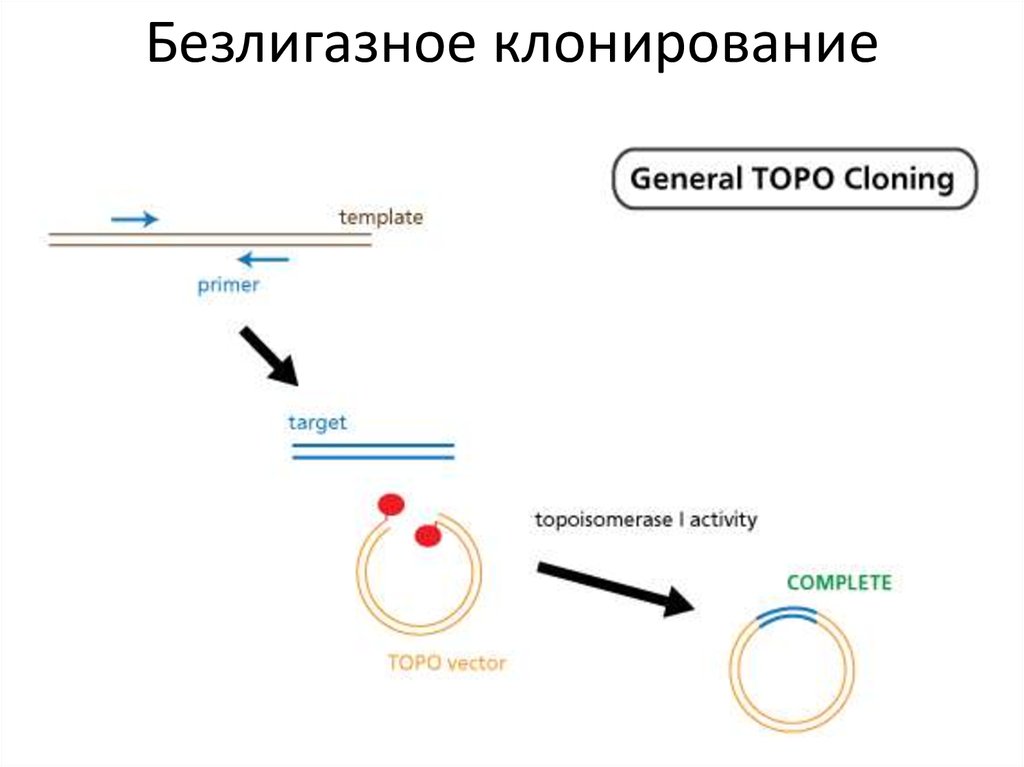
Безлигазное клонирование52.
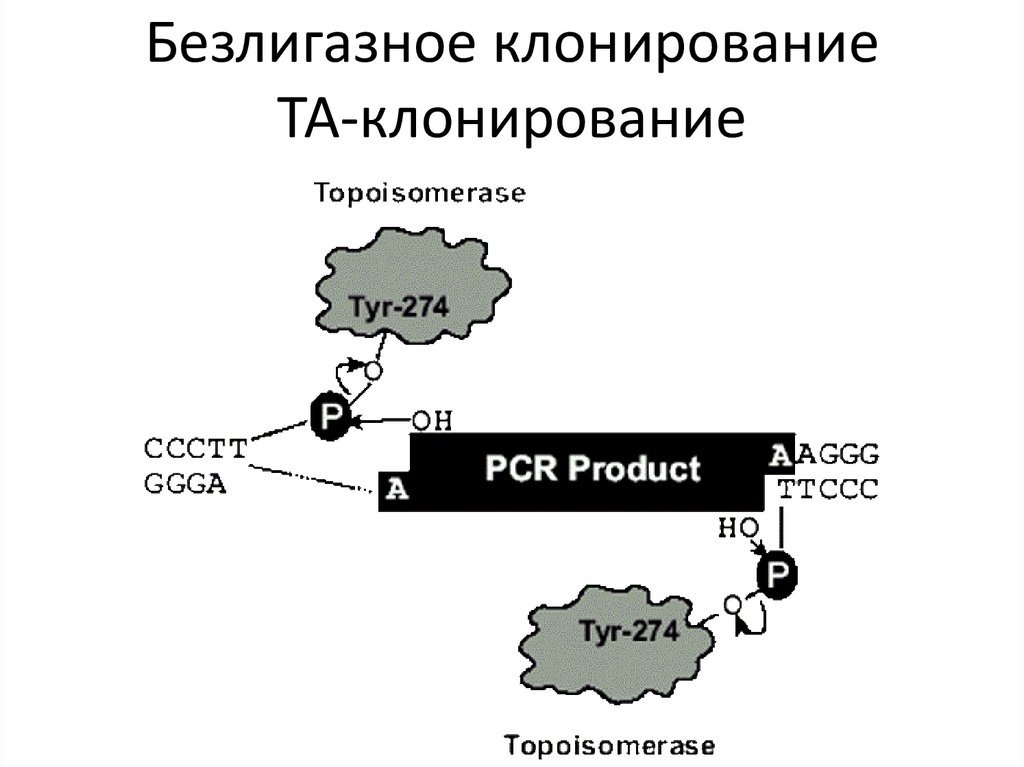
Безлигазное ТА-клонирование53.
Безлигазное клонированиеТА-клонирование
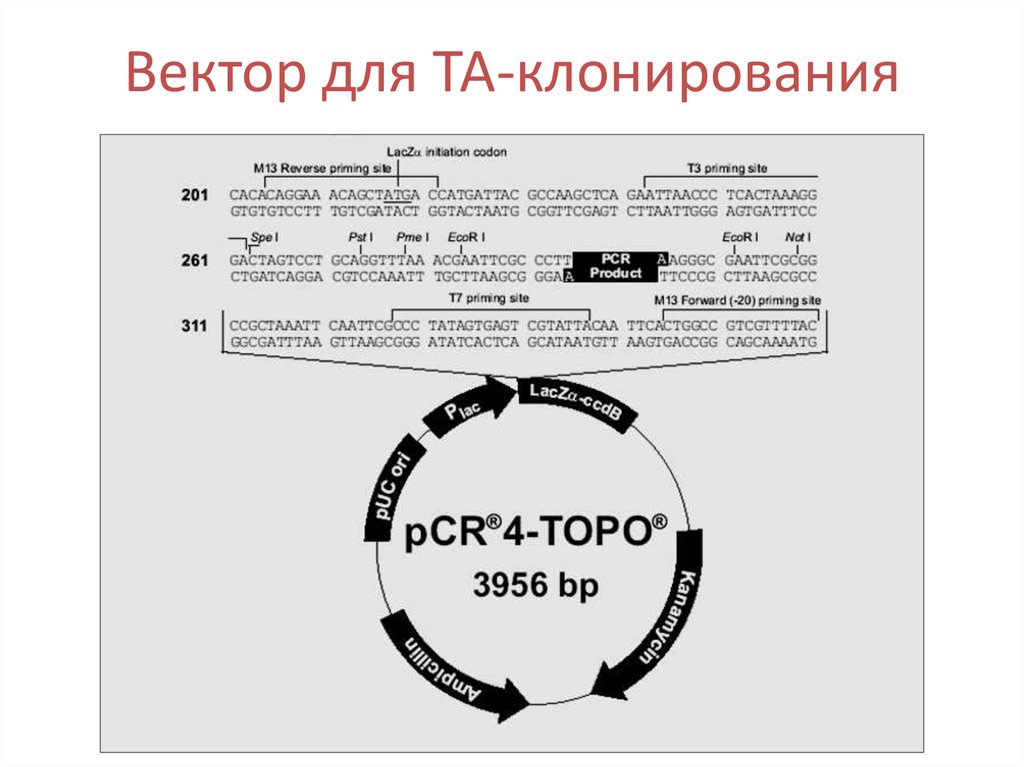
54.
Вектор для ТА-клонирования55. Бактериофаг М13
56.
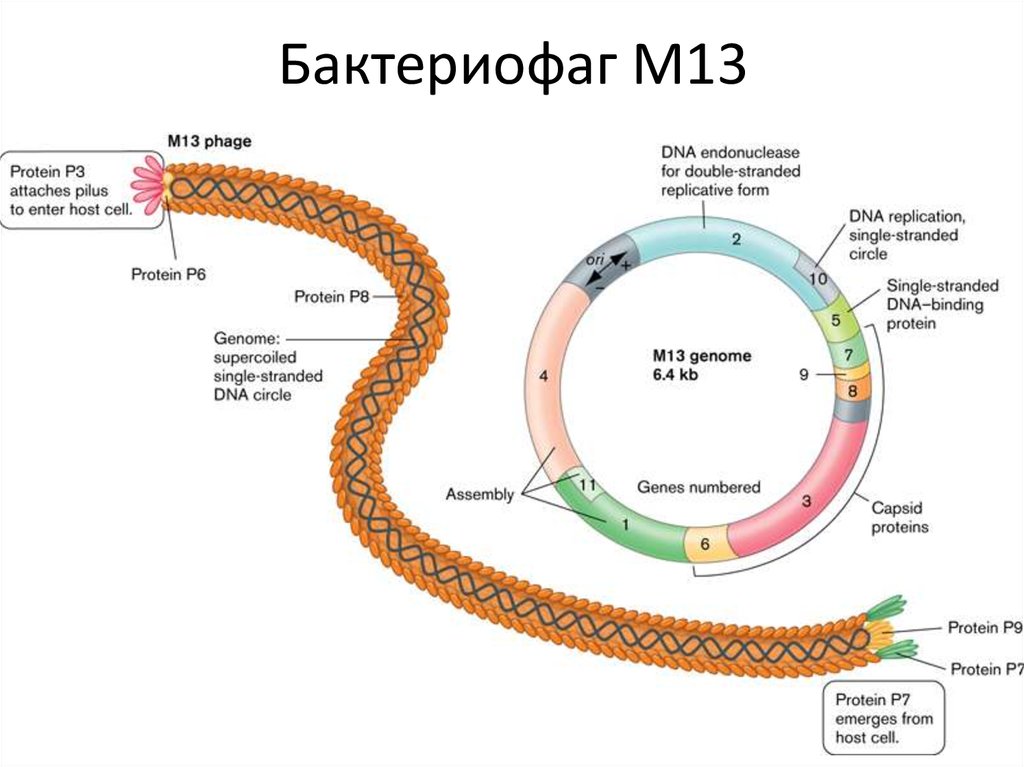
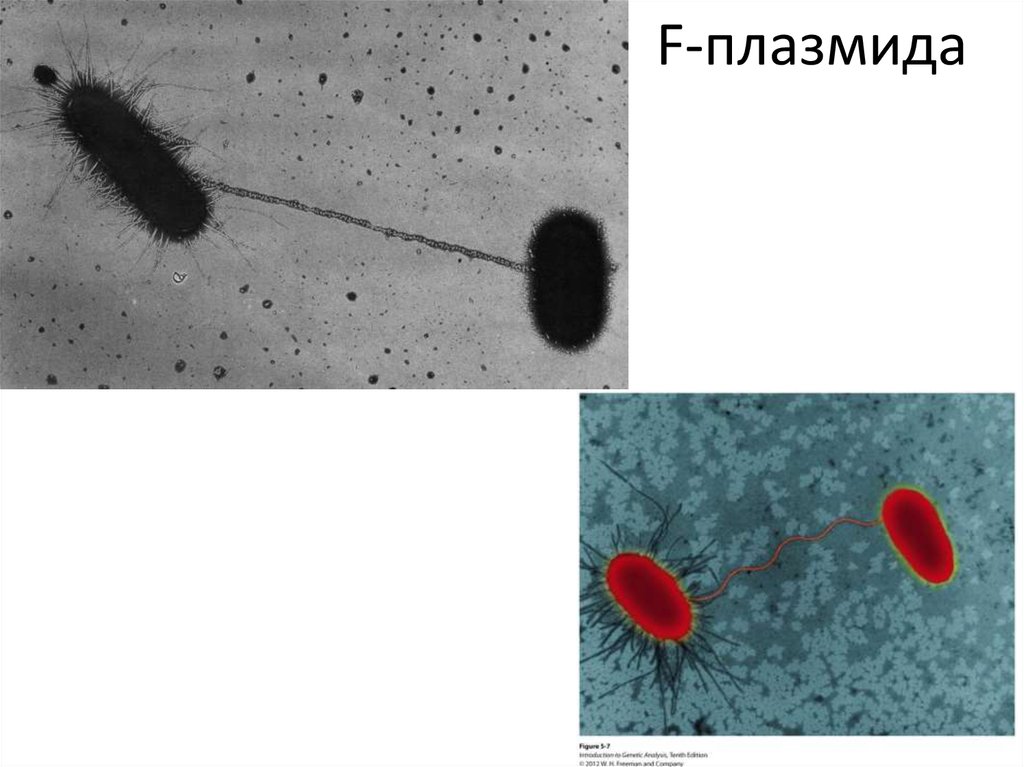
57. F-плазмида
58. Бактериофаг М13
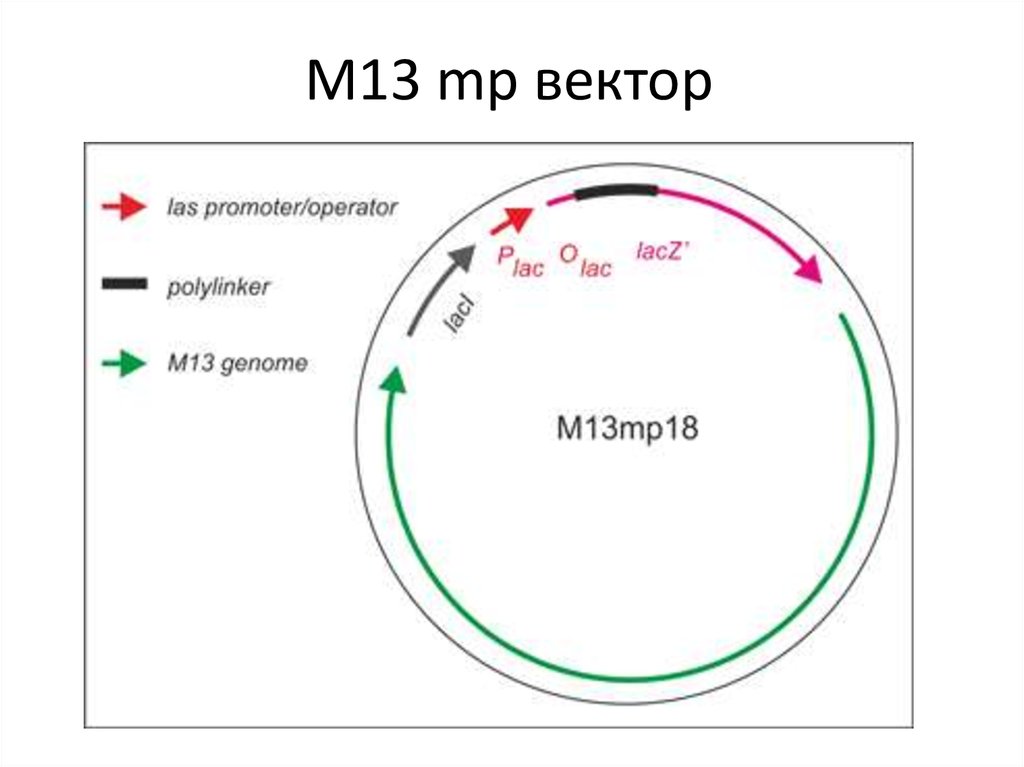
59. M13 mp вектор
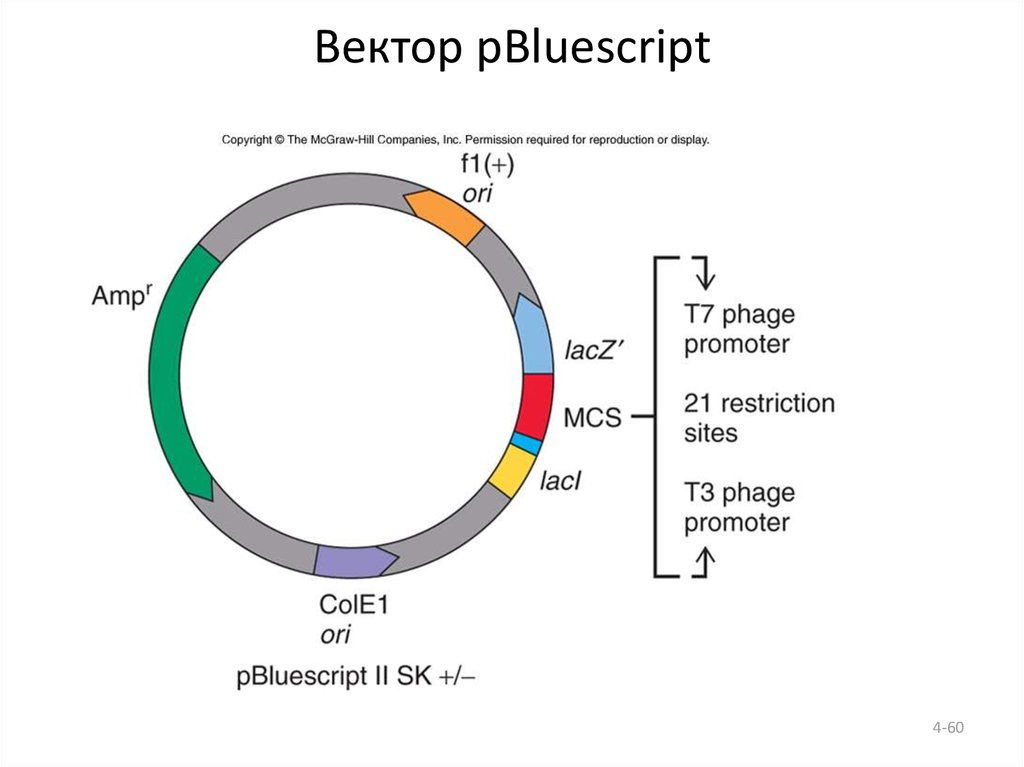
60. Вектор pBluescript
4-6061. Методы определения последовательности нуклеотидов
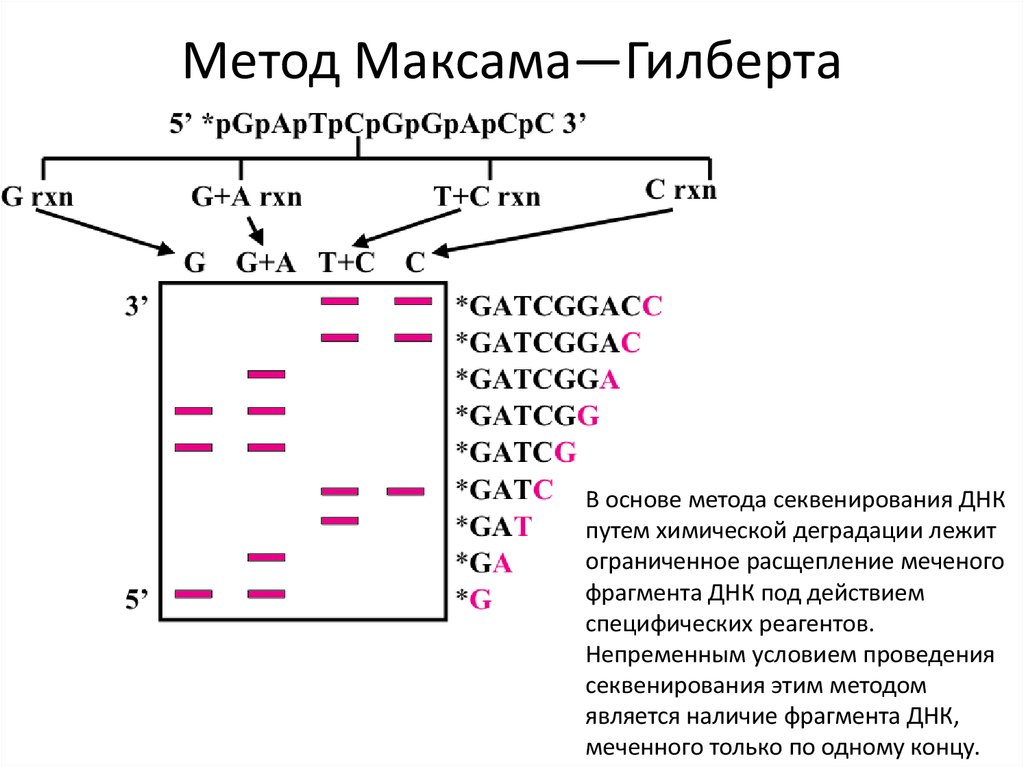
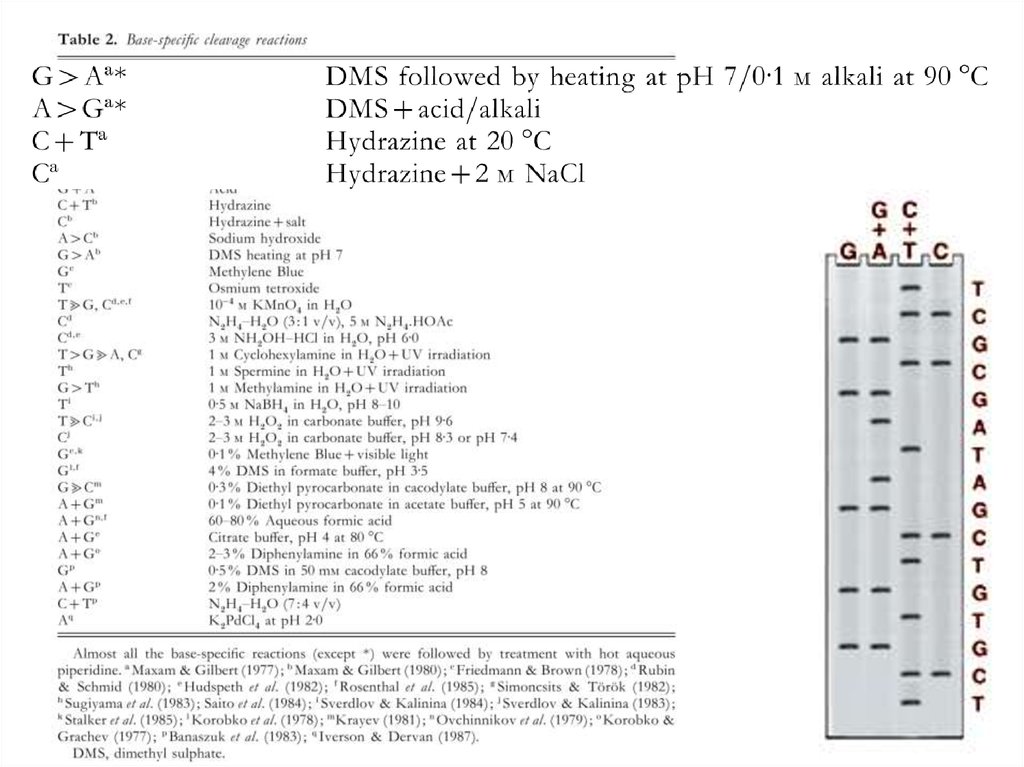
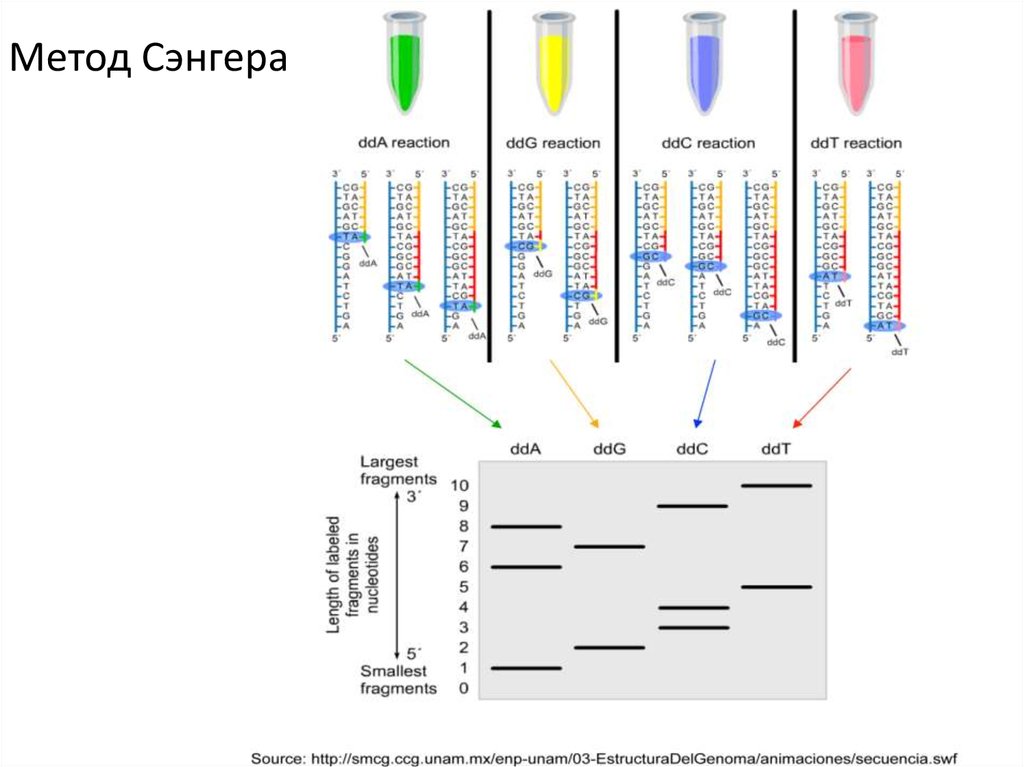
Метод Максама—ГилбертаМетод Сэнгера
1977 год
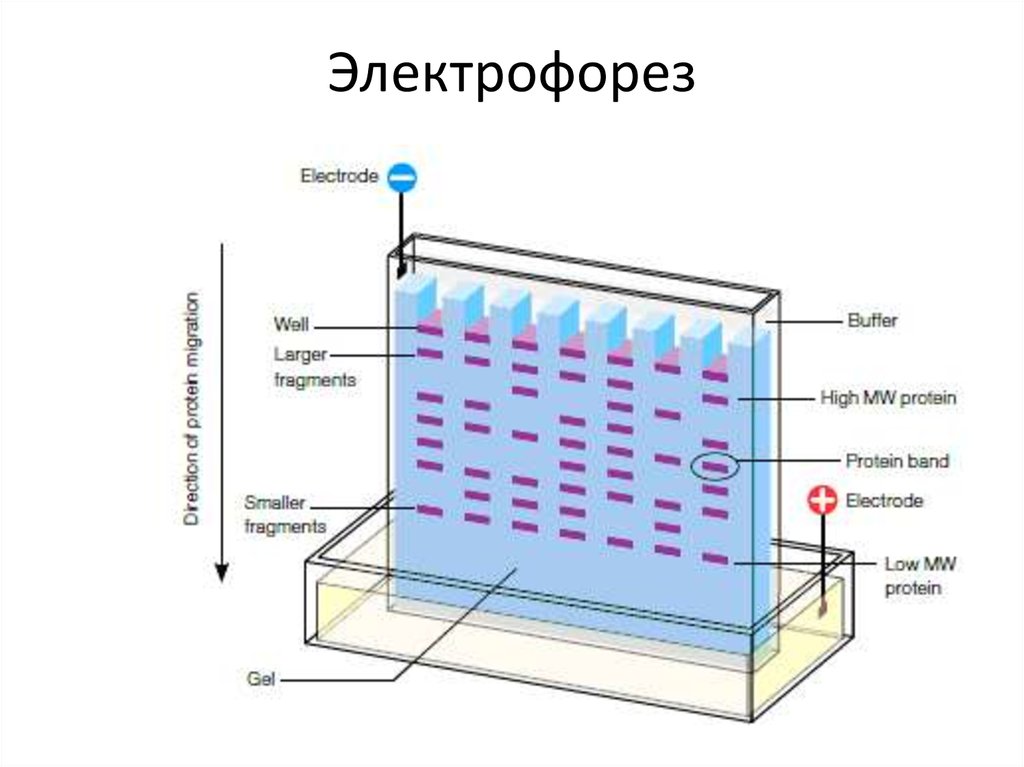
62.
Электрофорез63. Метод Максама—Гилберта
В основе метода секвенирования ДНКпутем химической деградации лежит
ограниченное расщепление меченого
фрагмента ДНК под действием
специфических реагентов.
Непременным условием проведения
секвенирования этим методом
является наличие фрагмента ДНК,
меченного только по одному концу.
64.
65.
Метод Сэнгера66.
Метод Сэнгера67.
Метод СэнгераAGCT
68.
Для разделения фрагментовДНК с разрешением
в 1 нуклеотид используют:
8–10% полиакриламидный гель
7М мочевина
Температура в геле
при проведении
электрофореза 60–80 С
69.
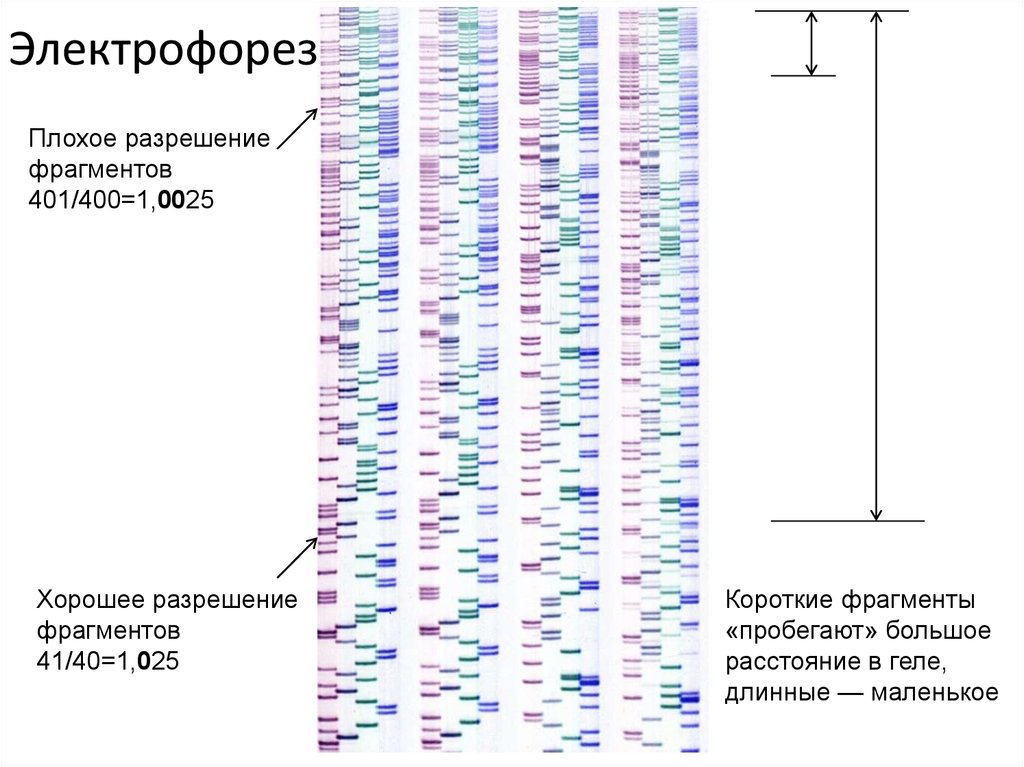
ЭлектрофорезПлохое разрешение
фрагментов
401/400=1,0025
Хорошее разрешение
фрагментов
41/40=1,025
Короткие фрагменты
«пробегают» большое
расстояние в геле,
длинные — маленькое
70.
Размер одного«прочтения»
400–700 букв
71.
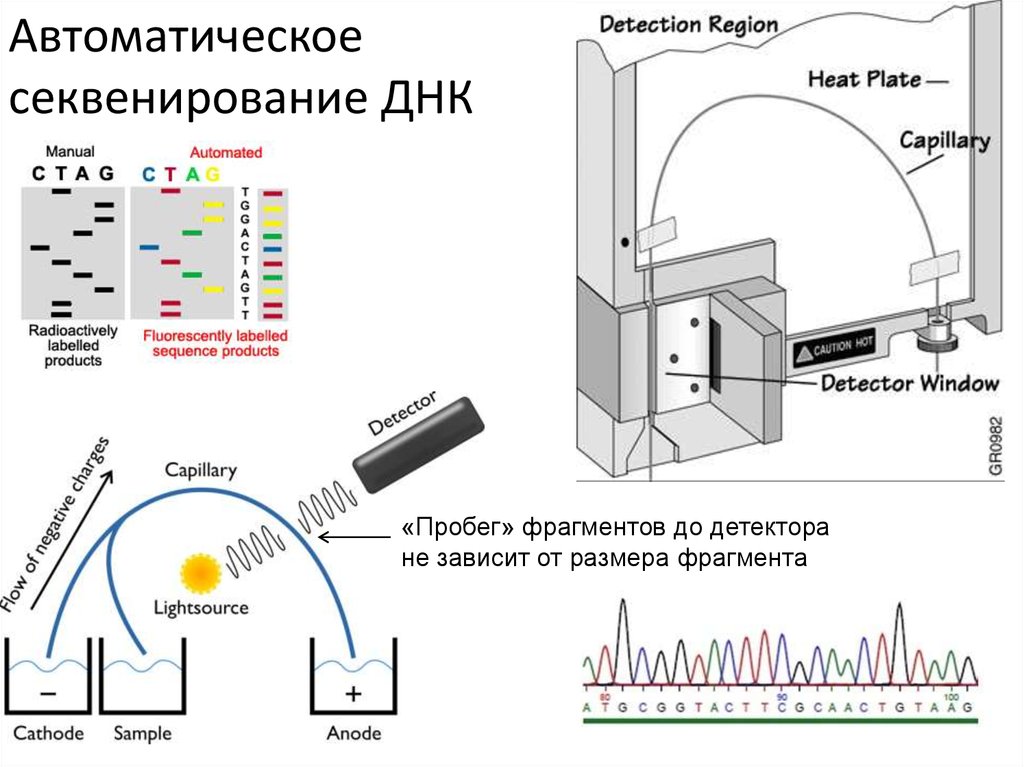
Автоматическоесеквенирование ДНК
«Пробег» фрагментов до детектора
не зависит от размера фрагмента
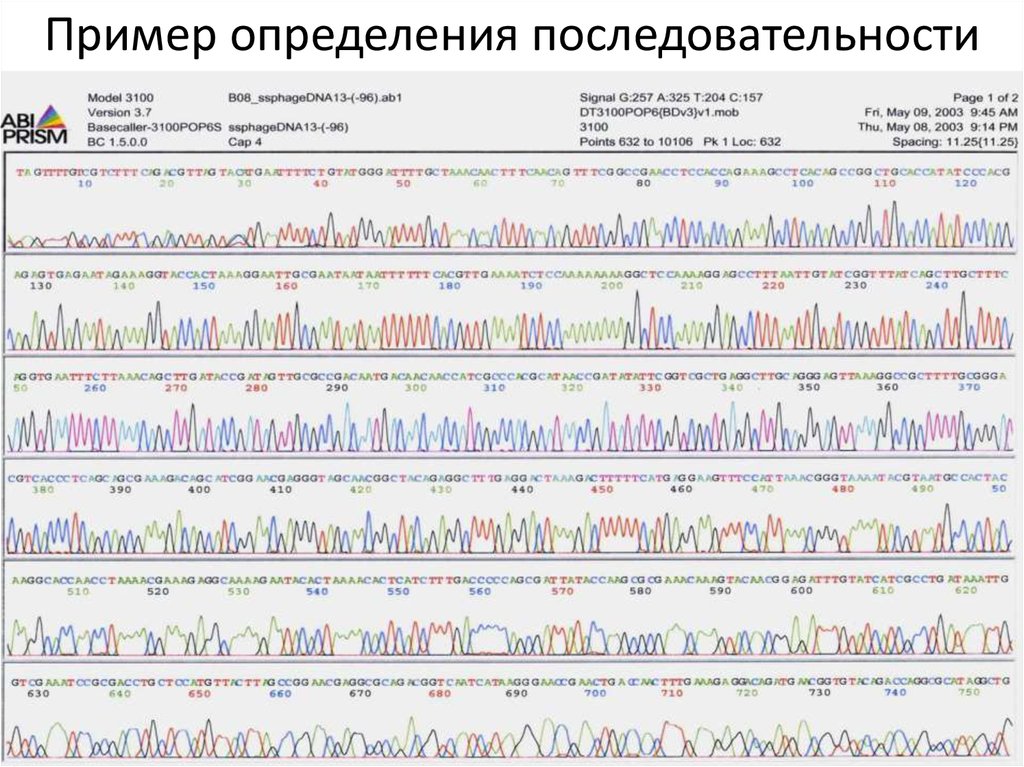
72. Пример определения последовательности
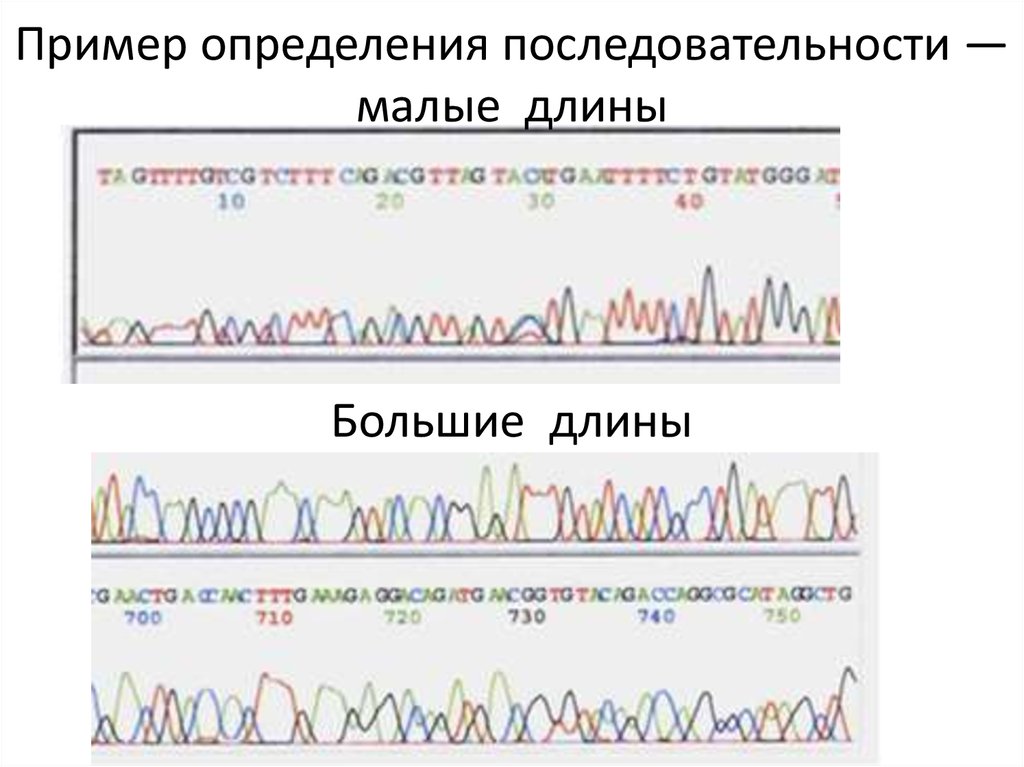
73.
Пример определения последовательности —малые длины
Большие длины
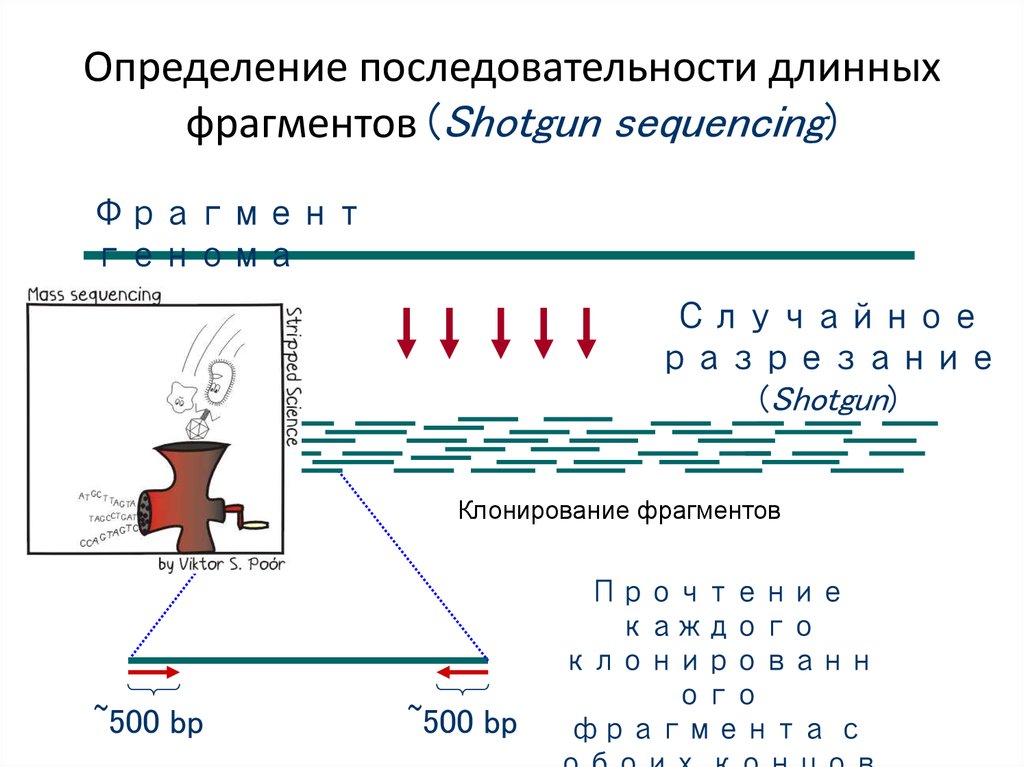
74. Определение последовательности длинных фрагментов (Shotgun sequencing)
Фрагментгенома
Случайное
разрезание
(Shotgun)
Клонирование фрагментов
~500 bp
~500 bp
Прочтение
каждого
клонированн
ого
фрагмента с
75.
Реконструирование последовательности(Fragment Assembly)
reads
Необходимо 7–10 кратное прочтение
каждого участка
76. Редактирование генома
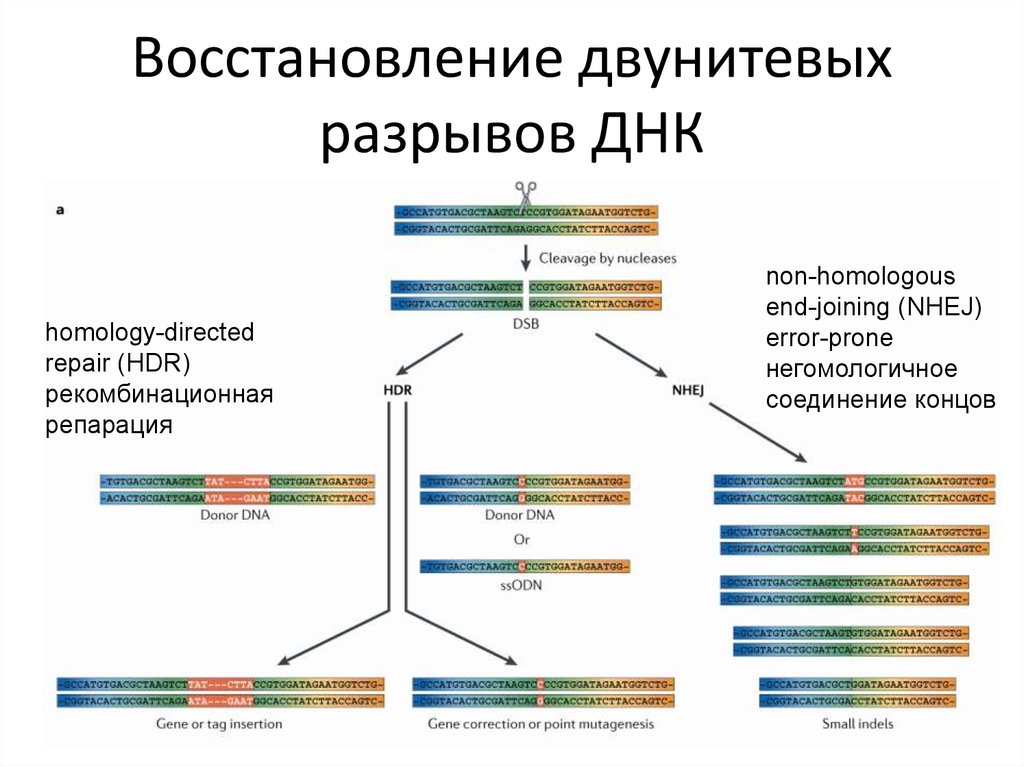
77. Восстановление двунитевых разрывов ДНК
homology-directedrepair (HDR)
рекомбинационная
репарация
non-homologous
end-joining (NHEJ)
error-prone
негомологичное
соединение концов
78. 4L = 109 (размер генома человека) log4L = log109 2L log2 = 9 L = 15
79.
80.
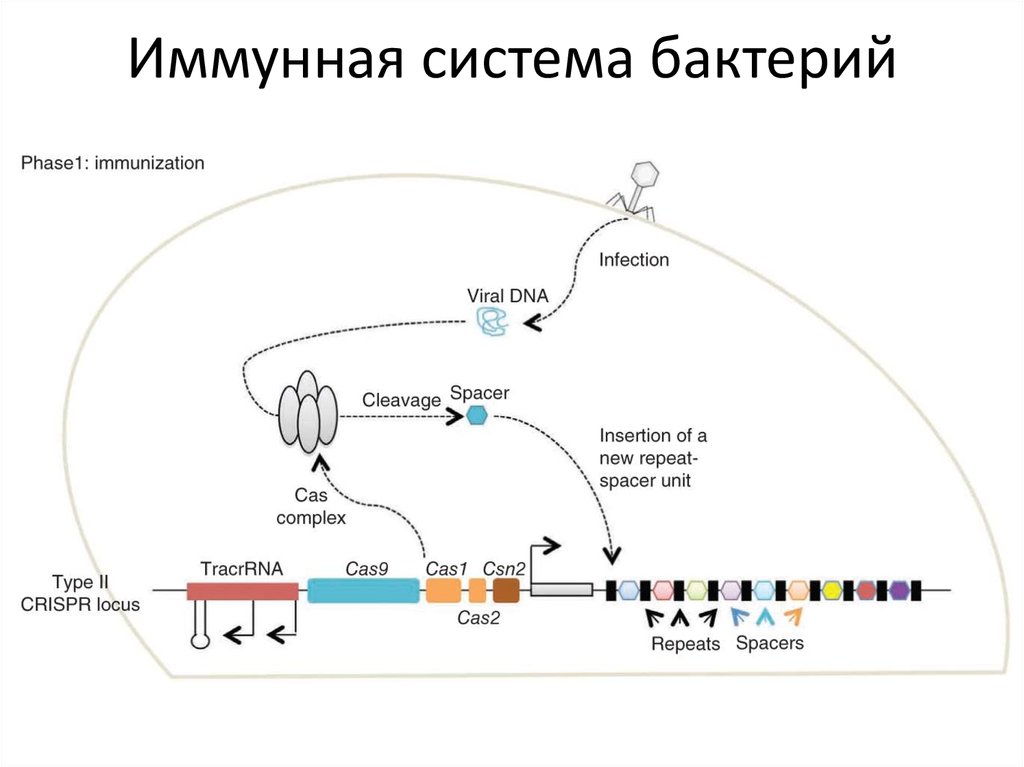
Хромосома бактерииCRISPR (Clustered Regularly
Interspaced Short Palindromic
Repeats) — это
кластеризованные
регуляторные разделенные
промежутками короткие
палиндромные повторы или
участки ДНК, содержащие
множественные повторы,
разделенные уникальными
участками — спейсерами
Cavanagh & Garrity, “CRISPR Mechanism”, CRISPR/Cas9, Tufts University, 2014.
https://sites.tufts.edu/crispr/ (Date of Access)
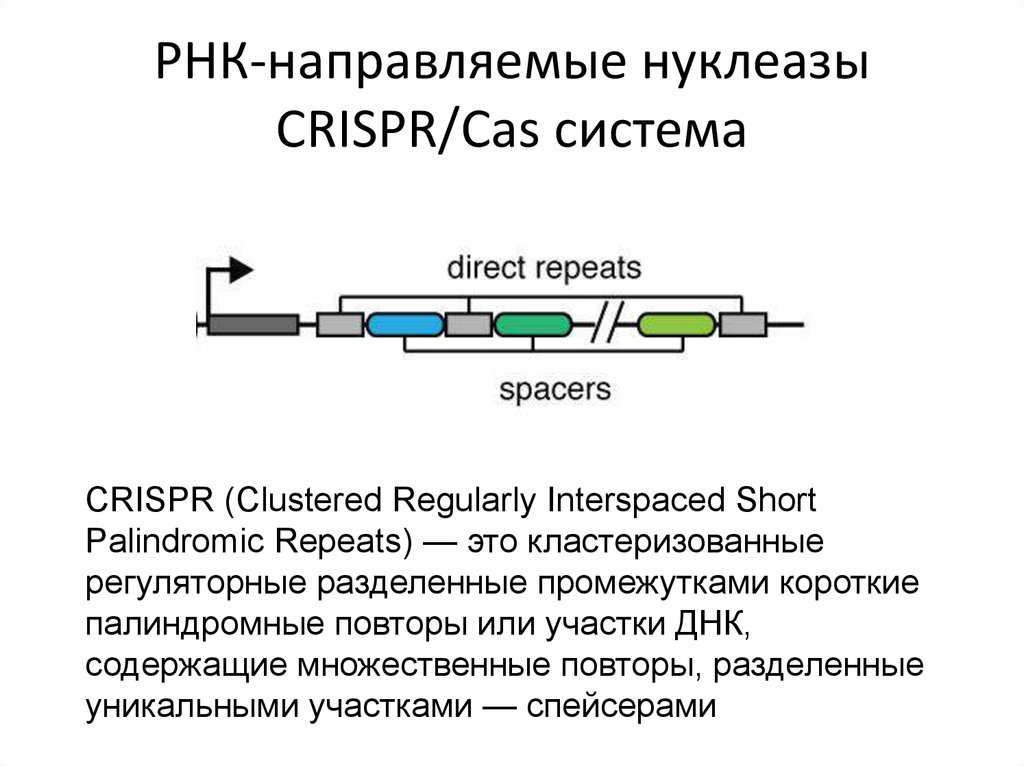
81. РНК-направляемые нуклеазы CRISPR/Cas система
CRISPR (Clustered Regularly Interspaced ShortPalindromic Repeats) — это кластеризованные
регуляторные разделенные промежутками короткие
палиндромные повторы или участки ДНК,
содержащие множественные повторы, разделенные
уникальными участками — спейсерами
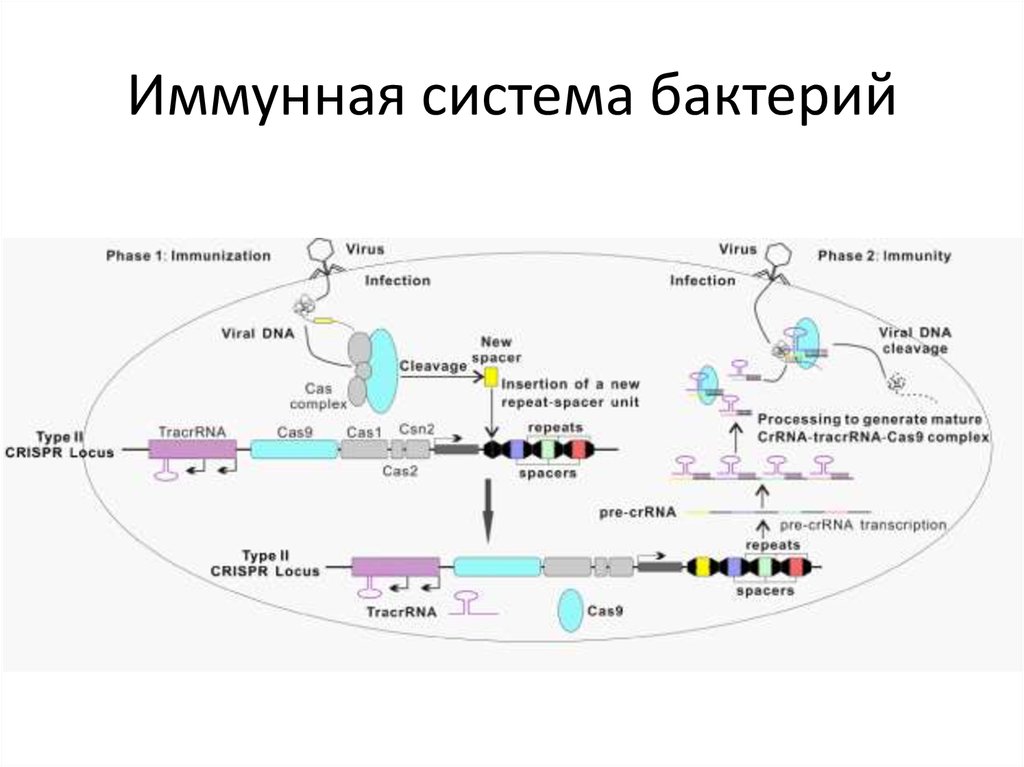
82. Иммунная система бактерий
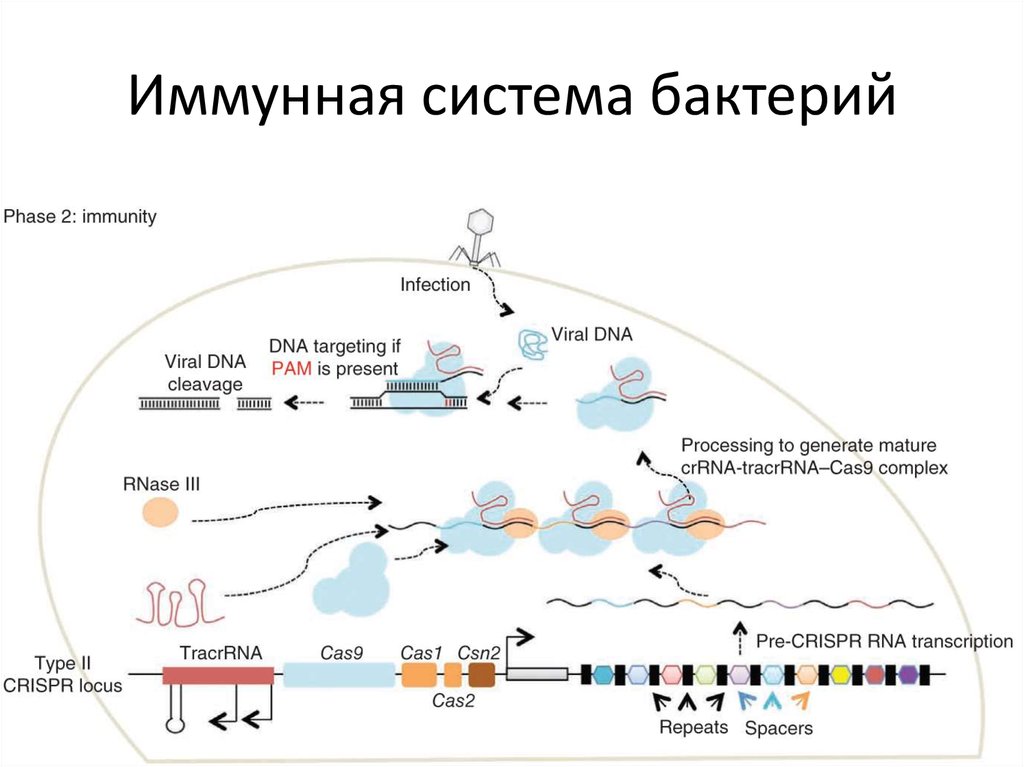
83. Иммунная система бактерий
84. Иммунная система бактерий
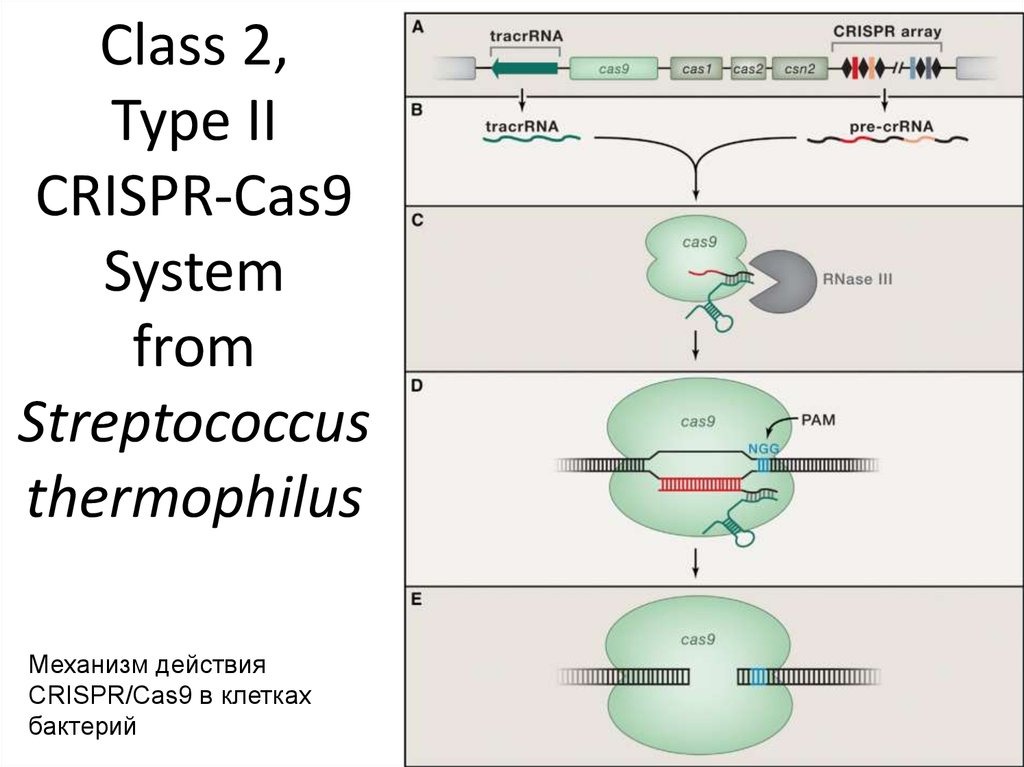
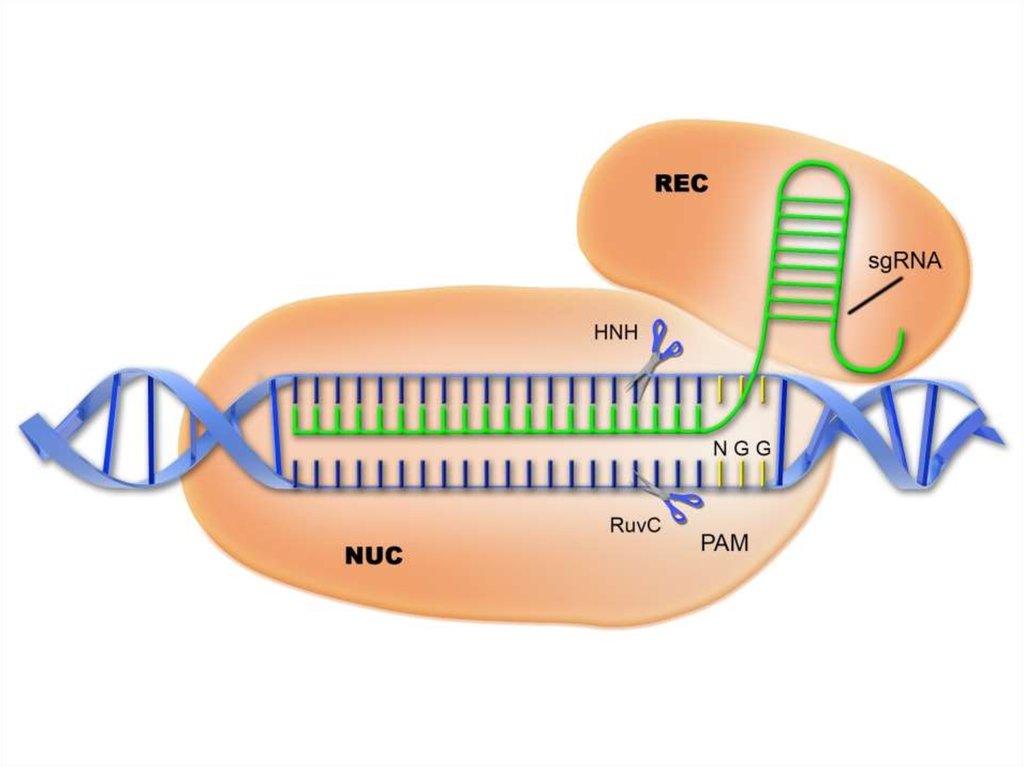
85. Class 2, Type II CRISPR-Cas9 System from Streptococcus thermophilus
Механизм действияCRISPR/Cas9 в клетках
бактерий
86.
87.
88.
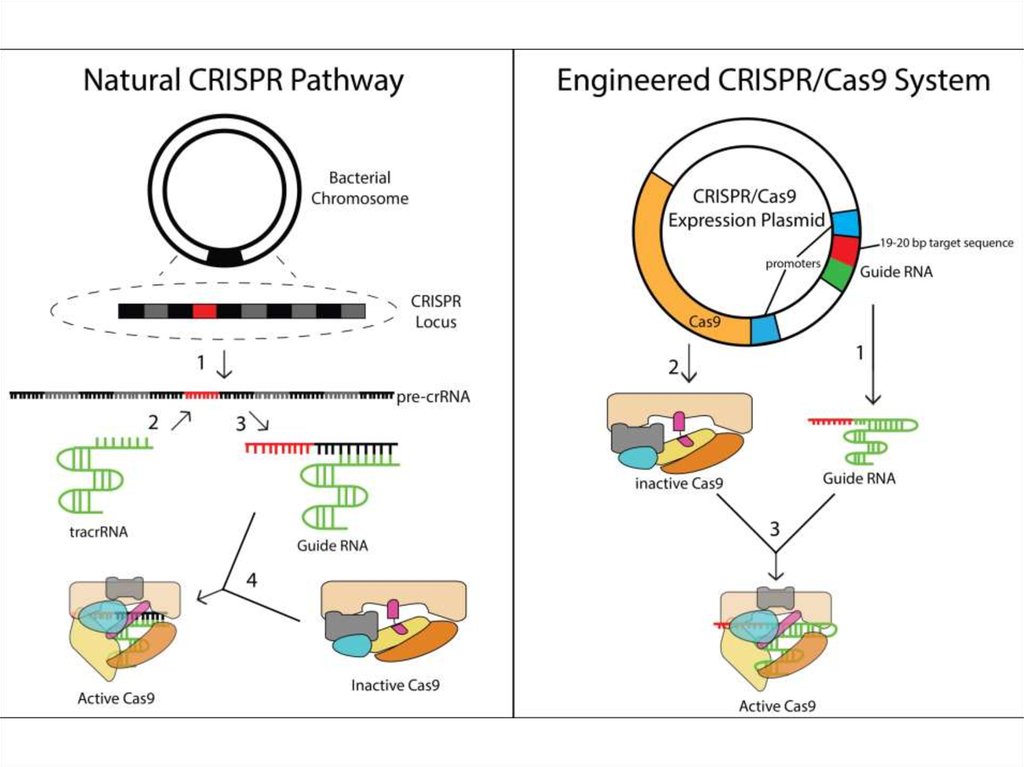
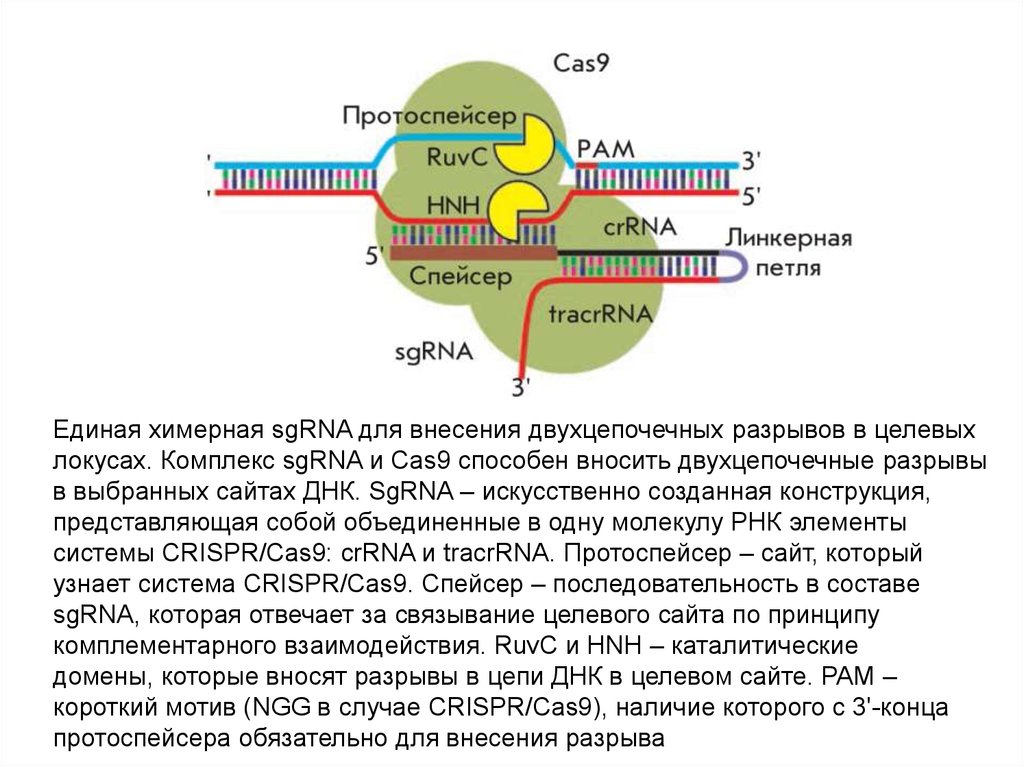
Единая химерная sgRNA для внесения двухцепочечных разрывов в целевыхлокусах. Комплекс sgRNA и Cas9 способен вносить двухцепочечные разрывы
в выбранных сайтах ДНК. SgRNA – искусственно созданная конструкция,
представляющая собой объединенные в одну молекулу РНК элементы
системы CRISPR/Cas9: crRNA и tracrRNA. Протоспейсер – сайт, который
узнает система CRISPR/Cas9. Спейсер – последовательность в составе
sgRNA, которая отвечает за связывание целевого сайта по принципу
комплементарного взаимодействия. RuvC и HNH – каталитические
домены, которые вносят разрывы в цепи ДНК в целевом сайте. PAM –
короткий мотив (NGG в случае CRISPR/Cas9), наличие которого с 3'-конца
протоспейсера обязательно для внесения разрыва
89. Создание RGEN (RNA-guided engineered nuclease)
90. Создание RGEN (RNA-guided engineered nuclease)
91.
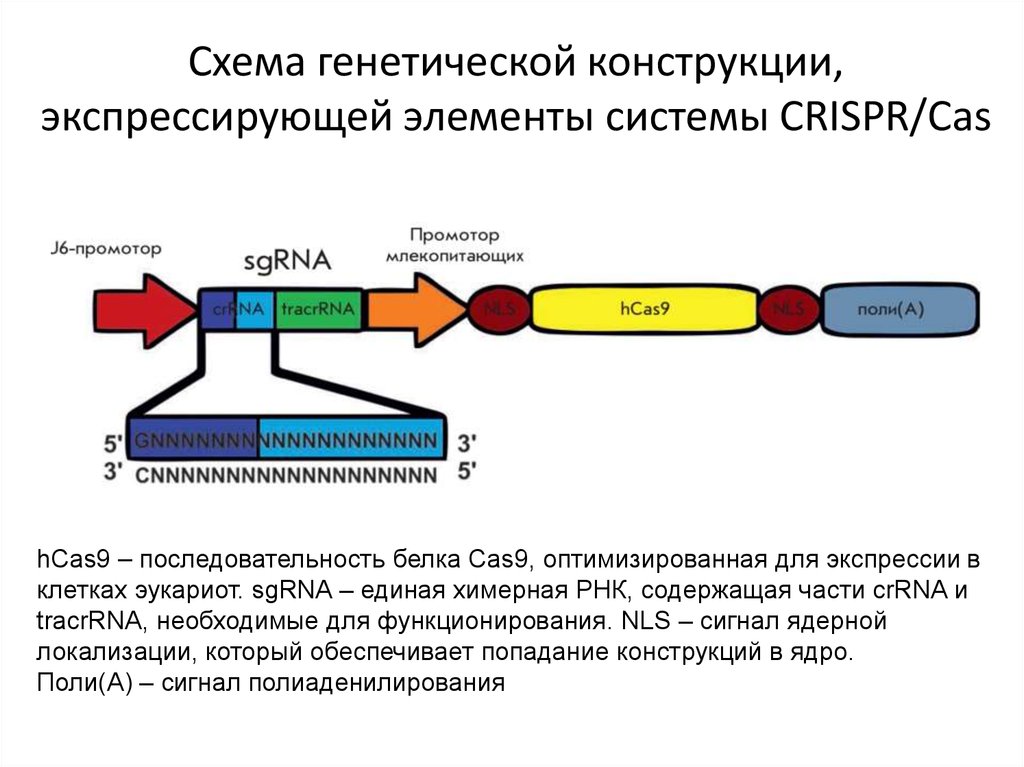
Схема генетической конструкции,экспрессирующей элементы системы CRISPR/Cas
hCas9 – последовательность белка Cas9, оптимизированная для экспрессии в
клетках эукариот. sgRNA – единая химерная РНК, содержащая части crRNA и
tracrRNA, необходимые для функционирования. NLS – сигнал ядерной
локализации, который обеспечивает попадание конструкций в ядро.
Поли(А) – сигнал полиаденилирования
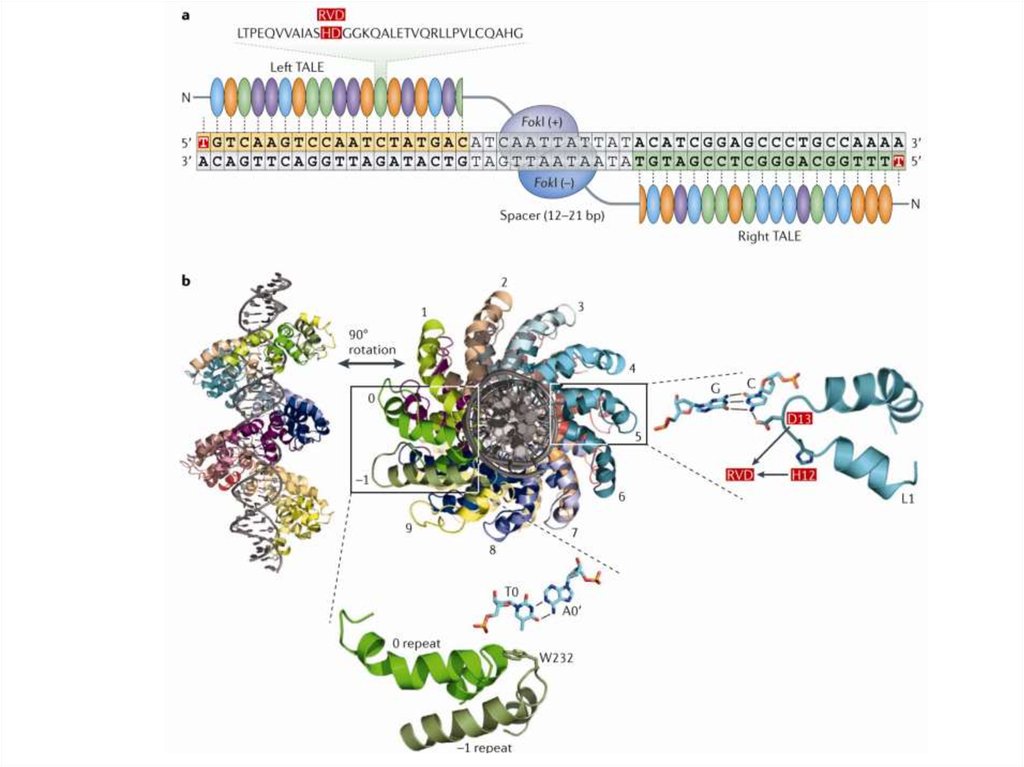
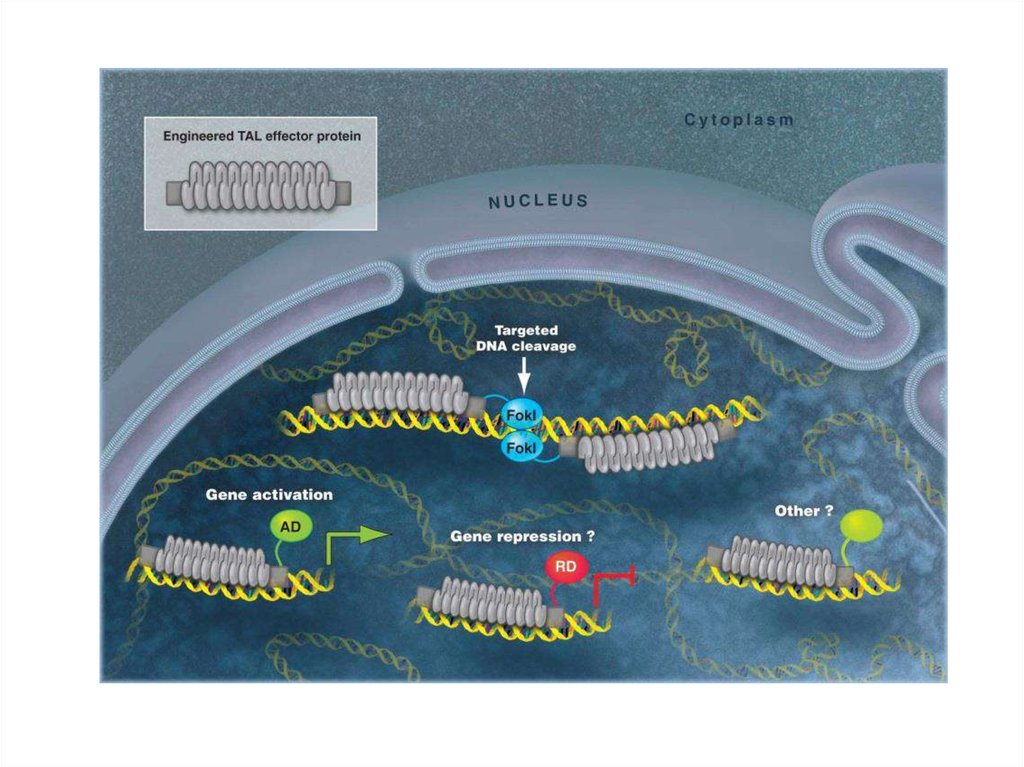
92. TALEN (Transcription Activator-Like Effector Nucleases; эффекторные нуклеазы, подобные активаторам транскрипции)
93. Transcription Activator-Like Effectors, TALE
TALE белки были обнаружены у бактерий родаXanthomonas поражающих рис, перец, хлопок и
другие растения.
Бактерии секретируют в цитоплазму растительных
клеток эффекторные белки (TALE), которые влияют
на процессы в растительной клетке и увеличивают
ее восприимчивость к патогену.
Эффекторные белки способны связываться с ДНК
и активировать экспрессию своих генов-мишеней,
имитируя факторы транскрипции эукариот.
94. TALE состоят из центрального домена, ответственного за связывание ДНК, сигнала ядерной локализации (NLS) и домена,
активирующеготранскрипцию целевого гена (AD)
repeat-variable diresidue (RVD)
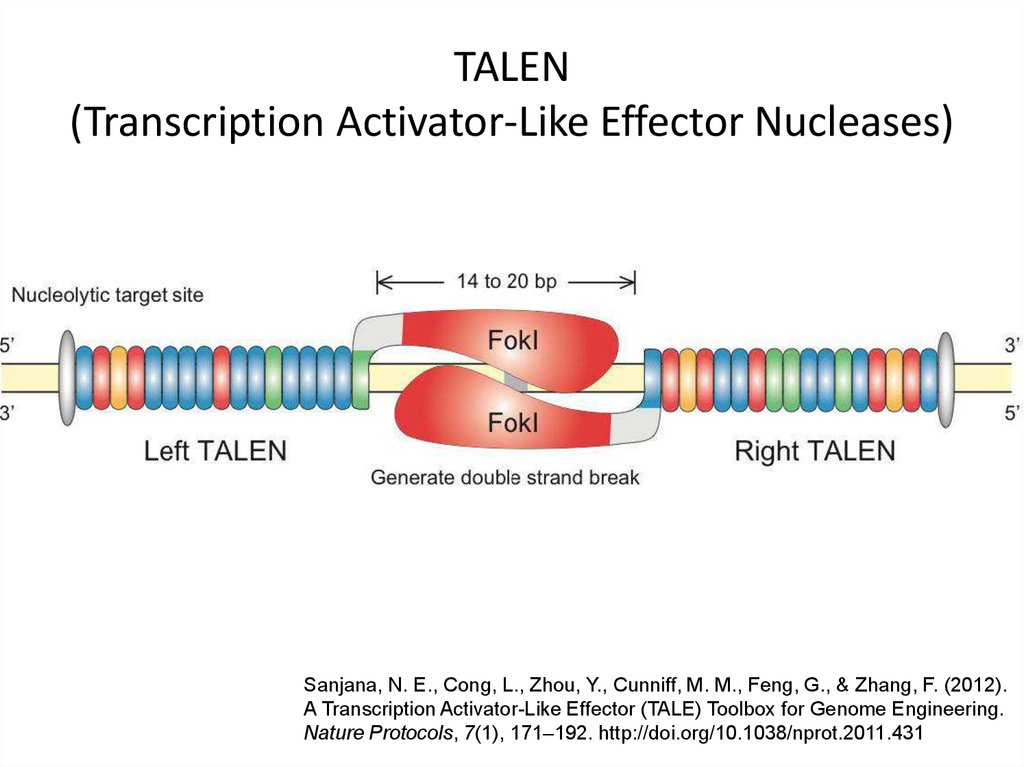
95. TALEN (Transcription Activator-Like Effector Nucleases)
Sanjana, N. E., Cong, L., Zhou, Y., Cunniff, M. M., Feng, G., & Zhang, F. (2012).A Transcription Activator-Like Effector (TALE) Toolbox for Genome Engineering.
Nature Protocols, 7(1), 171–192. http://doi.org/10.1038/nprot.2011.431
96.
97.
98. Транскрипционные факторы
Транскрипционные факторы содержат следующие домены:ДНК-связывающий домен (DBD) — взаимодействует со специфичными
последовательностями ДНК, характерными для промоторов и
энхансеров. Специфичность распознавания определенных
последовательностей определяет набор генов, подверженных
регуляции данным ТФ;
трансактивирующий домен (TAD) — содержит участки связывания
других белков, например, транскрипционных корегуляторов ;
сигналраспознающий домен (SSD) (например, лиганд-связывающий
домен), который чувствителен к внешнем сигналам и отвечающим за
передачу сигнала к другим компонентам транскрипционного
комплекса, что вызывает повышение или понижение уровня
экспрессии
99.
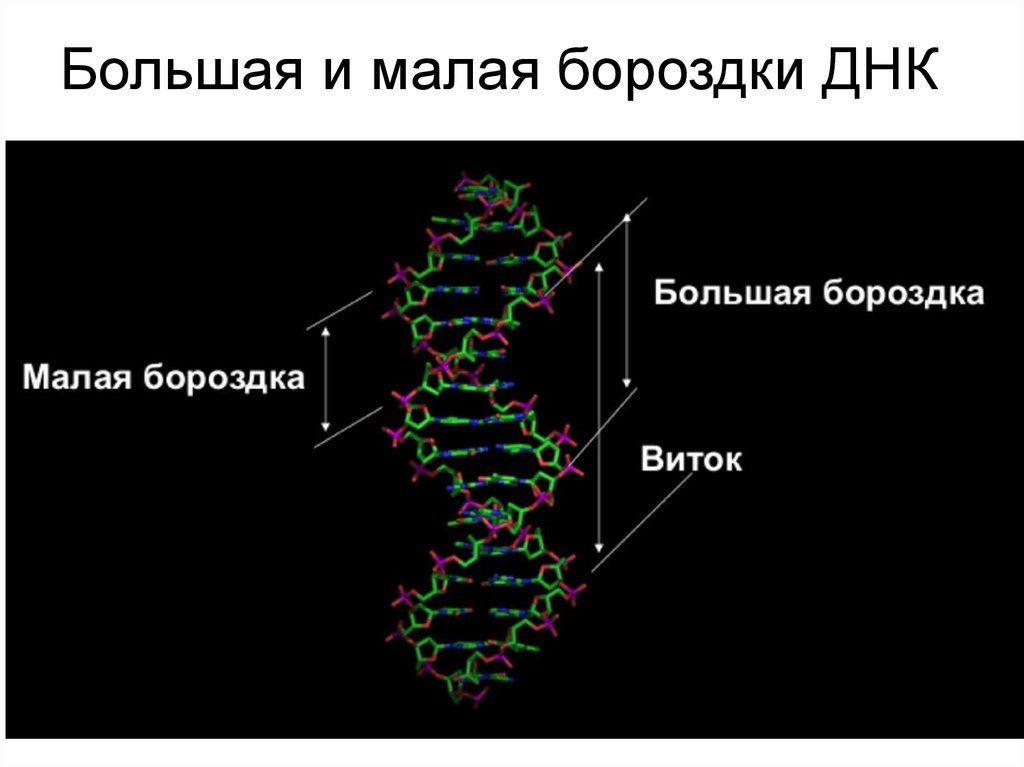
Большая и малая бороздки ДНК100.
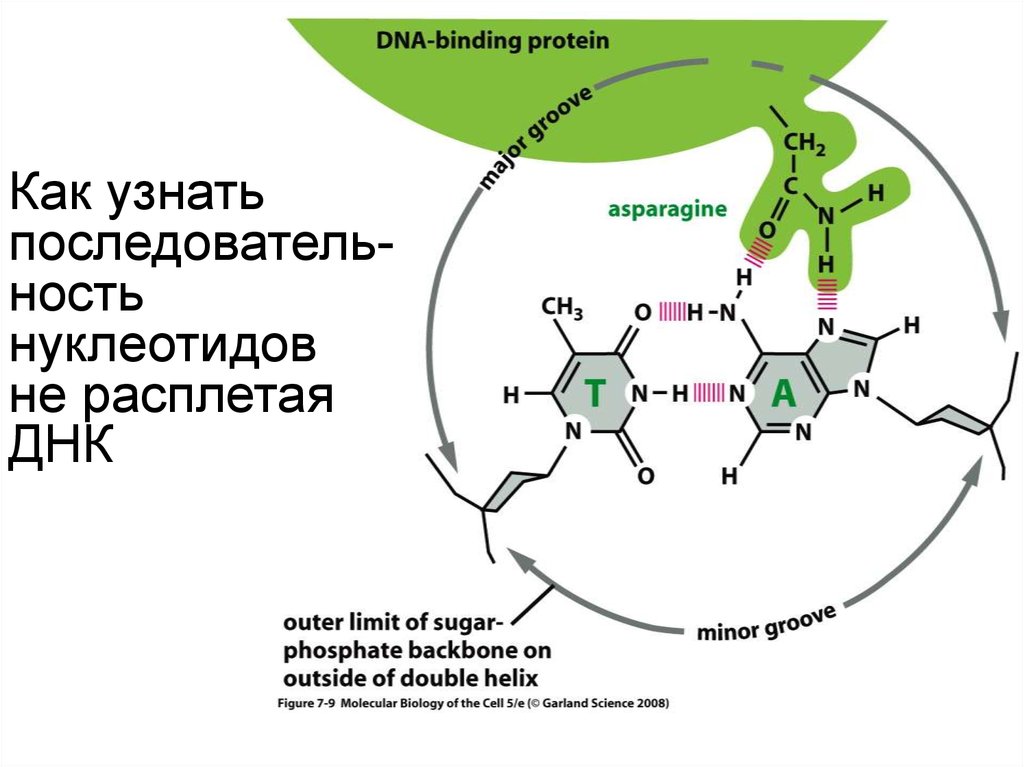
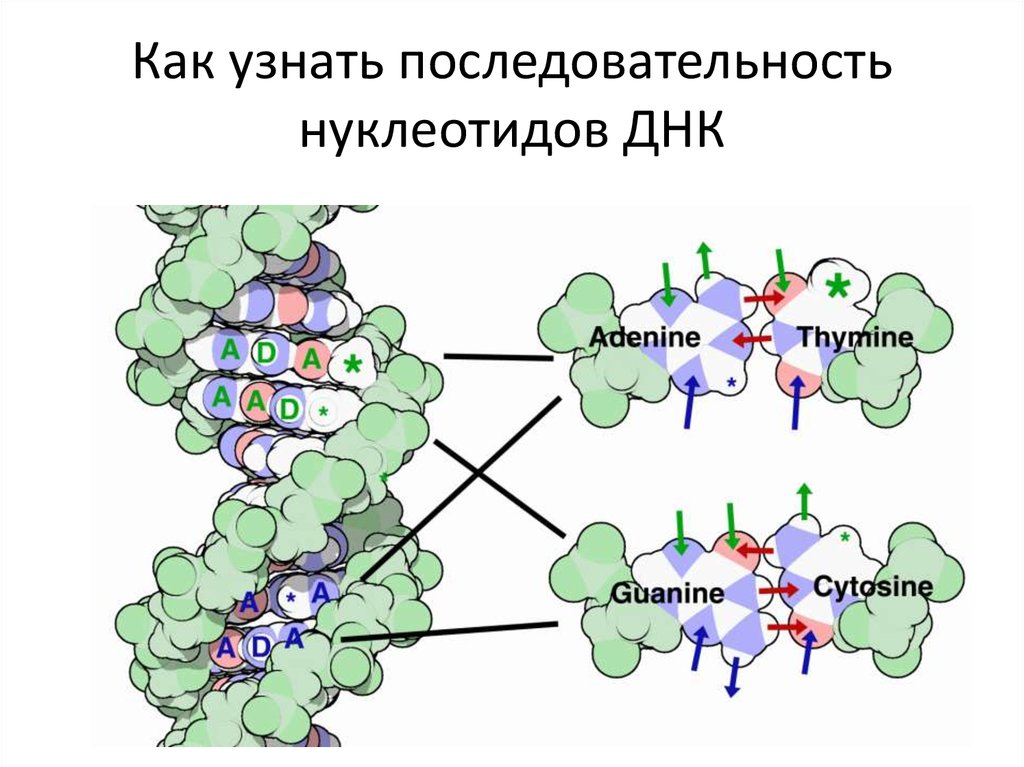
Как узнатьпоследовательность
нуклеотидов
не расплетая ДНК
101.
Как узнатьпоследовательность
нуклеотидов
не расплетая
ДНК
102. Как узнать последовательность нуклеотидов ДНК
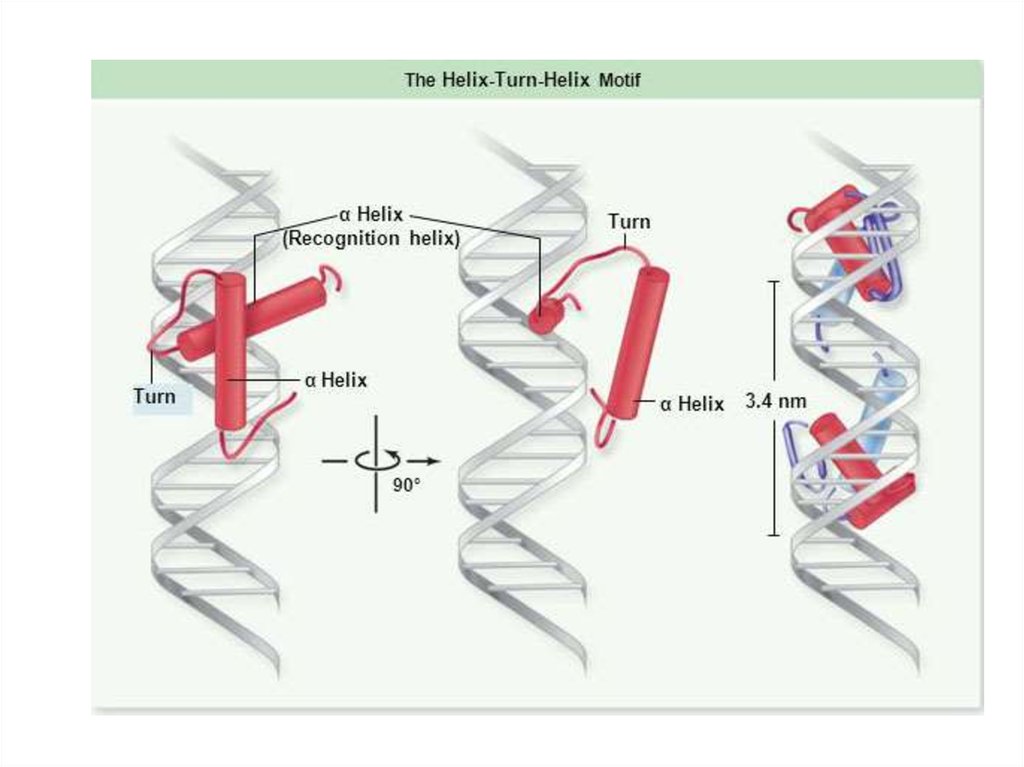
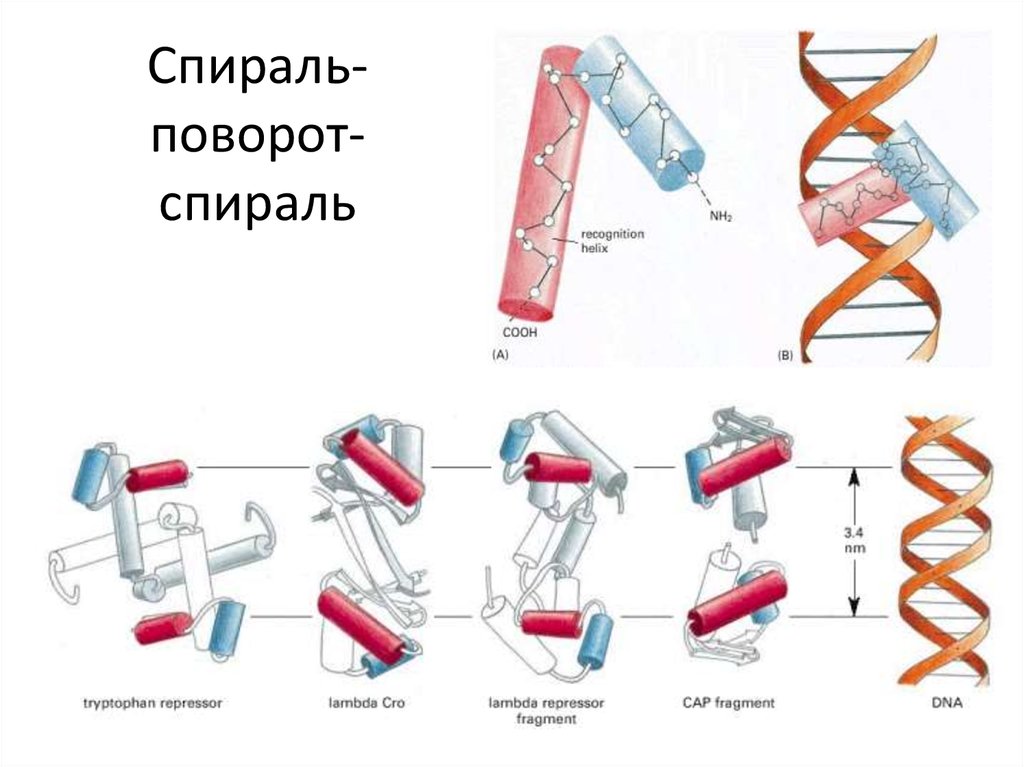
103. Спираль-поворот-спираль
Спиральповоротспираль104. Спираль-поворот-спираль
N-конецСпиральповоротспираль
С-конец
105.
106. Спираль-поворот-спираль
Спиральповоротспираль107. Lac-репрессор
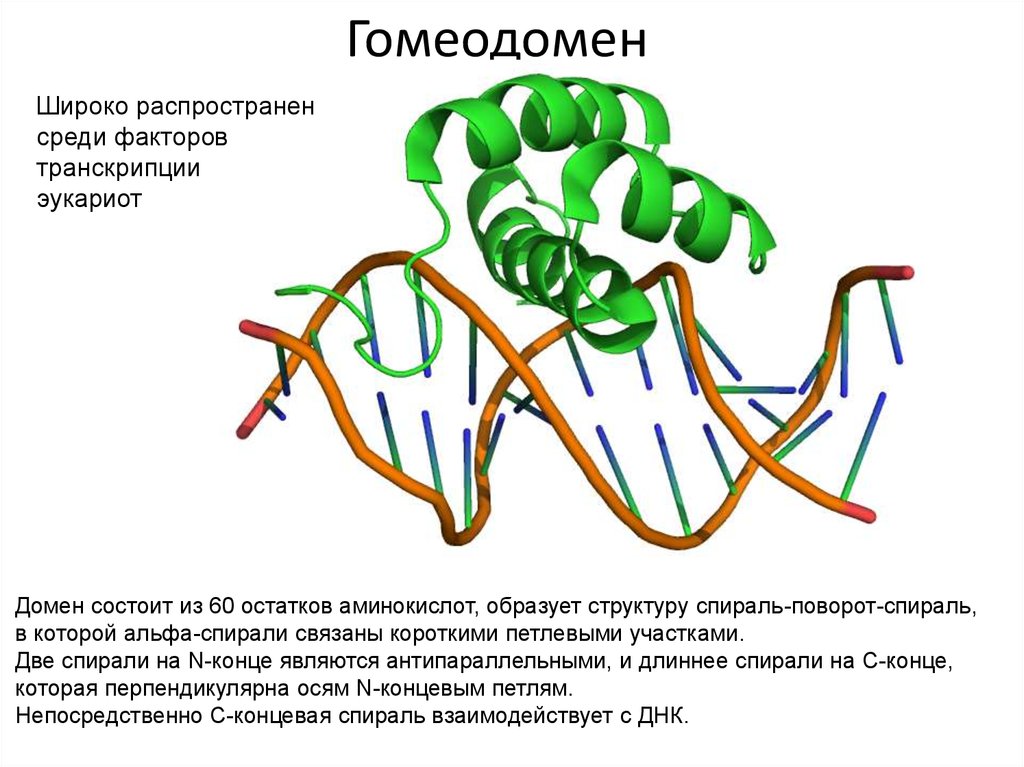
108. Гомеодомен
Широко распространенсреди факторов
транскрипции
эукариот
Домен состоит из 60 остатков аминокислот, образует структуру спираль-поворот-спираль,
в которой альфа-спирали связаны короткими петлевыми участками.
Две спирали на N-конце являются антипараллельными, и длиннее спирали на C-конце,
которая перпендикулярна осям N-концевым петлям.
Непосредственно С-концевая спираль взаимодействует с ДНК.
109. Спираль-петля-спираль
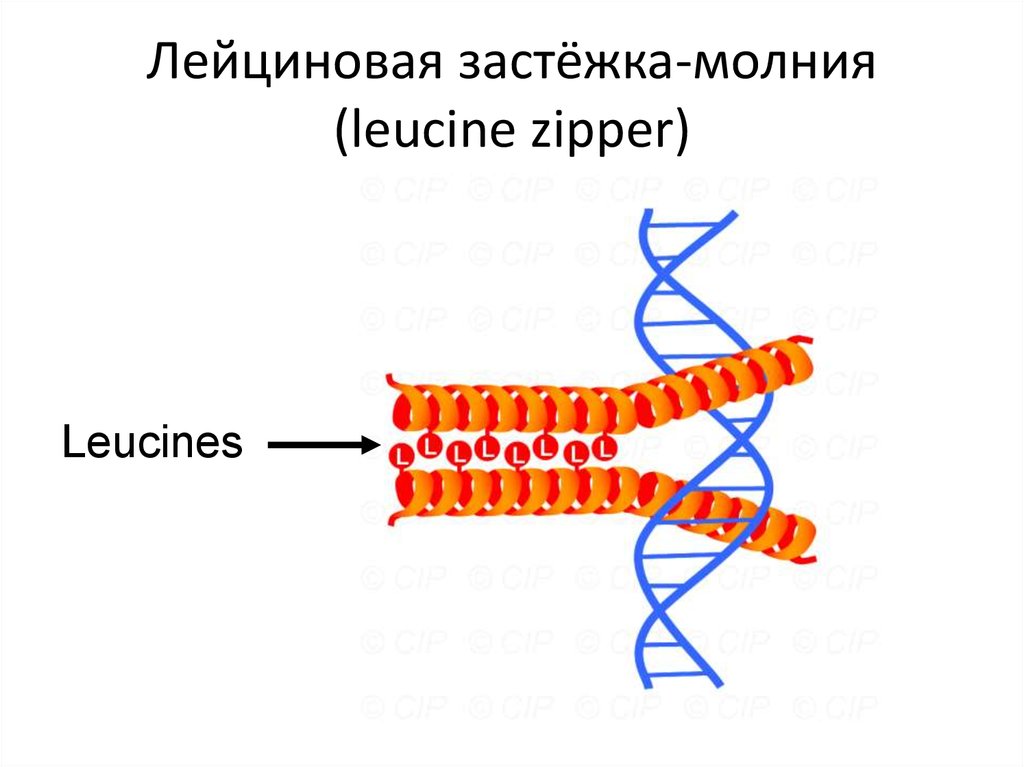
110. Лейциновая застёжка-молния (leucine zipper)
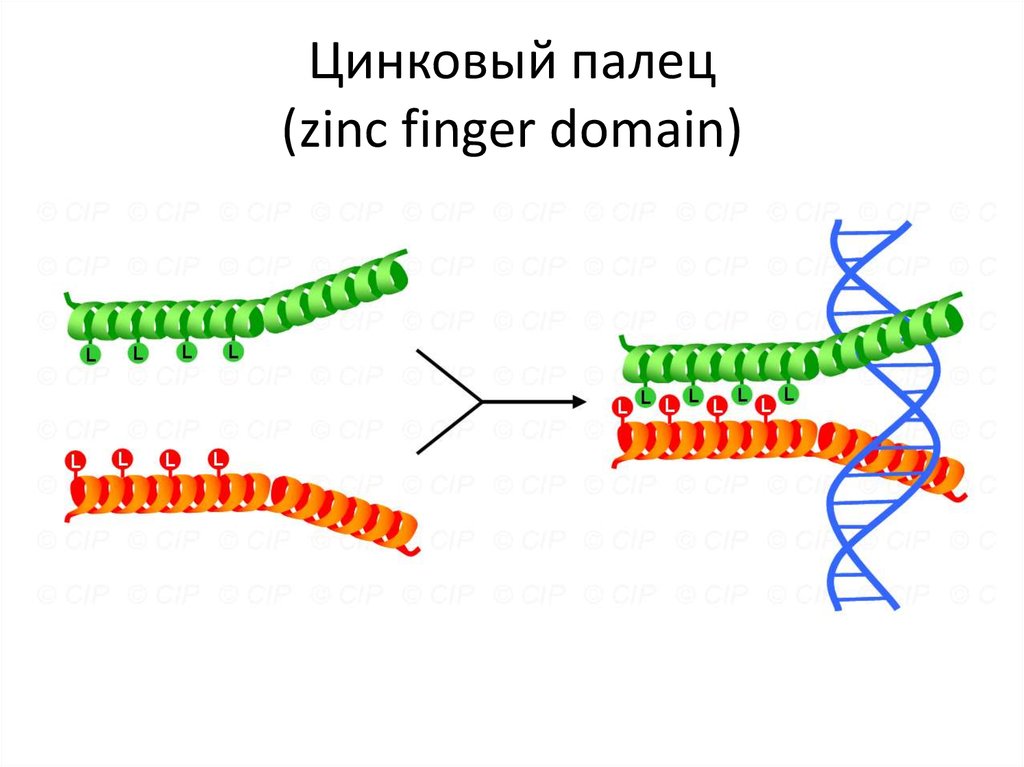
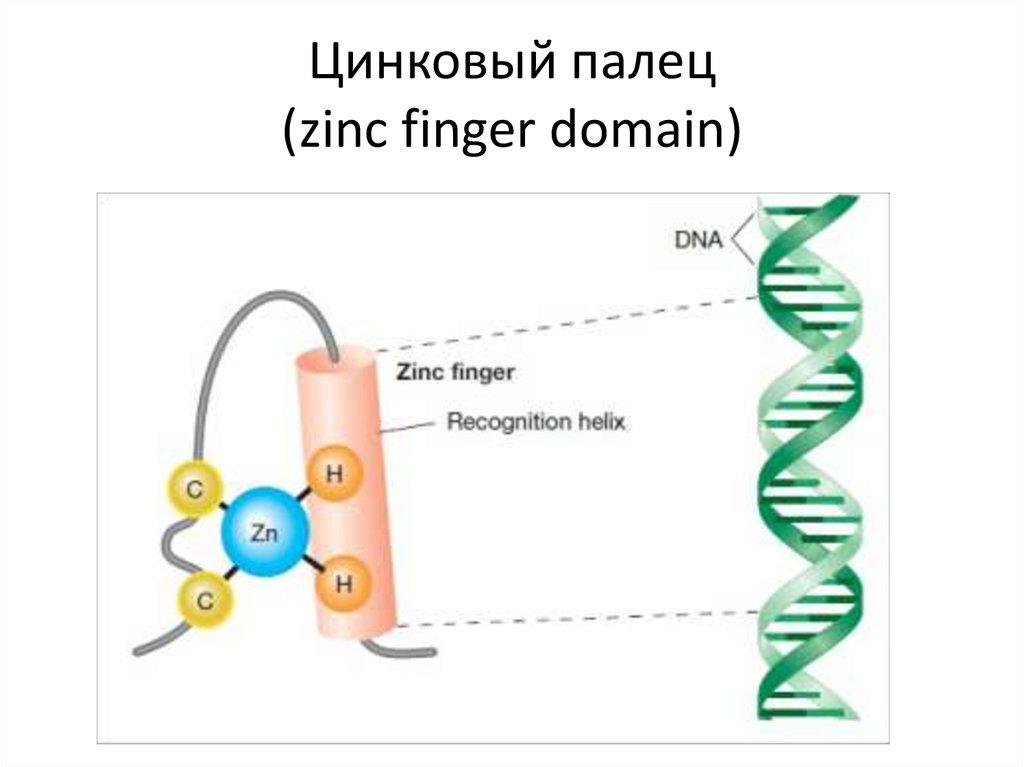
Leucines111. Цинковый палец (zinc finger domain)
112. Цинковый палец (zinc finger domain)
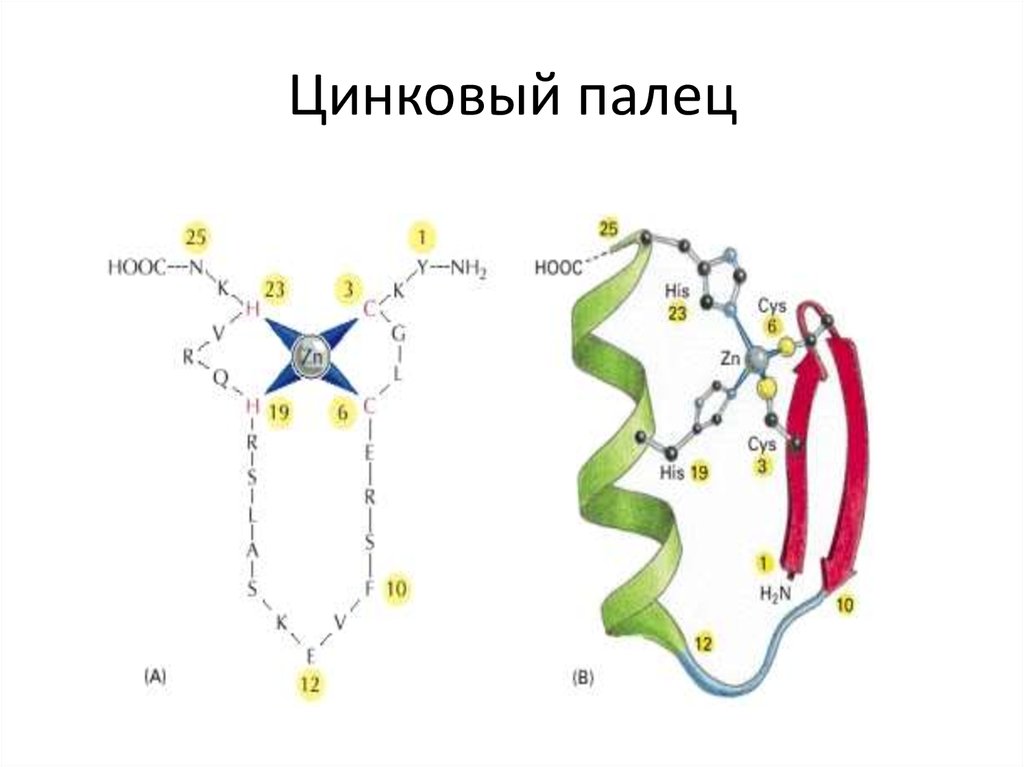
113. Цинковый палец
114.
115. Цинковый палец
116. Создание ZFN (zinc-finger nuclease)
117.
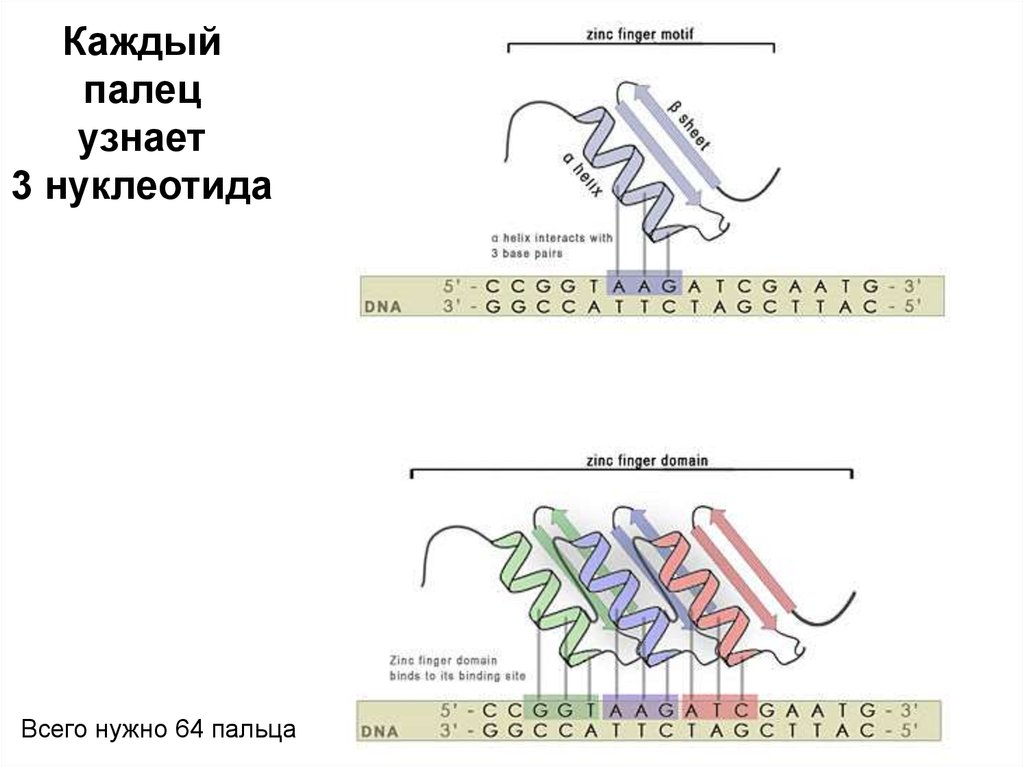
Каждыйпалец
узнает
3 нуклеотида
Всего нужно 64 пальца















































 Биология
Биология








