Похожие презентации:
Причины появления повреждений в ДНК. Лекция 3
1. DNA REPAIR
2.
3. Причины появления повреждений в ДНК
Для поддержания генетической стабильности организма необходимо не толькоточно реплицировать ДНК, но так же и исправлять возникающие в ней повреждения.
• Ошибки репликации
• Повреждения ДНК эндогенными агентами:
Гидролиз
(депуринизация, дезаминирование)
• Повреждения ДНК экзогенными агентами
облучение
повреждение химическими агентами (например,
алкилирование)
• Репликация «через повреждения» с использованием
полимераз, отличающихся низкой точностью
копирования
• и др.
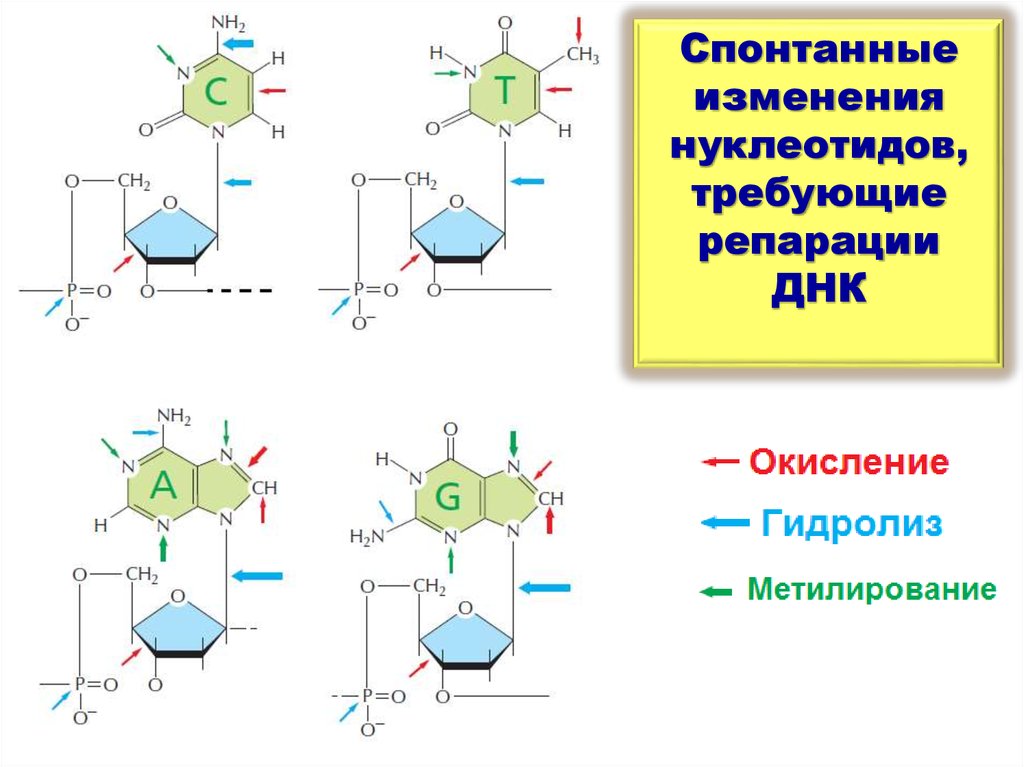
4.
Спонтанныеизменения
нуклеотидов,
требующие
репарации
ДНК
5.
Несмотря на высокую стабильность генетического материала, в нём происходятспонтанные изменения, которые приводили бы к мутациям, если их не исправлять.
Эндогенные повреждения ДНК в диплоидной клетке млекопитающих, за 24 часа.
6.
Спонтаннаядепуринизация
Дезаминирование
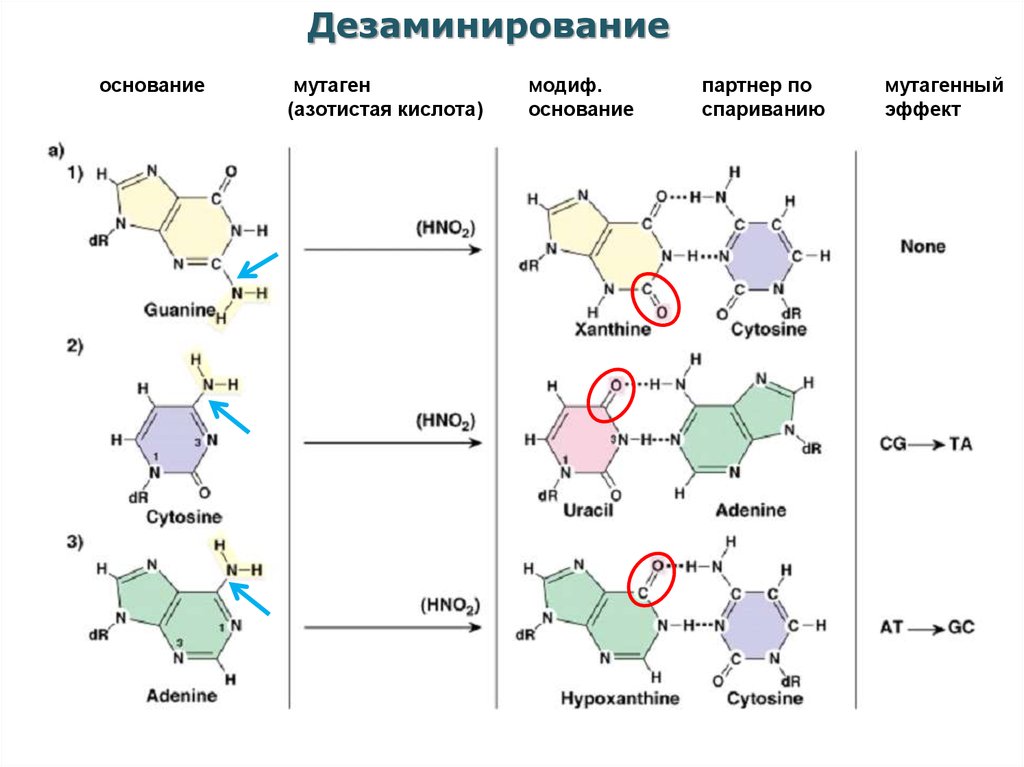
7.
Дезаминированиеоснование
мутаген
(азотистая кислота)
модиф.
основание
партнер по
спариванию
мутагенный
эффект
8.
АЛКИЛИРОВАНИЕэтилметансульфонат
(EMS)
9.
Под действием УФ света происходит образованиетимидиновых димеров
10.
11.
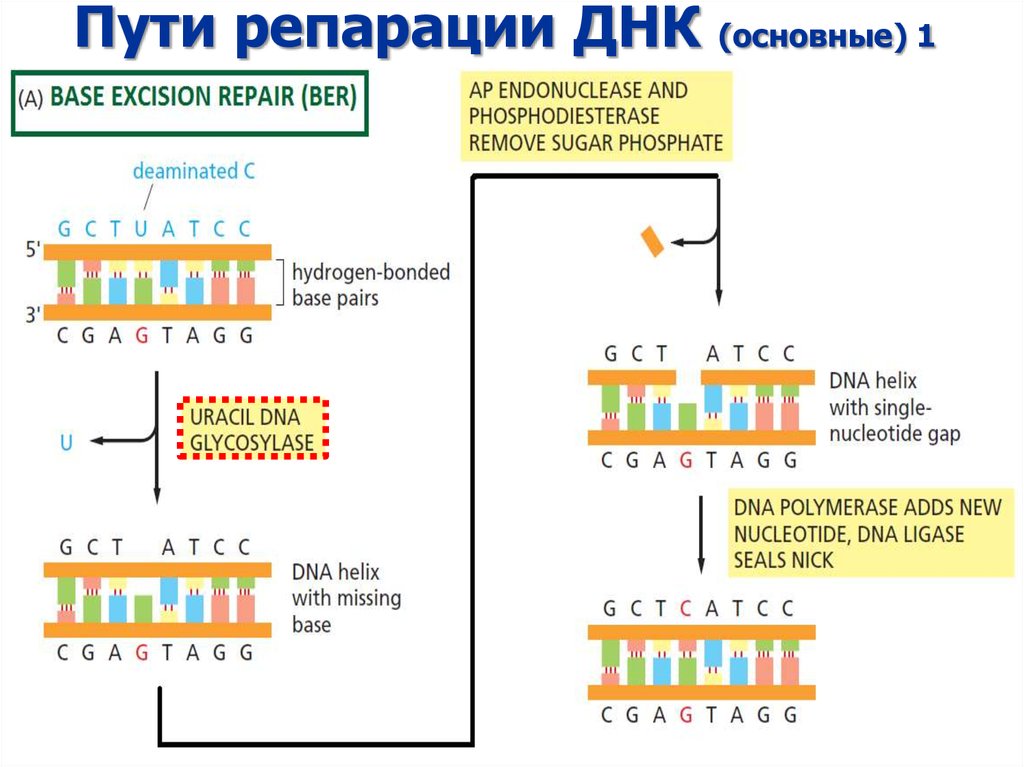
12. Пути репарации ДНК (основные) 1
13.
Как измененное основание узнается ферментом,находясь внутри двойной спирали?
Ключевую роль в этом играет
«выворачивание наружу» (“flipping-out”)
измененного нуклеотида из спирали,
что позволяет ДНК-гликозилазе
проверить основание на все виды повреждений.
Ферменты этого класса «ползают» по ДНК,
поочередно выворачивая основания
для оценки их состояния.
Если фермент узнает поврежденное основание,
то он расщепляет связь между ним и сахаром.
14. Основные типы повреждений, которые удаляются посредством BER (большая часть не блокирует репликацию)
• Окисленные основания, в том числе 8-oкси-G,который спаривается с А, вызывая GC --> TA
трансверсии
• Дезоксиурацил
• Различные продукты алкилирования оснований
(например, 3-meA)
• Спонтанно возникающие апуриновые сайты
15.
Пути репарации ДНК(основные) 2
Этот механизм используется для коррекции
«серьезных» повреждений, которые блокируют
репликацию (у человека таковыми являются,
в частности, тимидиновые димеры).
16.
У E. Coli четыре белка участвуют в эксцизии нуклеотидов: UvrA, UvrB, UvrC, UvrDУзнавание и связывание
UvrA находит повреждения и
размещает в поврежденном участке
UvrB, который вызывает
локальную денатурацию ДНК
Внесение
разрывов
UvrB привлекает UvrC.
UvrBC вносит
однонитевые разрывы
перед повреждением и
после него
Удаление поврежденного
фрагмента ДНК и
застройка бреши
Хеликаза UvrD
освобождает
вырезанный фрагмент
ДНК
17.
Названия многих белков человека,участвующих в NER происходят от названия
заболевания Xeroderma Pigmentosum
Идентифицированы 8 генов в экспериментах по комплементации
деффектов при слиянии клеток от разных больных:
XPA-XPG + hHR23B
18.
У эукариот механизм эксцизии нуклеотидов в общих чертах схожс прокариотическим, но существенно отличается в деталях
Повреждения в ДНК могут узнаваться либо особой группой белков
(global NER) либо РНК-полимеразой (transcription-coupled NER)
XPC в комплексе с hHR23B узнают
повреждения и вызывают локальную
денатурацию ДНК. XPA стабилизирует комплекс
и привлекает другие белки
XPB+XPD - субъединицы TFIIH
TFIIH является общим транскрипционным
фактором, обладающим хеликазной активностью.
В данном случае TFIIH расширяет локальноденатурированный участок
ERCCI-XPF – эндонуклеаза, вносящая 5’-разрыв
XPG – эндонуклеаза, вносящая 3’-разрыв
RPA помогает позиционировать нуклеазы по
краям расплавленного участка ДНК
Функции XPE не понятны. In vitro этот белок не
нужен
19.
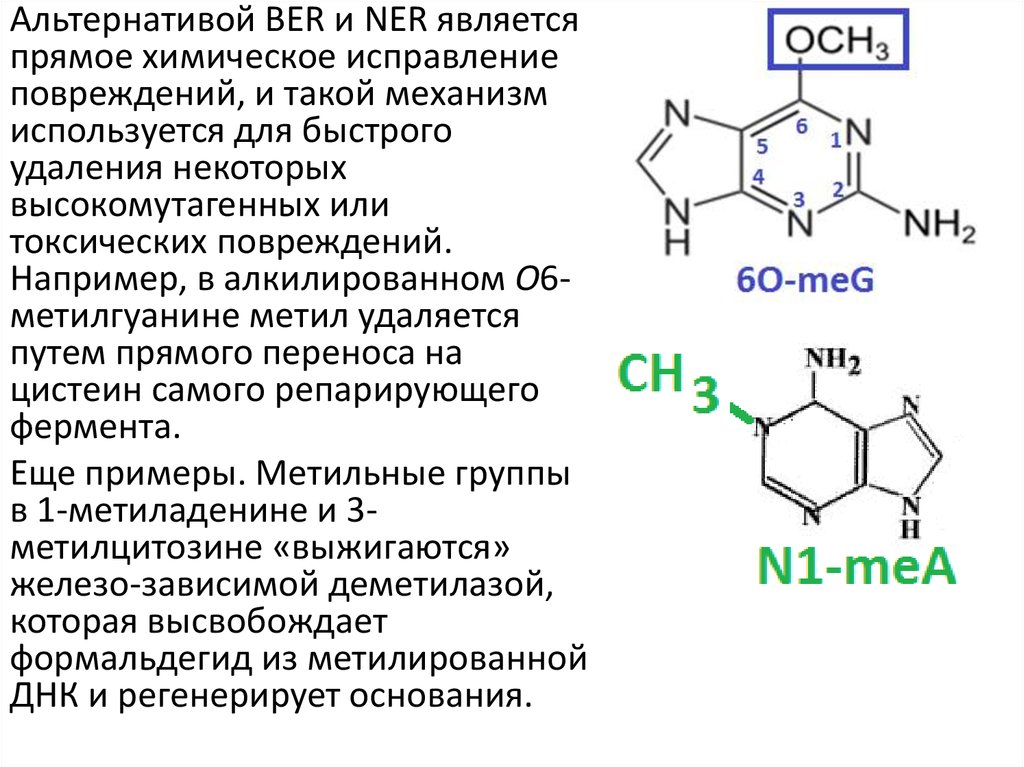
Альтернативой BER и NER являетсяпрямое химическое исправление
повреждений, и такой механизм
используется для быстрого
удаления некоторых
высокомутагенных или
токсических повреждений.
Например, в алкилированном O6метилгуанине метил удаляется
путем прямого переноса на
цистеин самого репарирующего
фермента.
Еще примеры. Метильные группы
в 1-метиладенине и 3метилцитозине «выжигаются»
железо-зависимой деметилазой,
которая высвобождает
формальдегид из метилированной
ДНК и регенерирует основания.
20.
Повреждения ДНКмогут вызвать
остановку
элонгирующей
РНК-полимеразы
Ферменты NER
узнают такой
задержанный
комплекс и
процессируют его
подобно
комплексу XPA-XPC –
hHR23B
21.
XPC damagerecognition
XPB & XPD =
TFII H DNA
helicase
XPA stabilizes ssDNA fragment
22.
ERCC1-XPF - 5’ incisionXPG - 3’ incision
23. Исправление ошибок репликации (Мismatch repair)
• ДНК полимеразы (даже те, у которых естькорректирующая активность) все равно делают ошибки,
которые надо исправлять
• Система репарации ошибок репликации должна
• 1. Быстро находить ошибки
• 2. Различать родительскую и новосинтезированную
цепь с тем, чтобы в неспаренном участке
заменить ошибочно включенный нуклеотид
24.
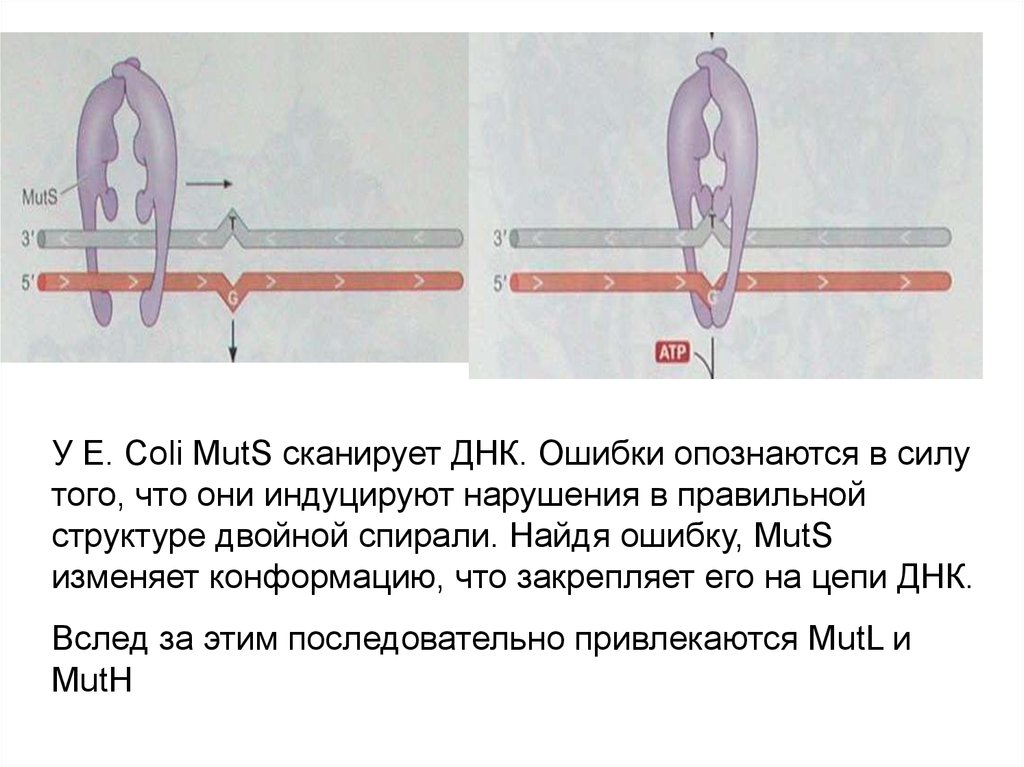
У E. Coli MutS сканирует ДНК. Ошибки опознаются в силутого, что они индуцируют нарушения в правильной
структуре двойной спирали. Найдя ошибку, MutS
изменяет конформацию, что закрепляет его на цепи ДНК.
Вслед за этим последовательно привлекаются MutL и
MutH
25. Что узнается ?
• MutS димер (у дрожжей, Msh2/Msh3 или Msh2/Msh6гетеродимер)
• Эксперименты по связыванию с ДНК in vitro и
репарации гетеродуплексов in vivo показали, что MMR
узнает все комбинации неспаренных оснований,
кроме C:C, а также короткие <4 п.н. делеции и
инсерции («инделы»)
• неправильные пары G:T and A:C и инсерции/делеции в
1 п. особенно хорошо узнаются. Эти нарушения
являются наиболее частыми ошибками ДНКполимераз
26.
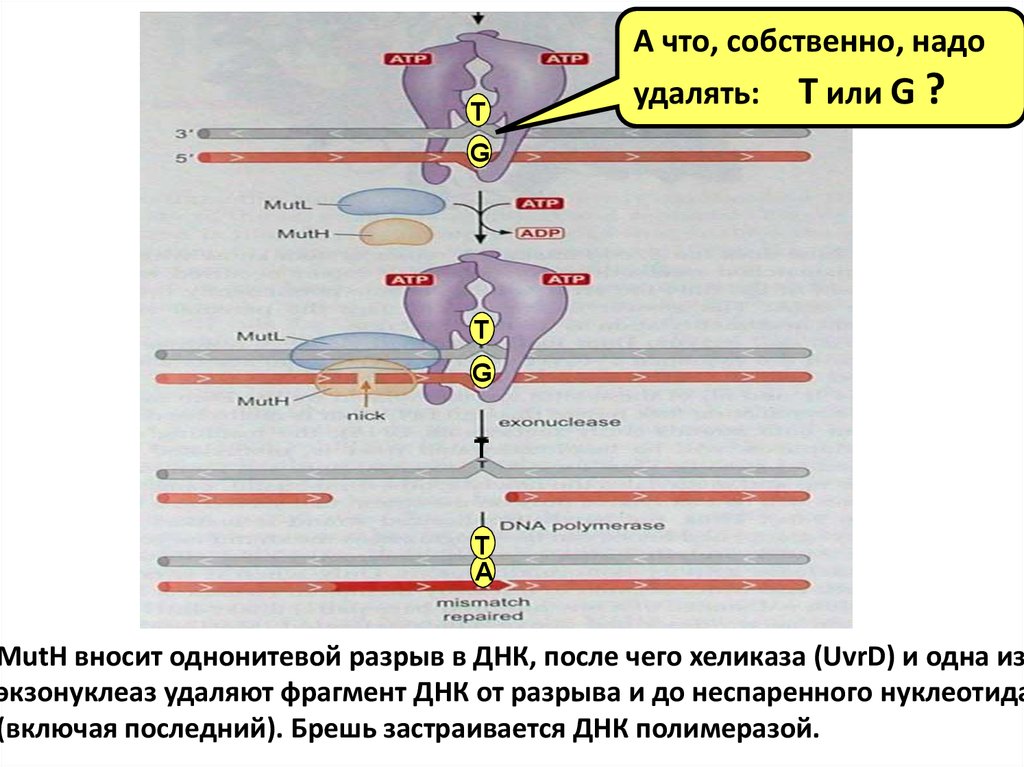
А что, собственно, надоT
удалять:
T или G ?
G
T
G
T
T
A
MutH вносит однонитевой разрыв в ДНК, после чего хеликаза (UvrD) и одна из
экзонуклеаз удаляют фрагмент ДНК от разрыва и до неспаренного нуклеотида
(включая последний). Брешь застраивается ДНК полимеразой.
27.
По логике событий ошибочно включенный нуклеотид должен находиться вновосинтезированной цепи ДНК. Эта цепь опознается благодаря отсутствию
Dam метилирования. MutH связывается только с неметилированной цепью ДНК
и вносит разрыв именно в эту цепь
GATC
CTAG
28.
В эукариотических клетках также существует системакоррекции ошибок репликации
Обнаружены гомологи MutS и MutL;
гомолога MutH не обнаружено
гомологи MutS (MSH — MutS homolog) образуют два
гетеродимерных комплекса:
♣ MSH2-MSH6 (MutSα) узнает неспаренные нуклеотиды
и короткие «инделы»
♣ MSH2-MSH3 (MutSβ) узнает длинные «инделы»
Механизм распознавания новосинтезированной цепи не известен
29.
Микросателлитная нестабильностьКлетки с поврежденной системой исправления ошибок
показывают повышенную степень микросателлитной нестабильности
Эта нестабильность выражается в изменениях в малых
повторяющихся последовательностях ДНК, таких как
(САС)n, (CA)n, (A)n и др.
8 nt
8 nt
микросателлит
MMR+
slippage
MMR--
10 nt
30.
А если не успели всё починить,а ДНК уже реплицируется?
31.
Обход препятствия посредством смены матричныхцепей (продолжение репликации с сохранением
ошибки в одной из родительских цепей)
Вариант 1
Вариант 2
32.
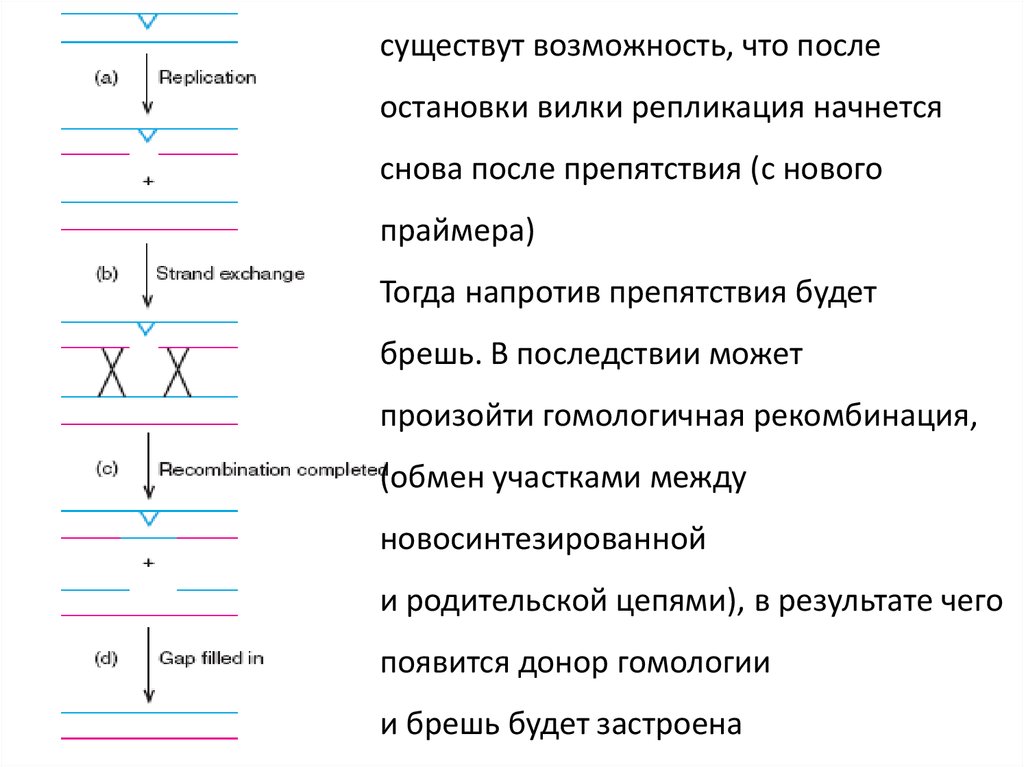
существут возможность, что послеостановки вилки репликация начнется
снова после препятствия (с нового
праймера)
Тогда напротив препятствия будет
брешь. В последствии может
произойти гомологичная рекомбинация,
(обмен участками между
новосинтезированной
и родительской цепями), в результате чего
появится донор гомологии
и брешь будет застроена
33.
Special Translesion (черезблоковые) DNAPolymerases Are Used in Emergencies
34.
«черезблоковый» синтез катализируемой«мутасомой» (DNA pol V)
Cowcatcher model
Locomotive
cowcatcher
(скотосбрасыватель)
35.
При наличии препятствия на пути ДНК поллимеразы III,эта полимераза диссоциирует от ДНК. Хеликаза продолжает
работать, генерируя однотитевой участок, с которым связывается
RecA
Pol V связывается со свободным «праймером». Для эффективного
связывания необходим контакт с Rec A и с бета-зажимом
Непосредственно в месте повреждения с ДНК связывается SSB
Pol V начинает синтез ДНК, одновременно вытесняя Rec A
филамент. Одновременно происходит процессивная диссоциация
Rec A филамента с противоположного конца, сопряженная с
гидролизом ATФ.
После удаления всего филамента Rec A Pol V диссоциирует от ДНК,
освобождая место для Pol III. Таким образом время работы Pol V
определяется временем существования Rec A филамента.
Pol V успевает включить несколько нуклеотидов. Напротив
тимидинового димера ТТ чаще всего включается GA
36. Эукариотические ДНК полимеразы
АР сайты (встраивает А)37. Репарация двунитевых разрывов (SOS)
Двунитевые разрывы в ДНК возникают:под действием ионизирующего излучения
под действием некоторых химических агентов, в частности,
ингибиторов ДНК топоизомеразы II
Существует два основных пути репарации
двунитевых разрывов:
гомологичная рекомбинация
негомологичное соединение концов ДНК (NHEJ)
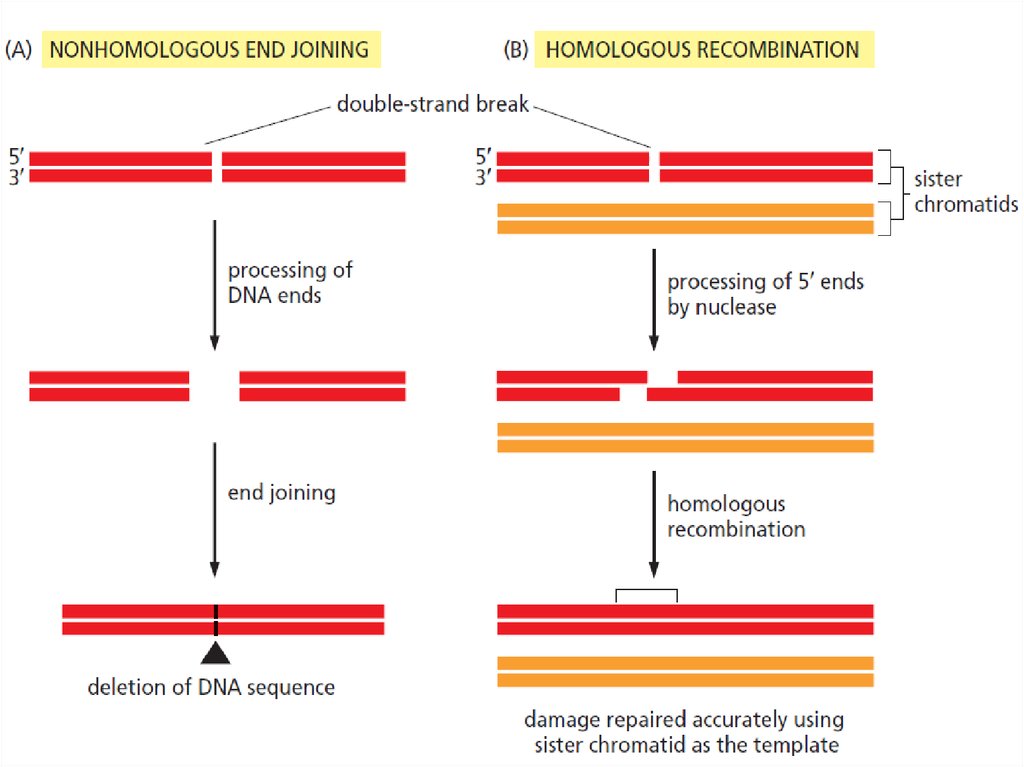
38.
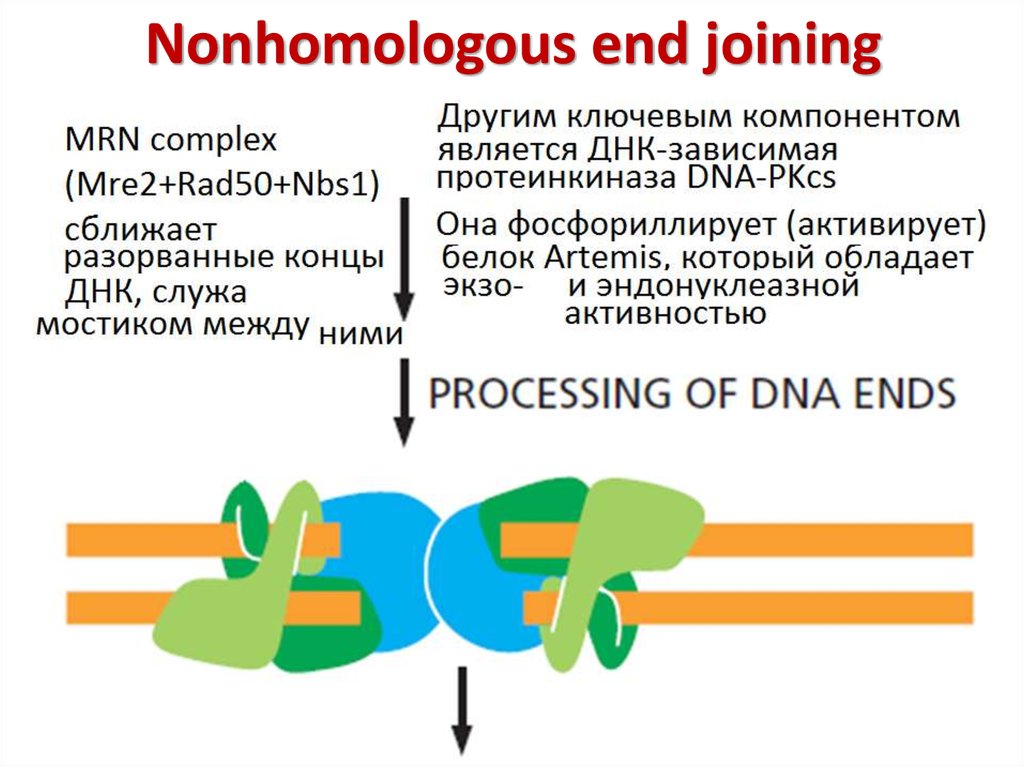
39. Nonhomologous end joining
Ku70/Ku80 гетеродимеры связываются с разрывами40. Nonhomologous end joining
41. Nonhomologous end joining
42. Nonhomologous end joining (summary)
43. Homologous recombination
Эксперимент поренатурации/гибридизации
Однако in vivo вся ДНК находится в двунитевой
форме, и механизм спаривания другой (далее)
гетеродуплекс
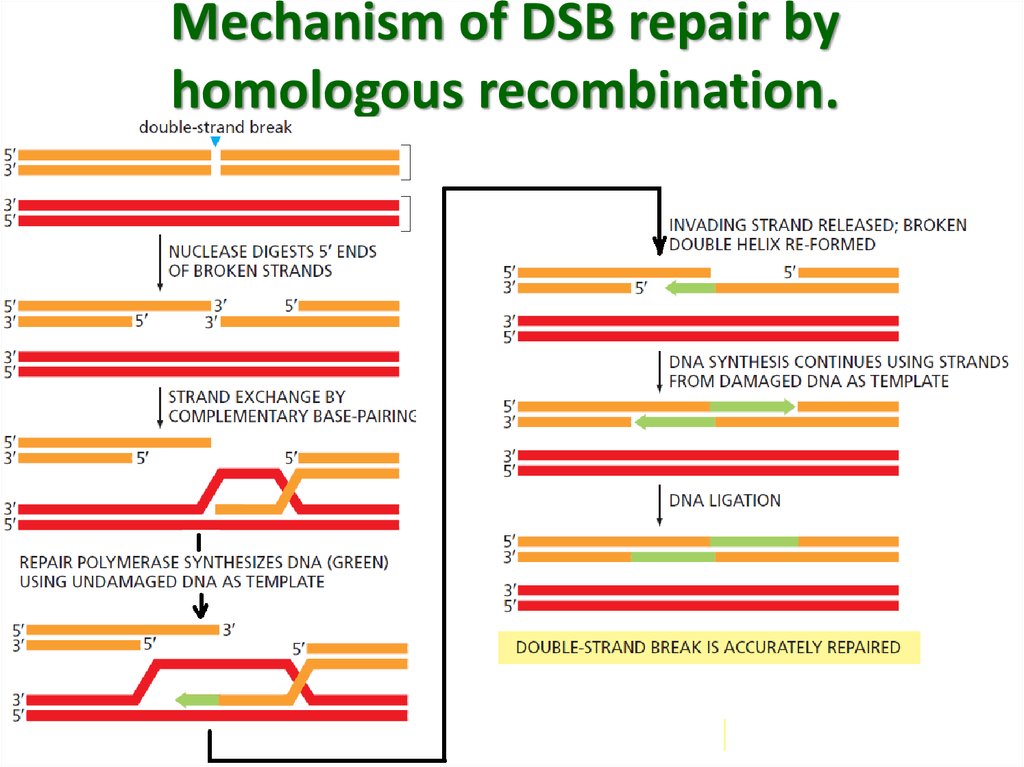
44.
В отличие от NHEJ-механизма, при гомологичнойрекомбинации требуется поврежденную цепь привести
в соприкосновение с неповрежденной гомологичной
цепью, которая будет служить в качестве образца.
Поэтому ГР может происходить сразу после репликации
ДНК, когда дочерние цепи лежат близко друг к другу.
45. Mechanism of DSB repair by homologous recombination.
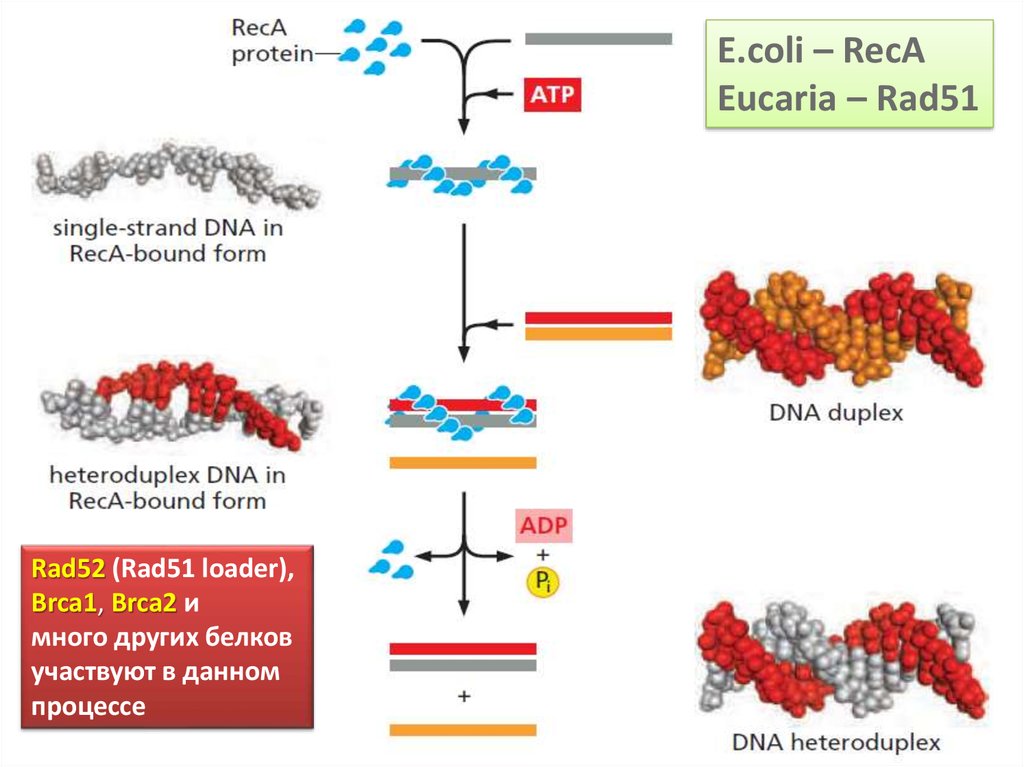
46.
E.coli – RecAEucaria – Rad51
Rad52 (Rad51 loader),
Brca1, Brca2 и
много других белков
участвуют в данном
процессе
47. Repair of a broken replication fork by homologous recombination
48. Потеря гетерозиготности
• Иногда при ГР используется не сестринская, агомологичная копия (вместо исправления
материнской разорванной хромосомы
используется не сестринская материнская, а
отцовская гомологичная хромосома и наоборот).
• Поскольку последовательности ДНК в гомологах
различаются, такой тип исправления может
привести к конверсии участка материнской
хромосомы в отцовский (и наоборот). Такая
конверсия называется потерей гетерозиготности и
может в некоторых случаях (если в отцовской
хромосоме в данном участке присутствуют
онкомутации) приводить к онкозаболеваниям.
49.
• BRCA1 и BRCA2 – участники гомологичнойрекомбинации. Мутации в них ведут к
наследственным формам рака груди и
яичника.
• BRCA1 регулирует ранний процессинг концов в
разорванной ДНК, и без него не происходит
должной подготовки DSB к ГР, вместо этого
такой разрыв репарируется по механизму
NHEJ, вносящим мутации.
• BRCA2 связывает Rad51 и не дает ему
полимеризоваться на ДНК без надобности,
поддерживая его в неактивной форме до той
поры, пока он не будет нужен. При
обнаружении поврежденной ДНК BRCA2
быстро доставляет Rad51 к месту повреждения
и позиционирует его на ssDNA активный
Rad51.
50.
Около 10%Почти 90% of DSB
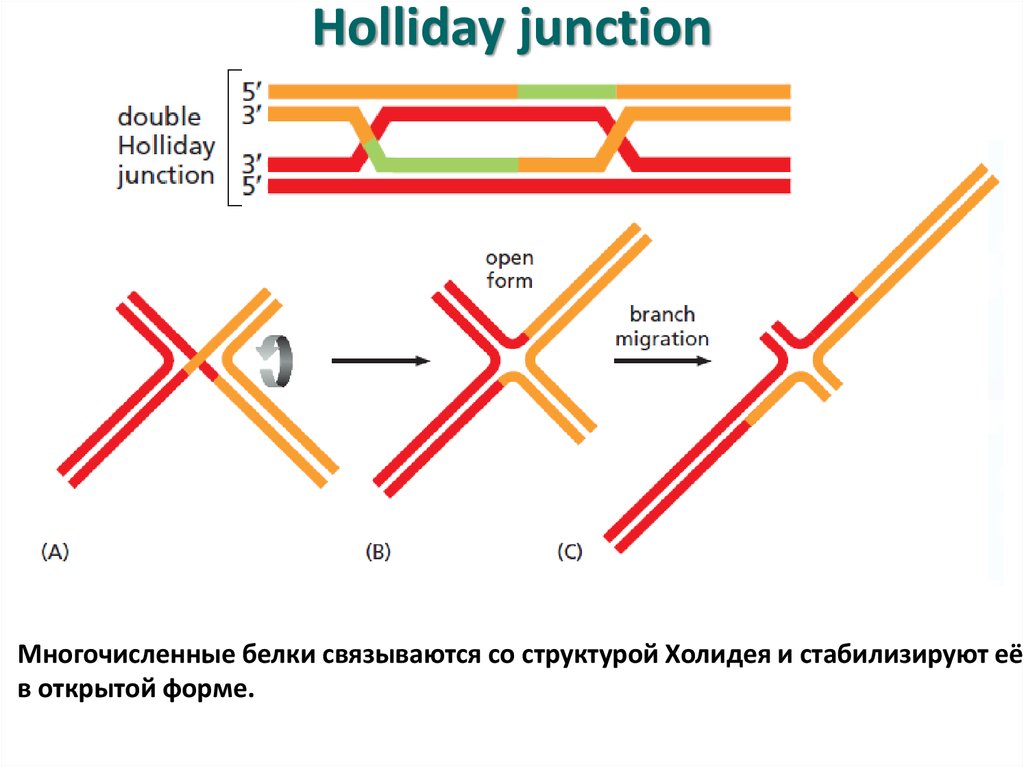
51. Holliday junction
Многочисленные белки связываются со структурой Холидея и стабилизируют еёв открытой форме.
52.
Branch migration53. Enzyme-catalyzed branch movement at a Holliday junction by branch migration
54.
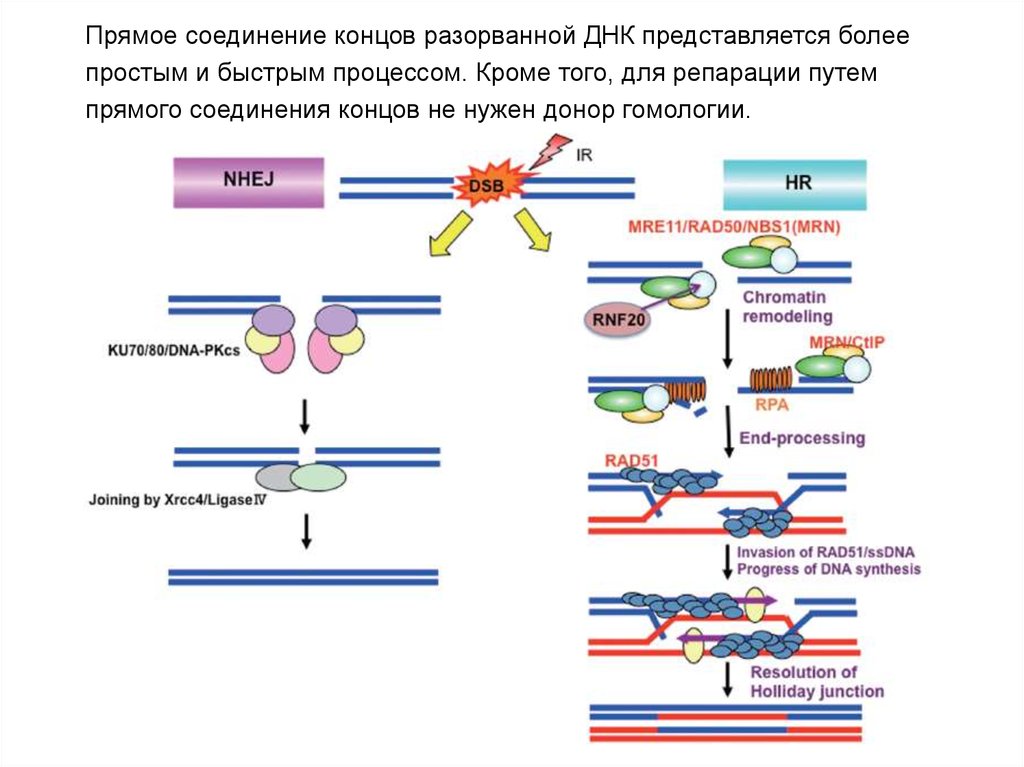
Прямое соединение концов разорванной ДНК представляется болеепростым и быстрым процессом. Кроме того, для репарации путем
прямого соединения концов не нужен донор гомологии.
55.
• Между хромосомамивозникает только несколько
перекрестов.
• Перекрест в одной позиции
ингибирует образование
перекреста посоедству –
это так называемый
«crossover control».
• С другой стороны,
каждая пара хромосом,
невзирая на их размер,
образует хотя бы один
перекрест.
• Для большинства
организмов в мейозе
каждая хромосома
образует два перекреста –
по одному на каждое плечо.
56. ГР: кроссинговер и генная конверсия
57.
58.
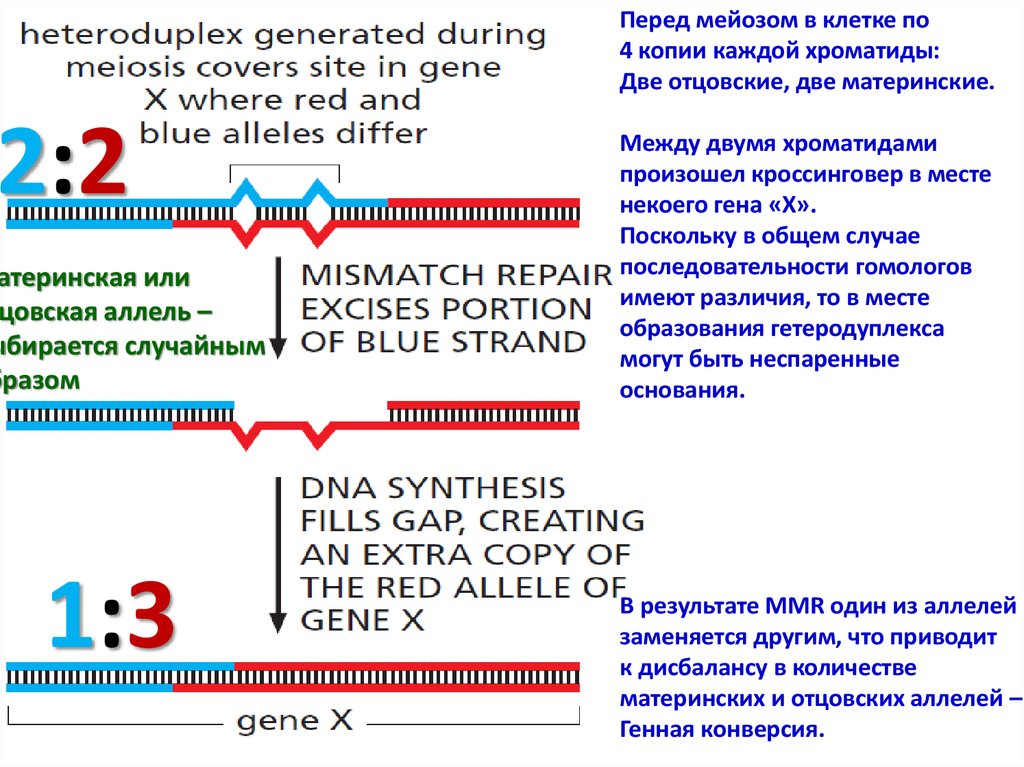
Перед мейозом в клетке по4 копии каждой хроматиды:
Две отцовские, две материнские.
2:2
атеринская или
тцовская аллель –
ыбирается случайным
бразом
1:3
Между двумя хроматидами
произошел кроссинговер в месте
некоего гена «Х».
Поскольку в общем случае
последовательности гомологов
имеют различия, то в месте
образования гетеродуплекса
могут быть неспаренные
основания.
В результате MMR один из аллелей
заменяется другим, что приводит
к дисбалансу в количестве
материнских и отцовских аллелей –
Генная конверсия.
59. TRANSPOSITION AND CONSERVATIVE SITE-SPECIFIC RECOMBINATION
60.
Мобильные генетические элементы(транспозоны)
ДНК транспозоны (у прокариот и эукариот)
для перемещения нужна транспозаза (интеграза)
возможно простое перемещение (без увеличения
числа копий) либо создание новых копий
Ретро-транспозоны (у эукариот)
перемещение всегда посредством создания новой
копии
промежуточная РНК-копия
обратная транскриптаза
61.
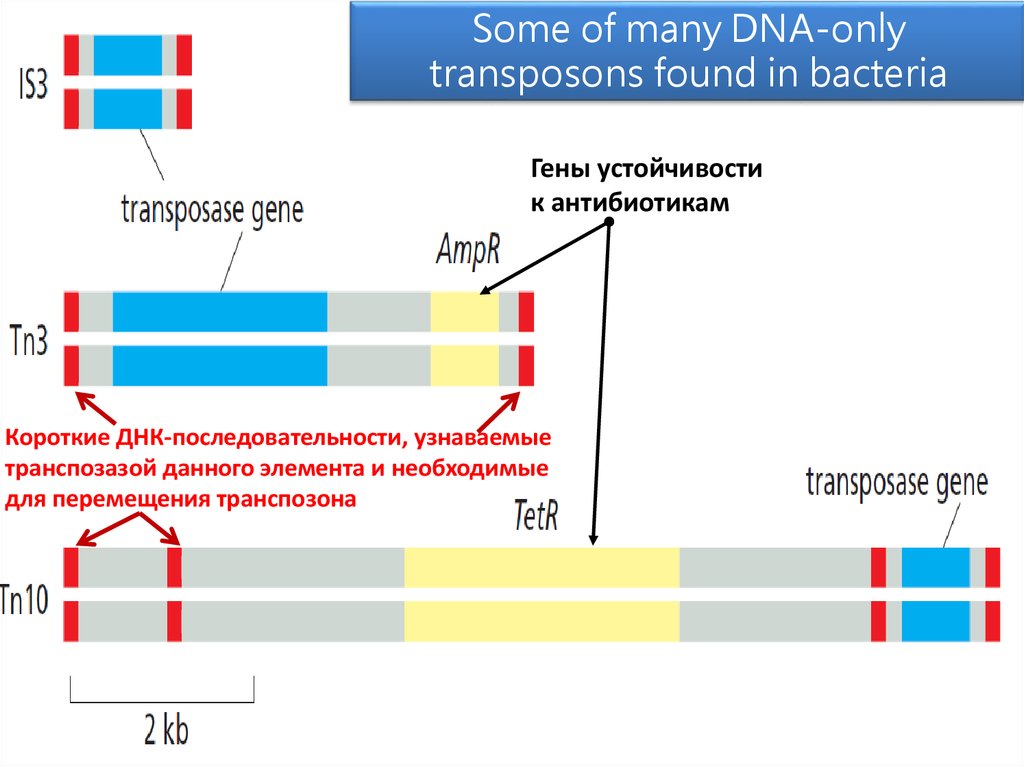
Some of many DNA-onlytransposons found in bacteria
Гены устойчивости
к антибиотикам
Короткие ДНК-последовательности, узнаваемые
транспозазой данного элемента и необходимые
для перемещения транспозона
62.
Так мобильные элементы сгенами устойчивости к
антибиотикам могут
передаваться горизонтальным
переносом
63.
64.
65. retroviral-like retrotransposons
Сходны с ретровирусами, но сами не могут покидатьхозяйскую клетку.
1. Весь ретротранспозон транскрибируется системой
транскрипции хозяйской клетки.
2. После трансляции с него синтезируется обратная
транскриптаза, которая синтезирует на dsDNA на
матрице РНК, используя РНК-ДНК гибрид в качестве
интермедиата.
3. Как и ретровирус, линейная dsDNA теперь интегрируется в геном хозяйской клетки, используя закодированную в ретротранспозоне интегразу.
4. Теперь мобильный элемент может передаваться
потомству клетки.
66. nonretroviral retrotransposons
Третий большой класс мобильных элементов, большинство изкоторых в геноме человека неподвижно, однако некоторые
представители являются подвижными. Например L1-элемент
(от аббревиатуры LINE - Long Interspersed Nuclear Element
(ок. 500 тыс копий)
L1-элемент ответственнен за один из типов гемофилии,
возникающей вследствие встраивания этого элемента
в ген участвующего в свертывании крови белка
Фактор VIII (Антигемофильный глобулин)
67.
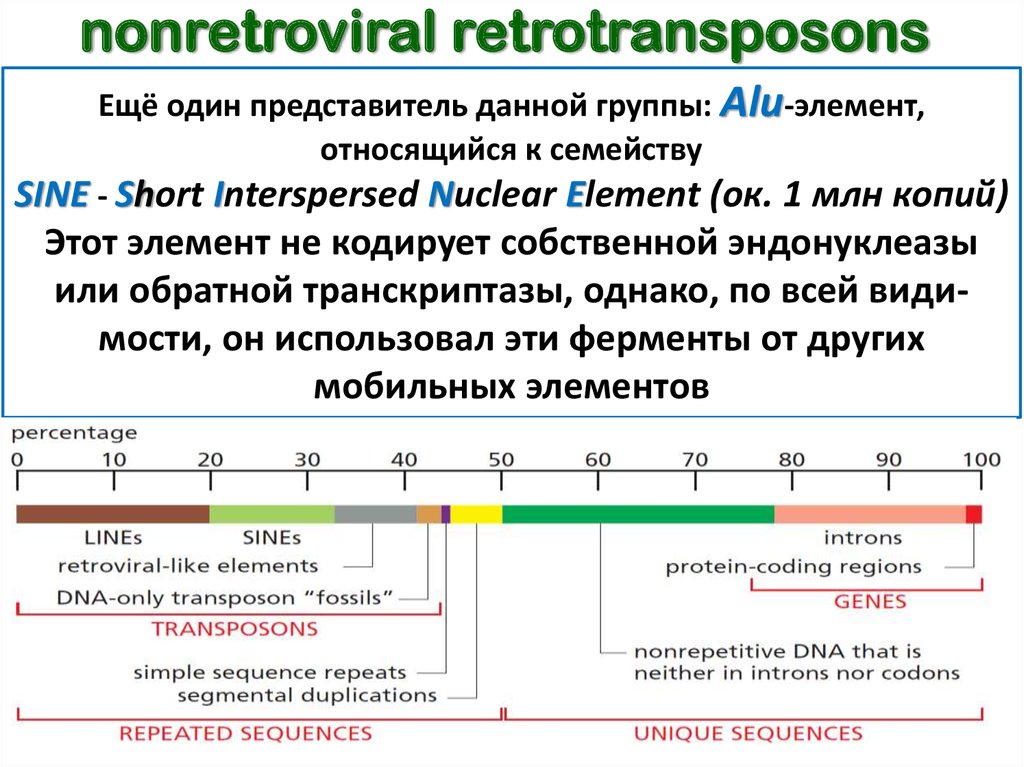
68. nonretroviral retrotransposons
Ещё один представитель данной группы: Alu-элемент,относящийся к семейству
SINE - Short Interspersed Nuclear Element (ок. 1 млн копий)
Этот элемент не кодирует собственной эндонуклеазы
или обратной транскриптазы, однако, по всей видимости, он использовал эти ферменты от других
мобильных элементов
69.
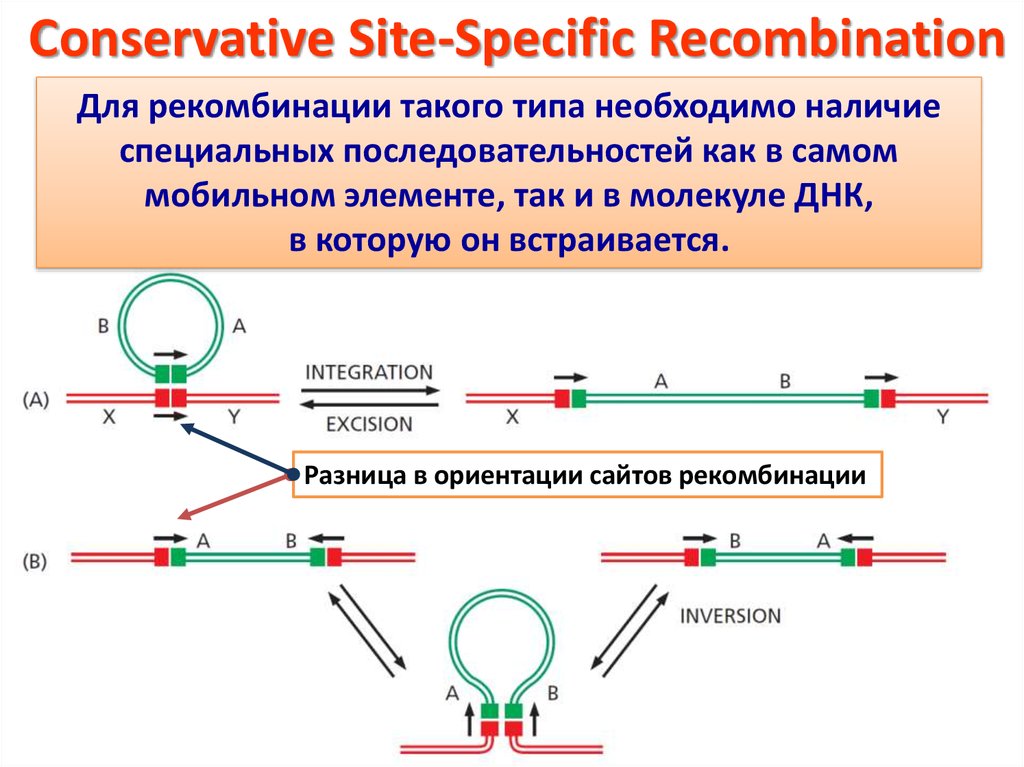
Conservative Site-Specific RecombinationДля рекомбинации такого типа необходимо наличие
специальных последовательностей как в самом
мобильном элементе, так и в молекуле ДНК,
в которую он встраивается.
Разница в ориентации сайтов рекомбинации
70. Сайт-специфическая рекомбинация используется некоторыми ДНК-содержащими вирусами
Сайт-специфическая рекомбинацияиспользуется некоторыми ДНКсодержащими вирусами
• При этом вирус встраивает свою ДНК в ДНК
клетки-хозяина, беспрепятственно
реплицируется в ней (как часть клеточной
ДНК) и передается ничего не подозревающей
клеткой её потомкам.
• Однако при неблагоприятных обстоятельствах
ДНК вируса может вырезаться из клеточной и
«покидать тонущий корабль» ввиде
собравшихся вирусных частиц.
71. Отличия сайт-специфической рекомбинации от транспозиции
• SSR требует наличия специальных сайтов у обоихучастников процесса (сайт-специфичность). Для
транспозиции необходимо наличие специальных
(узнаваемых рекомбиназой) лишь у транспозона,
последовательность же хозяйской ДНК для
большинстватранспозонов может быть любой.
• Сами механизмы фундаментально различны.
В SSR рекомбиназа действует подобно топоизомеразам,
образовывая транзиентную высокоэнергетичную
ковалентную связь с ДНК, используя энергию этой связи
для перегруппировки ДНК. При этом все разорванные
фосфодиэфирные связи к концу процесса возвращаются
на прежнее место (консервативность). Транспозиция не
действует по такому механизму и после неё зачастую
остаются бреши в ДНК, которые должны быть
репарированы ДНК-полимеразами.
72. Консервативная SSR может использоваться для включения/выключения генов
Используется бактерией Salmonella для«фазовой вариации» - переключает синтез
поверхностного антигена – флагеллина –
с одной формы на другую






















































 Биология
Биология








