Похожие презентации:
Компьютерные методы анализа нуклеотидных последовательностей
1. Компьютерные методы анализа нуклеотидных последовательностей
Биоинформационные подходы,позволяющие планировать эксперименты.
Обзор биоинформационных подходов и
программ для планирования эксперимента.
Физические карты геномов. Анализ
рестрикционных карт геномов.
2.
Существует много программ, интегрирующих различныебиоинформационные инструменты и позволяющие
молекулярному биологу планировать эксперименты и
обрабатывать их результаты. Это такие программы, как
• Vector NTI (коммерческая)
• SnapGene (коммерческая)
• SimVector (коммерческая)
• Geneious (коммерческая)
• DNASTAR Lasergene (коммерческая)
• Unipro UGENE (свободная)
• BioEdit (свободная)
• DNA Master (свободная)
• и др.,
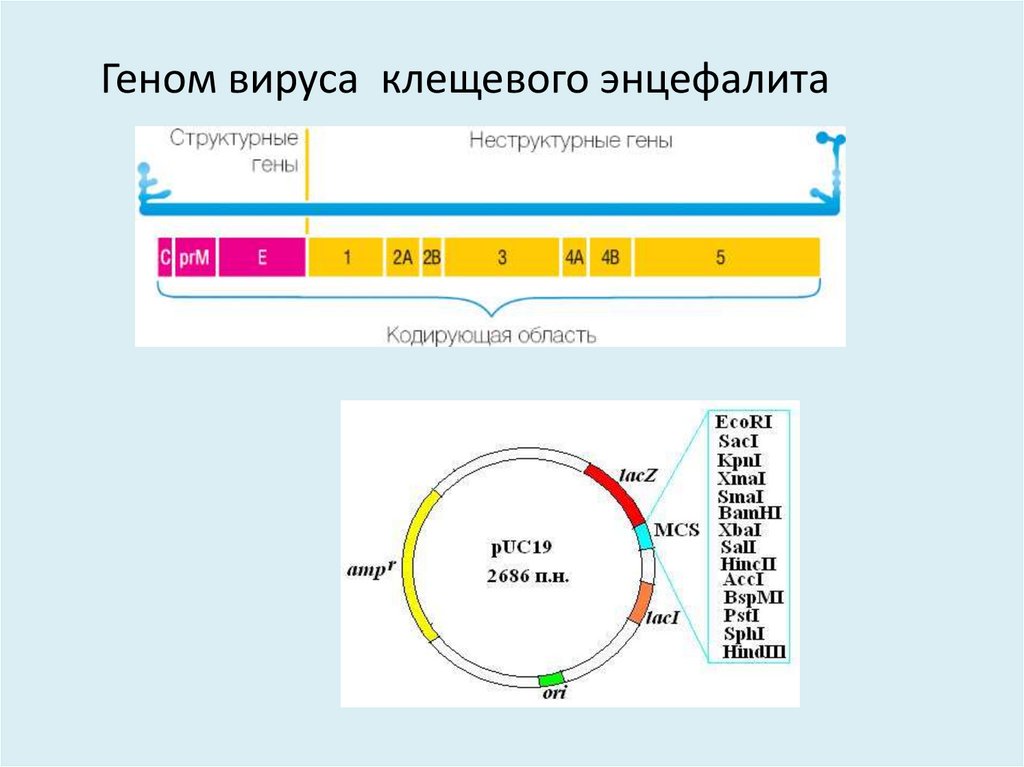
3. Геном вируса клещевого энцефалита
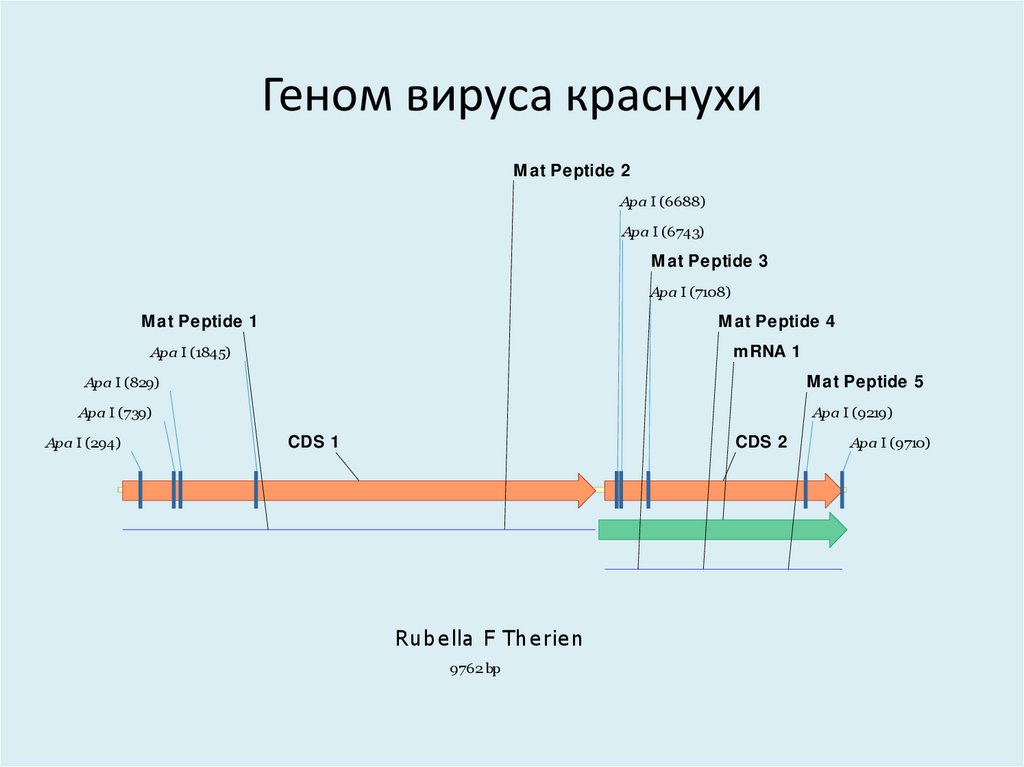
Геном вируса клещевого энцефалита4. Геном вируса краснухи
M at Peptide 2Apa I (6688)
Apa I (6743)
M at Peptide 3
Apa I (7108)
M at Peptide 4
M at Peptide 1
Apa I (1845)
mRNA 1
Apa I (829)
M at Peptide 5
Apa I (9219)
Apa I (739)
Apa I (294)
CDS 2
CDS 1
Rube lla F The rie n
9762 bp
Apa I (9710)
5.
··
·
·
·
·
·
·
·
·
·
·
·
·
·
·
·
·
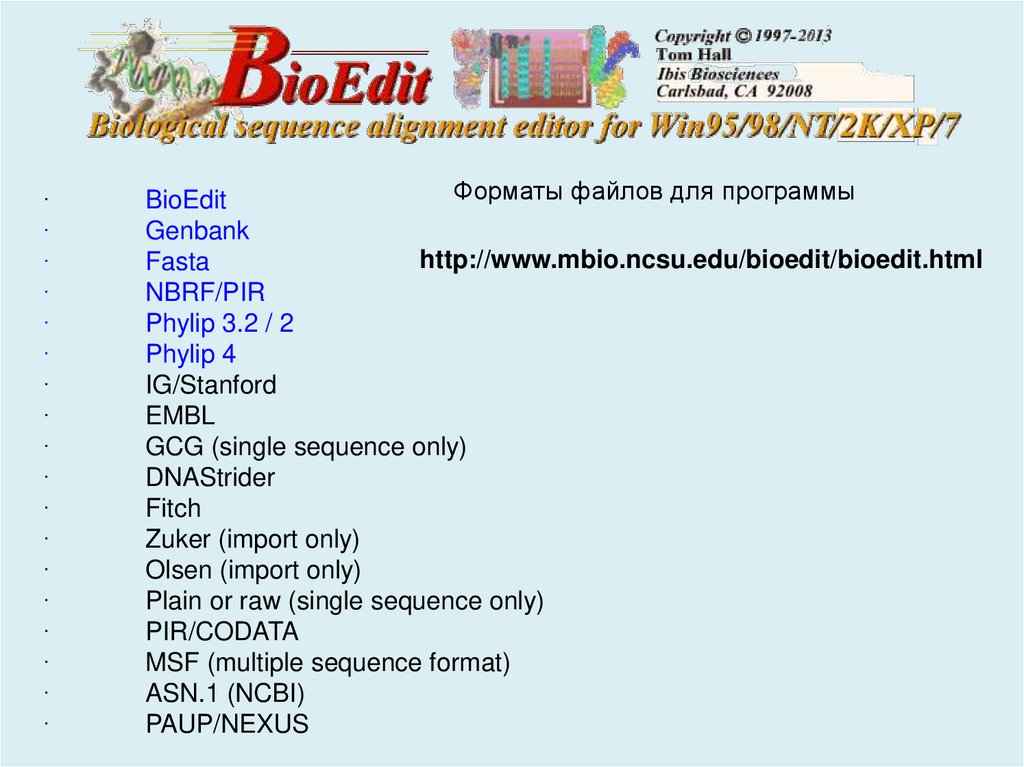
Форматы файлов для программы
BioEdit
Genbank
http://www.mbio.ncsu.edu/bioedit/bioedit.html
Fasta
NBRF/PIR
Phylip 3.2 / 2
Phylip 4
IG/Stanford
EMBL
GCG (single sequence only)
DNAStrider
Fitch
Zuker (import only)
Olsen (import only)
Plain or raw (single sequence only)
PIR/CODATA
MSF (multiple sequence format)
ASN.1 (NCBI)
PAUP/NEXUS
6. FASTA-формат
>Rattus_norvegicus | Rattus norvegicus heat shock 20kDa protein(Loc192245), mRNA
GCAGGATGGAGATCCGGGTGCCTGTGCAGCCTTCTTGGCTGCGCCGTGCTTCAGCT
CCTTTACCGGGTTTTTCCACTCCGGGACGCCTCTTTGACCAGCGTTTCGGCGAAGG
GCTGCTTGAGGCAGAGCTGGCTTC
>Homo_sapiens | Homo sapiens cDNA FLJ32389 fis, clone SKMUS1000138,
highly similar to HEATSHOCK 20 KDA LIKEPROTEIN P20.
ACTGCAACGCGGAGGAGCAGGATGGAGATCCCTGTGCCTGTGCAGCCGTCTTGGC
TGCGCCGCGCCTCGGCCCCGTTGCCCGGACTTTCGGCGCCCGGACGCCTCTTTGAC
CAGCGCTTCGGCGAGGGGCTGCTG
>Mus_musculus | Mus musculus similar to heat shock 20kDa protein
(LOC243912), mRNA.
GGCAGCGTAGGAACAGGATGGAGATCCCCGTGCCTGTGCAGCCTTCTTGGCTGCGC
CGTGCTTCAGCTCCTTTACCAGGTTTCTCTGCTCCGGGACGCCTCTTTGACCA
7. Главное окно программы
8. Меню «Файл»
9. Поиск целевых сайтов рестрикции
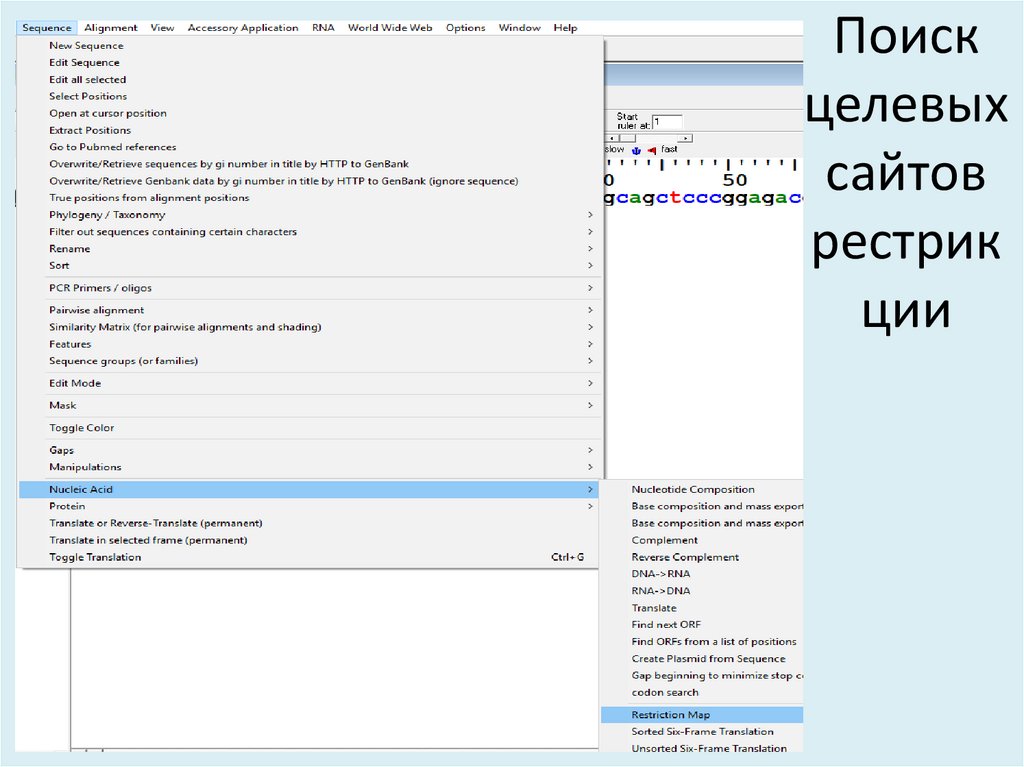
10. Поиск целевых сайтов рестрикции
Поискцелевых
сайтов
рестрик
ции
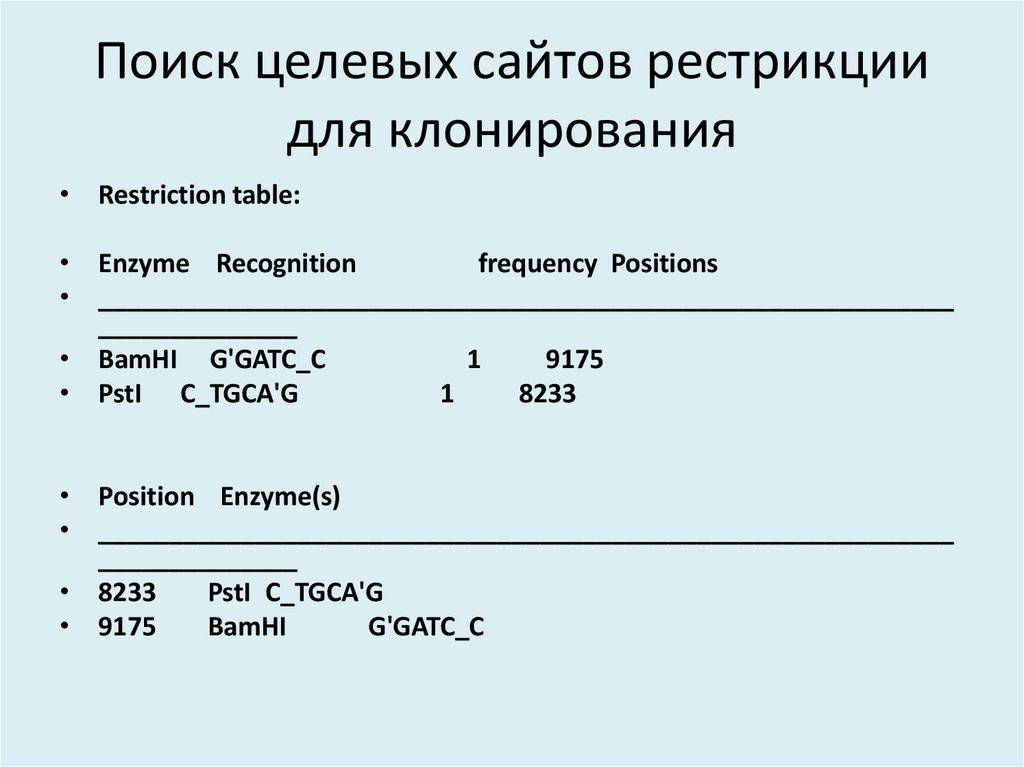
11. Поиск целевых сайтов рестрикции для клонирования
12. Поиск целевых сайтов рестрикции для клонирования
• Restriction table:• Enzyme Recognition
frequency Positions
• ____________________________________________________________
______________
• BamHI G'GATC_C
1
9175
• PstI C_TGCA'G
1
8233
• Position Enzyme(s)
• ____________________________________________________________
______________
• 8233
PstI C_TGCA'G
• 9175
BamHI
G'GATC_C
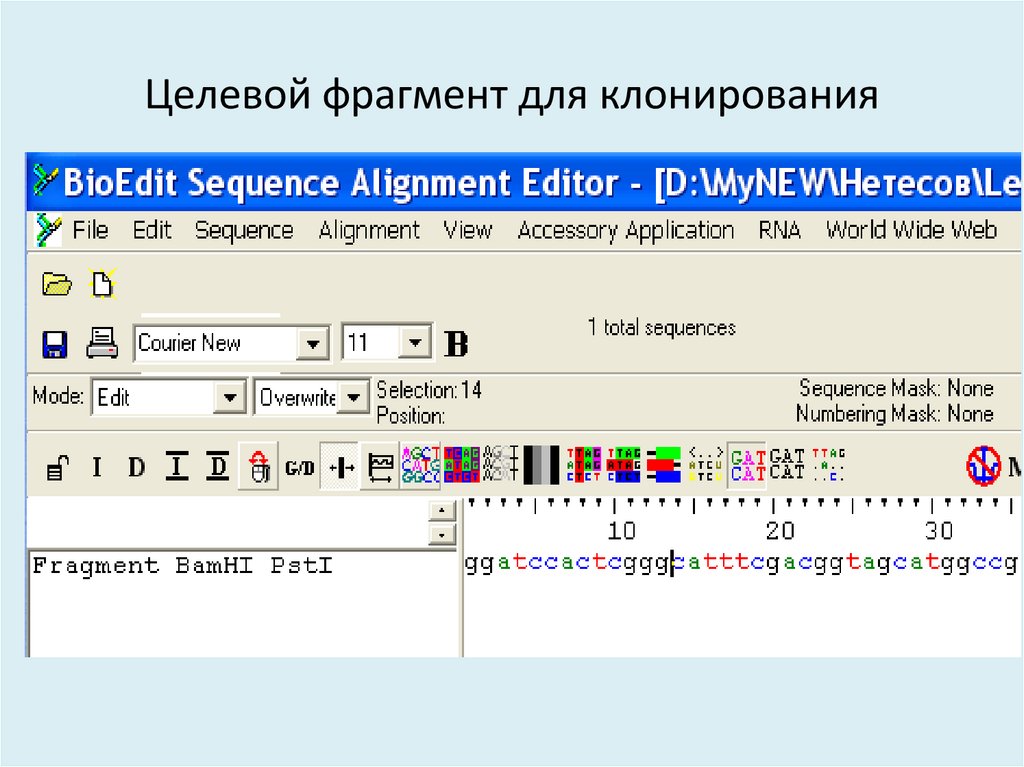
13. Целевой фрагмент для клонирования
14. Вектор для клонирования
15. Рекомбинантная плазмида
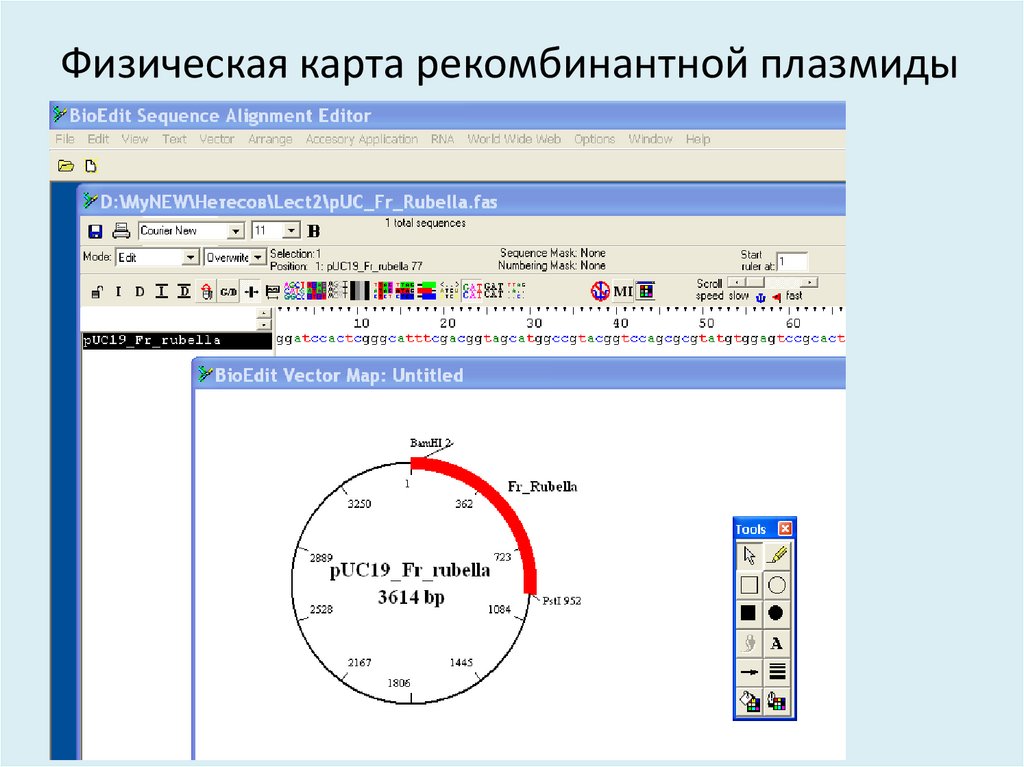
16. Физическая карта рекомбинантной плазмиды
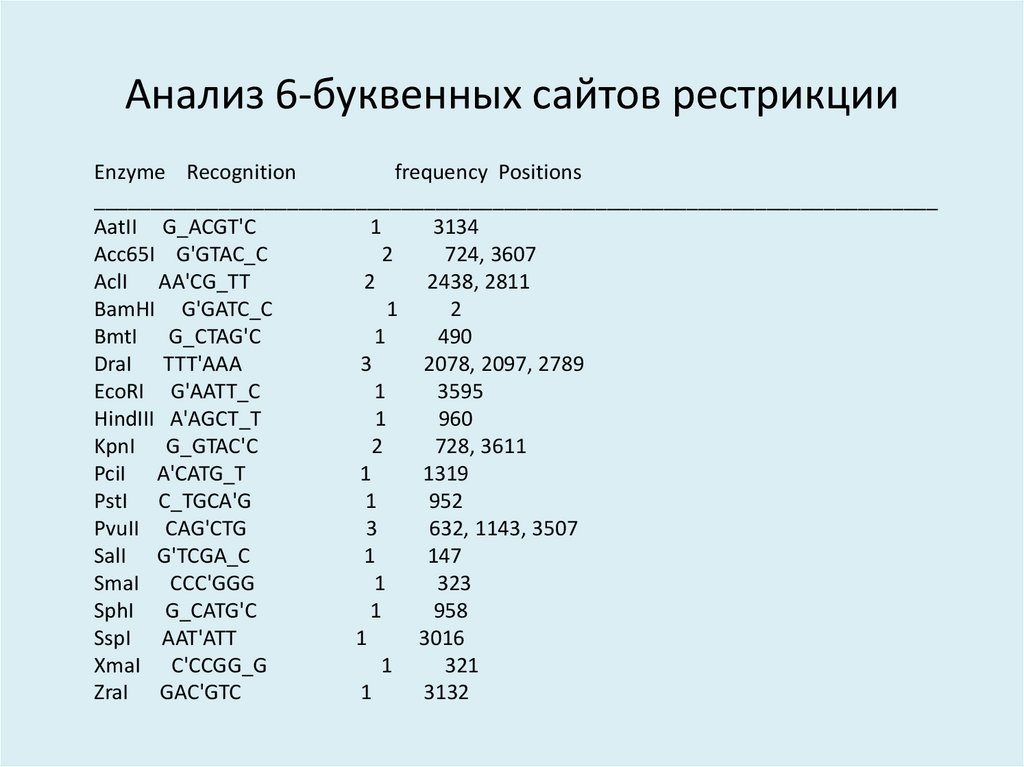
17. Анализ 6-буквенных сайтов рестрикции
Enzyme Recognitionfrequency Positions
__________________________________________________________________________
AatII G_ACGT'C
1
3134
Acc65I G'GTAC_C
2
724, 3607
AclI AA'CG_TT
2
2438, 2811
BamHI G'GATC_C
1
2
BmtI G_CTAG'C
1
490
DraI TTT'AAA
3
2078, 2097, 2789
EcoRI G'AATT_C
1
3595
HindIII A'AGCT_T
1
960
KpnI G_GTAC'C
2
728, 3611
PciI A'CATG_T
1
1319
PstI C_TGCA'G
1
952
PvuII CAG'CTG
3
632, 1143, 3507
SalI G'TCGA_C
1
147
SmaI CCC'GGG
1
323
SphI G_CATG'C
1
958
SspI AAT'ATT
1
3016
XmaI C'CCGG_G
1
321
ZraI GAC'GTC
1
3132
18.
• Четыре режима ручного выравнивания.• Встраивание и редактирование цвета с помощью отдельных таблиц и полный
контроль над цветами фона.
• Интерфейс рисования плазмид для автоматического создания кольцевой
графики из последовательностей ДНК. Легко отмечать позиции, добавлять
объекты ввиде стрелок и изогнутых прямоугольников и отмечать сайты
ферментов рестрикции. Также возможно отмечать полилинкеры и рисовать
стрелки и фигуры с помощью инструментов рисования.
• Динамическое затенение выравнивания на основе различной информации.
• Редактирование таблицы цветов
• Отображение и печать ABI хроматограмм.
• Группировать последовательности в группы или семейства.
• Аннотировать последовательности с графическими функциями с
динамическим представлением в окнах выравнивания, включая
всплывающие подсказки.
• Блокировать случайные изменения последовательностей.
• Указывать символы которые считаются действительными для расчетов
аминокислотных и нуклеотидных последовательностей.
19.
• Сортировать последовательности по имени, LOCUS, DEFINITION,ACCESSION, PID/NID, REFERENCES, COMMENTS или по частоте остатков
в выбранной колонке.
• Объединить выравнивания через референсную последовательность.
• Добавить одно выравнивание в конец другого.
• Простой просмотрщик филогенетических деревьев (для деревьев в phylip
формате), который позволяет редактировать узлы и печатать.
• Читать и записывает файлы в форматах Genbank, Fasta, Phylip 3.2, Phylip 4
GCG, Clustal и NBRF / PIR.
• Использовать ReadSeq для автоматического импорта и экспорта 11
дополнительных форматов, включая MSF, ASN.1, IG / Stanford и EMBL.
• Импортировать совместимые форматы непосредственно из буфера обмена
без сохранения в файл.
• Легкая настройка ярлыков меню окна редактора
• Сравнительный анализ РНК, включая ковариацию и потенциальные
спаривания.
• Просмотр и редактирование выравниваний до 20 000 последовательностей.
• Формат BioEdit Project для быстрого открытия и сохранения больших
выравниваний.
20.
• Поиск ОРТ с пользовательскими настройками• Трансляция последовательностей нуклеиновых кислот с информацией об
использовании кодонов, выбор одно- или трехбуквенных аминокислотных
кодов. Трансляция только выбранной области нуклеиновой кислоты и
выбор start/stop кодонов.
• Разделить окно для одновременного и синхронизированного редактирования
двух разных мест в одном файле - разбиение окна по вертикали или по
горизонтали
• Анализ аминокислотного и нуклеотидного состава и плоты.
• Выровнивание последовательности нуклеиновой кислоты, кодирующей
белок, через аминокислотную последовательность.
• Множественное выравнивание ClustalW с автоматическим обновлением
информациии в формате GenBank.
• Графики гидрофобности / гидрофильности белков
• Графические матрицы гидрофобных доменов белка
• Выбор шрифтов
• Рестрикционное картирование с любой трансляцией, множеством вариантов
выбора ферментов и возможностью анализа кольцевой ДНК
21.
• Обзор ферментов рестрикции• Возможность манипулировать последовательностями длиной не более 4,6
МБ.
• Six-frame translations capable of raw translation of entire genomes Трансляция в
шести рамках
• Сохранять файлы формата GenBank с полями LOCUS, DEFINITION,
ACCESSION, PID, NID, DBSOURCE, KEYWORDS, SOURCE, REFERENCE,
COMMENT, and FEATURES. Изменять поля или добавьте свою собственную
информацию.
• Вывод графической информации с выделением степени идентичности и
подобия и несколькими вариантами форматирования.
• Экспорт текста в формате выравнивания
• Он-лайн справочная система.
• Плот энтропии.
• Интерфейс нескольких документов.
• Основные манипуляции с последовательностями (обратная /
комплементарная, трансляция, ДНК-> РНК-> ДНК)
• Легкий экспорт текста и настраиваемая печать.
• Полный пакет NCBI для локальных программ BLAST.
22.
• Настройка и запуск вспомогательных приложений через графическийинтерфейс BioEdit. BioEdit поставляется с:
– TreeView
– CAP-ассемблер
– FastDNml
– Программы Phylip
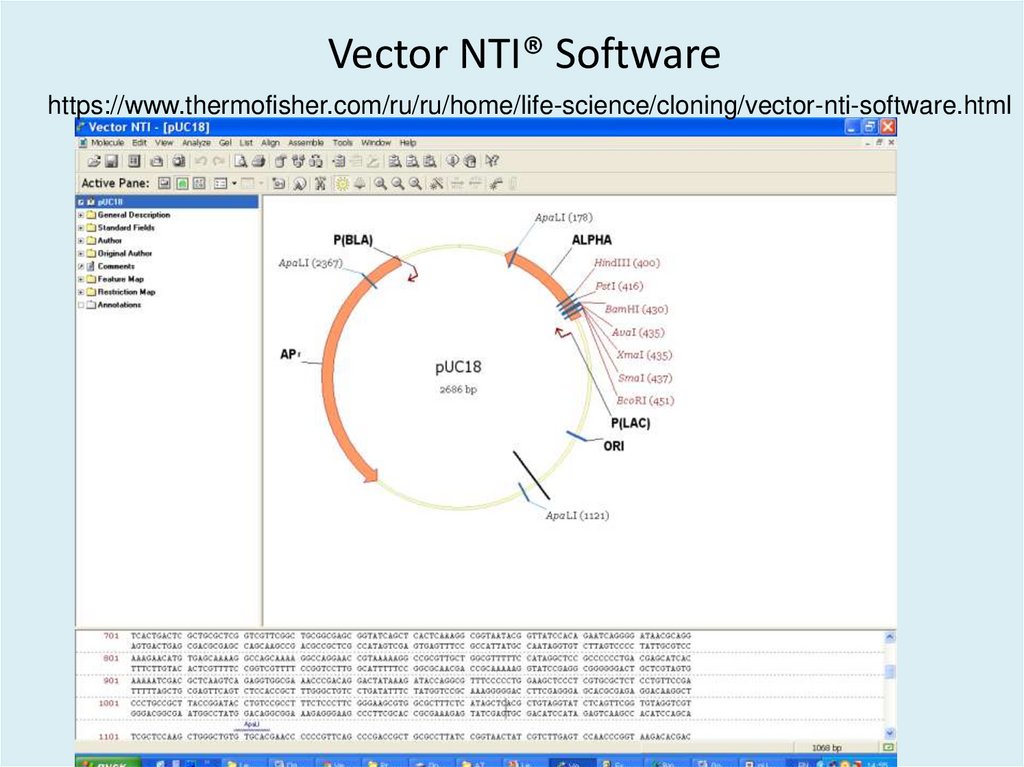
23. Vector NTI® Software
https://www.thermofisher.com/ru/ru/home/life-science/cloning/vector-nti-software.html24.
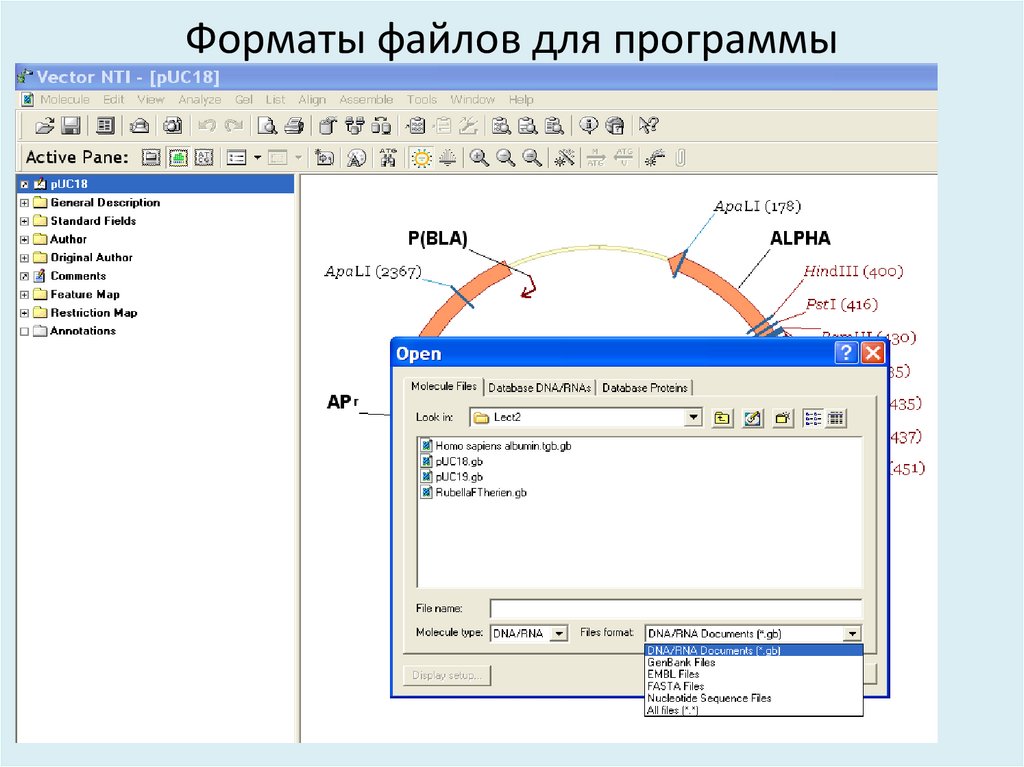
25. Форматы файлов для программы
26. GenBank-формат
LOCUS
DEFINITION
KEYWORDS
SOURCE
ORGANISM
COMMENT
GenBank-формат
pUC18
2686 bp
DNA
circular
ATCC 37253: very usable general purpose vector.
ATCC.
13-APR-2013
This file is created by Vector NTI
http://www.informaxinc.com/
FEATURES
Location/Qualifiers
promoter
complement(2521..2521)
/vntifkey="30"
/label=P(BLA)
/note=" bla gene promoter "
CDS
complement(1629..2486)
/vntifkey="4"
/label=AP(R)
/note=" bla gene- Ap(r) determinant "
promoter
complement(507..507)
/vntifkey="30"
/label=P(LAC)
/note=" lac promoter "
rep_origin
867..867
/vntifkey="33"
/label=ORI
/note=" RNaseH cleavage point "
CDS
complement(149..469)
/vntifkey="4"
/label=ALPHA
/note=" lacZ gene fragment coding for LacZ alpha peptide "
BASE COUNT
666 a
677 c
684 g
659 t
ORIGIN
1 tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca
61 cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg
121 ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc
181 accatatgcg gtgtgaaata ccgcacagat gcgtaaggag aaaataccgc atcaggcgcc
241 attcgccatt caggctgcgc aactgttggg aagggcgatc ggtgcgggcc tcttcgctat
301 tacgccagct ggcgaaaggg ggatgtgctg caaggcgatt aagttgggta acgccagggt
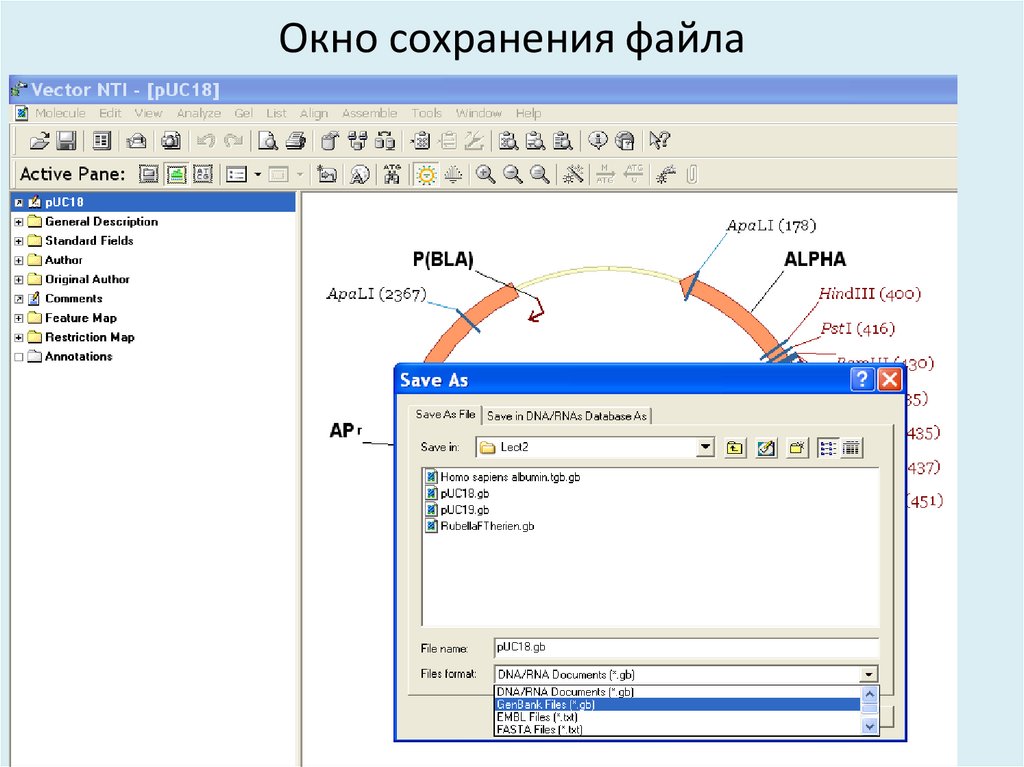
27. Окно сохранения файла
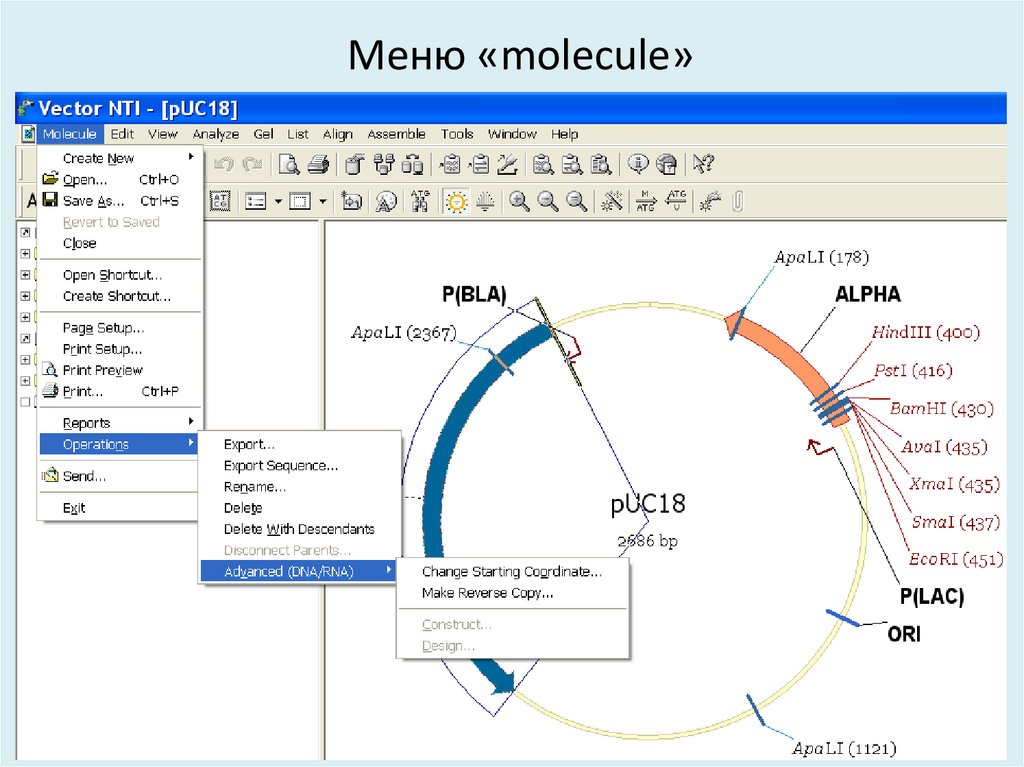
28. Меню «molecule»
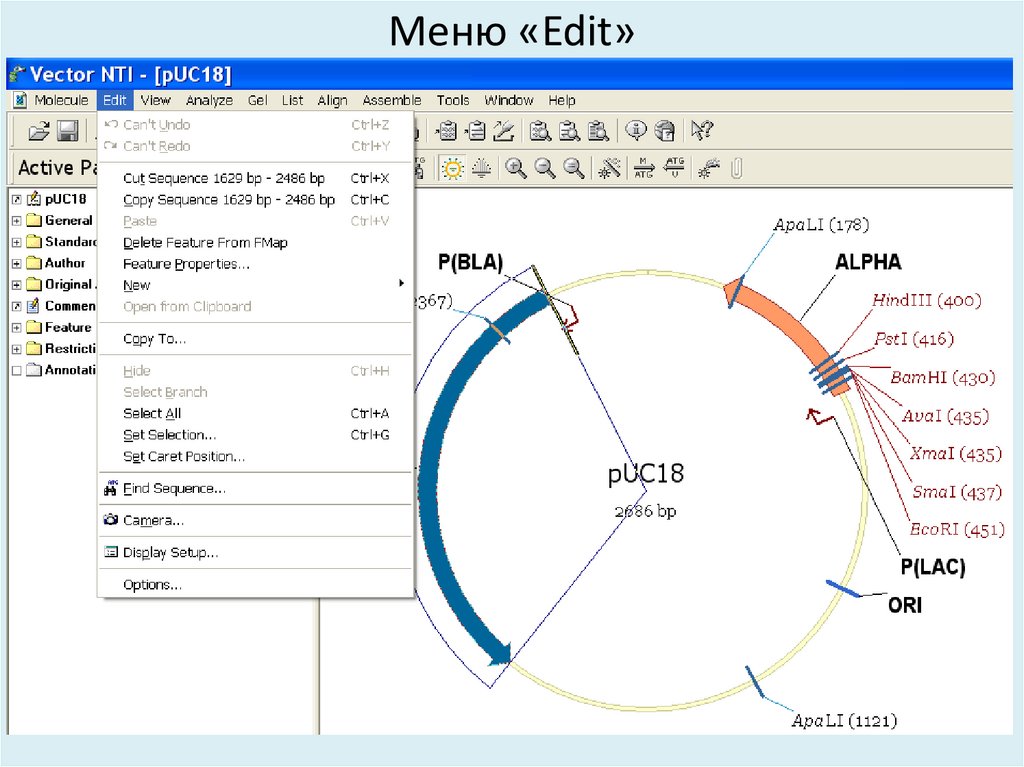
29. Меню «Edit»
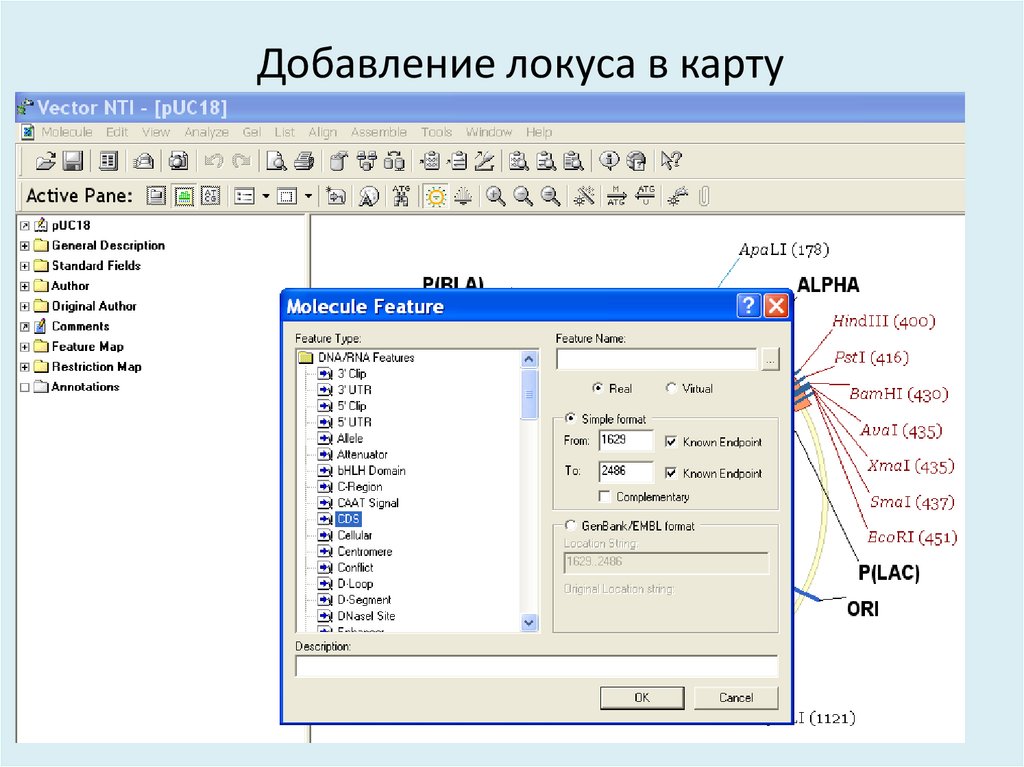
30. Добавление локуса в карту
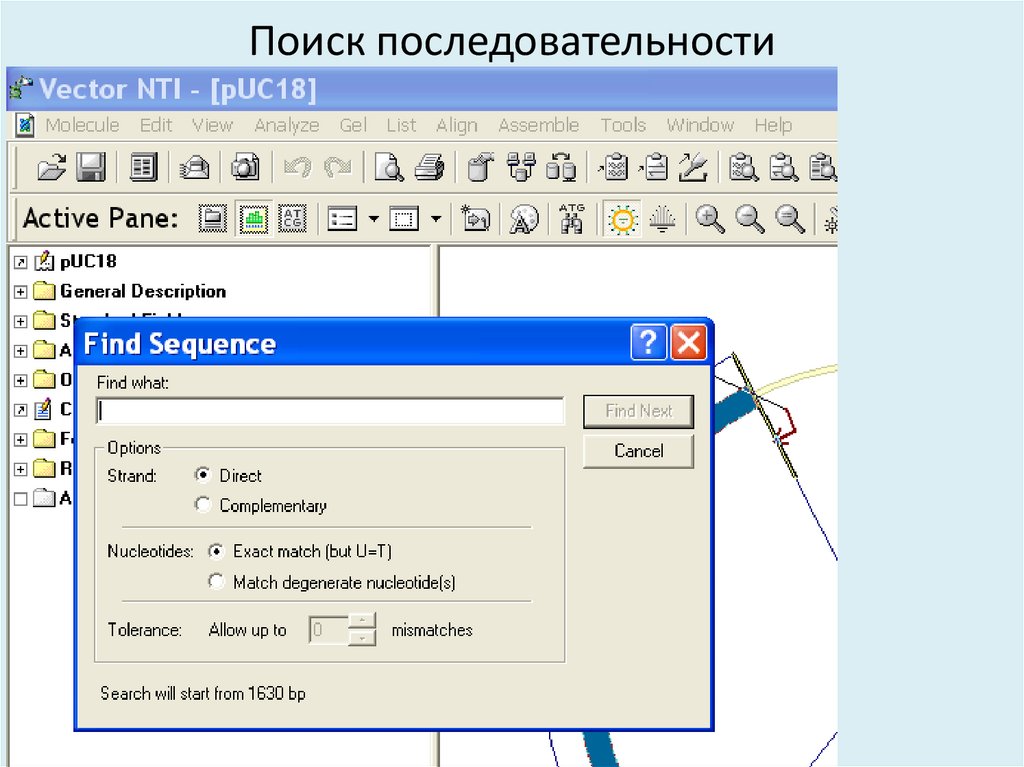
31. Поиск последовательности
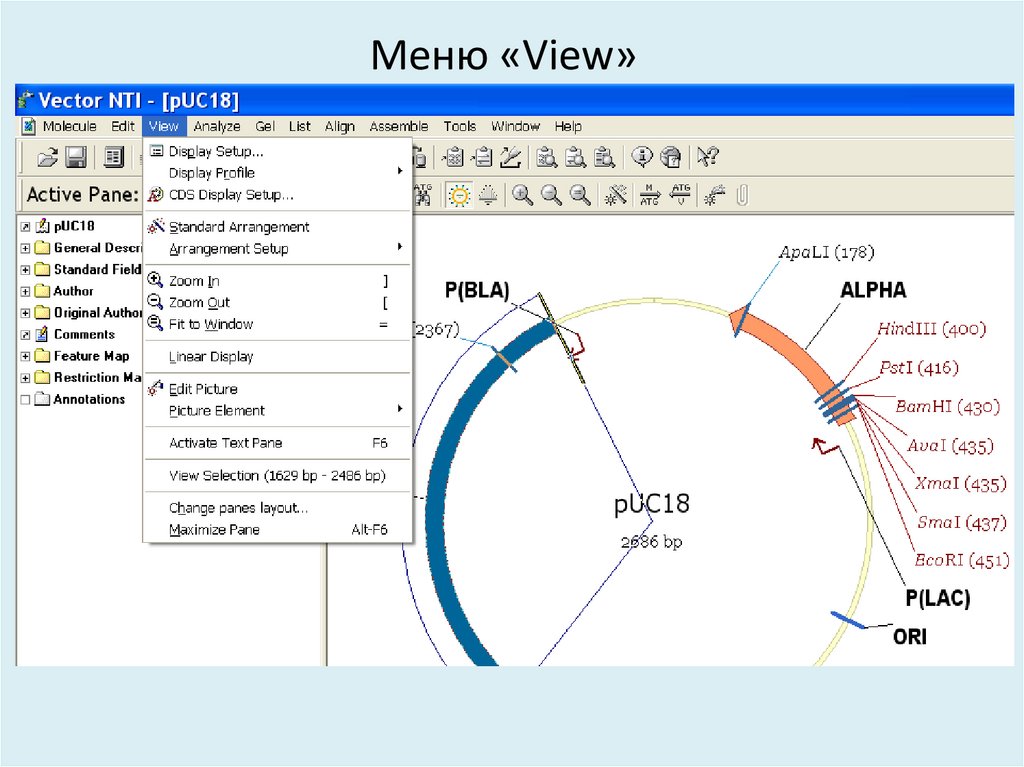
32. Меню «View»
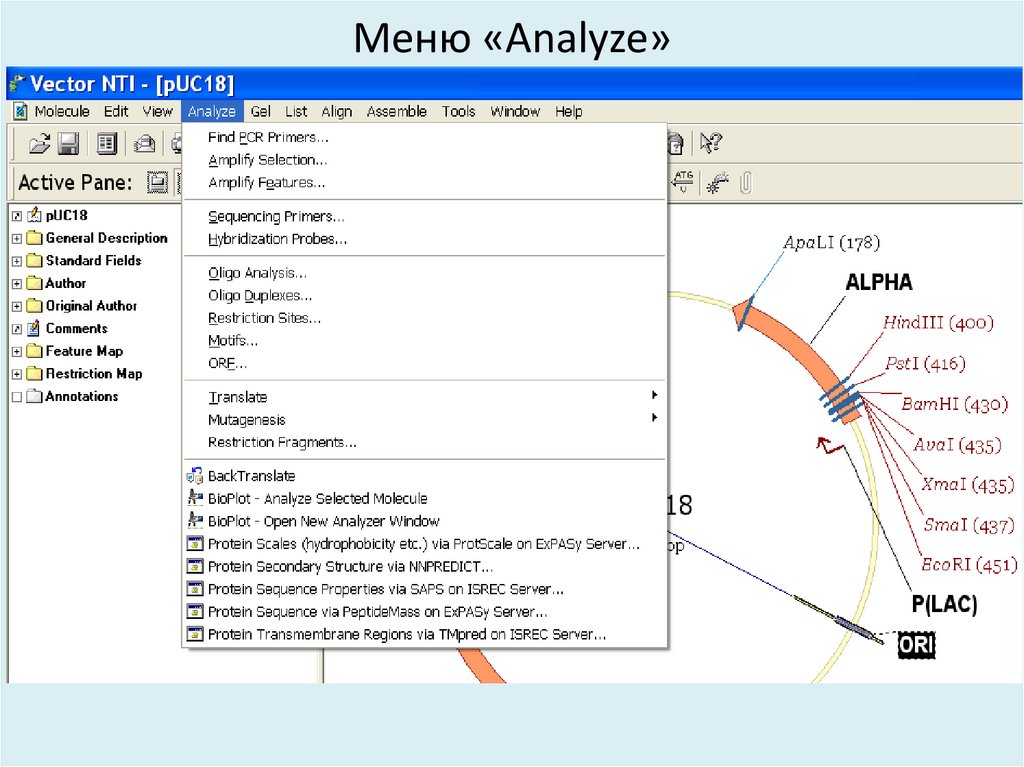
33. Меню «Analyze»
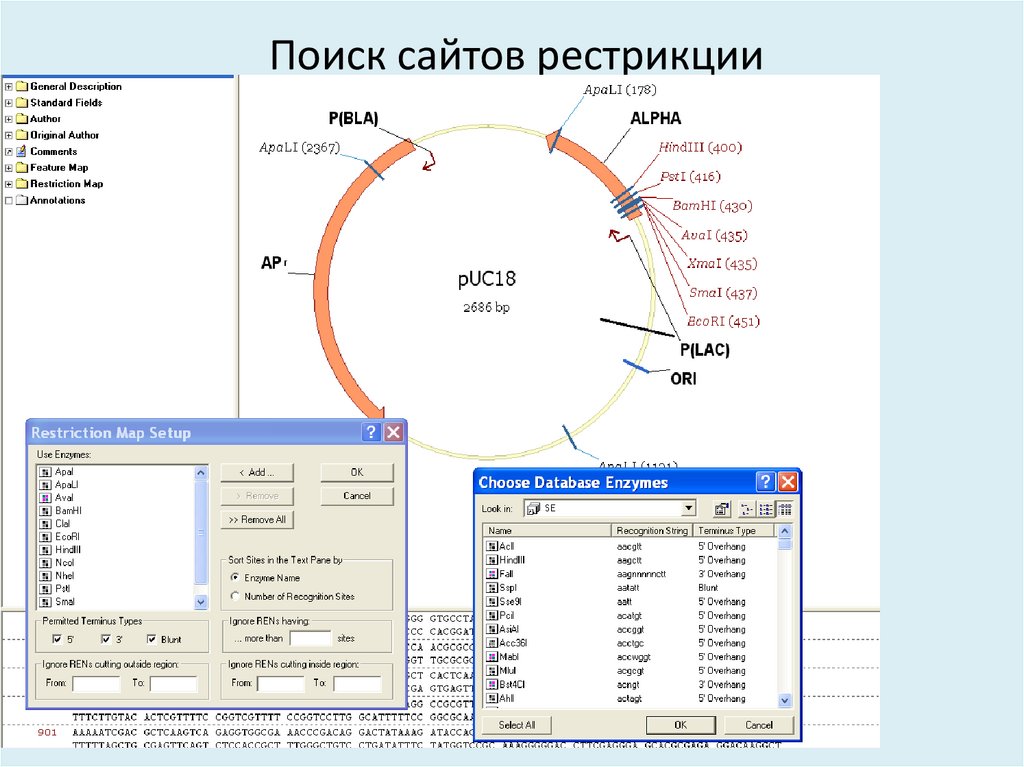
34. Поиск сайтов рестрикции
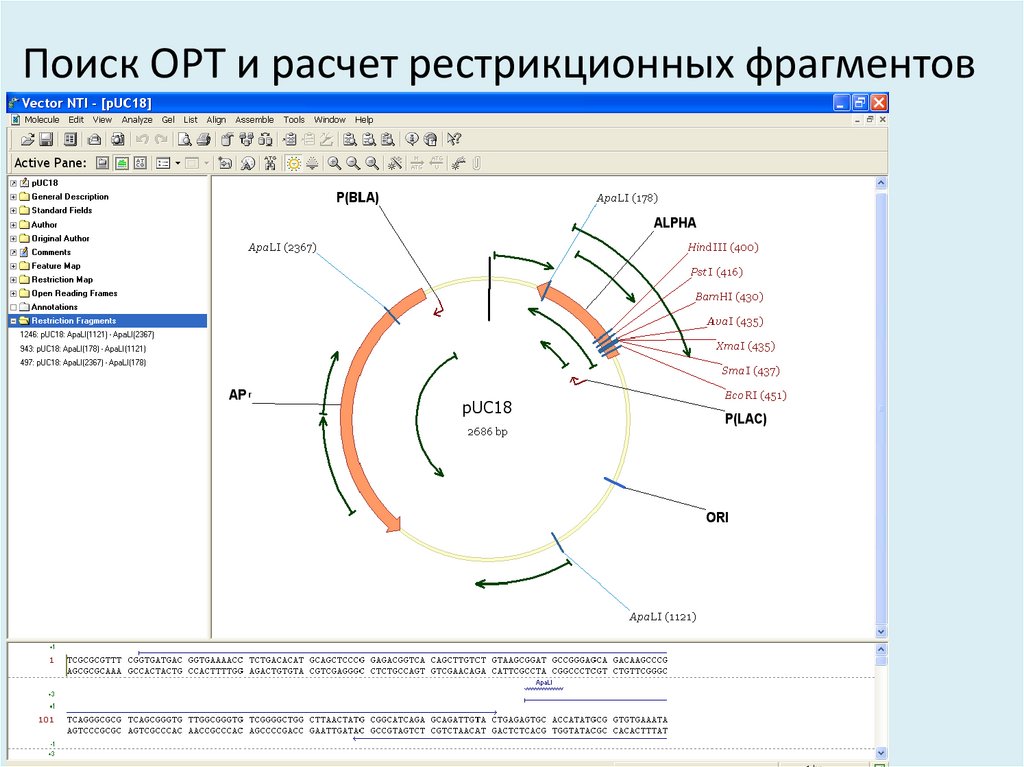
35. Поиск ОРТ и расчет рестрикционных фрагментов
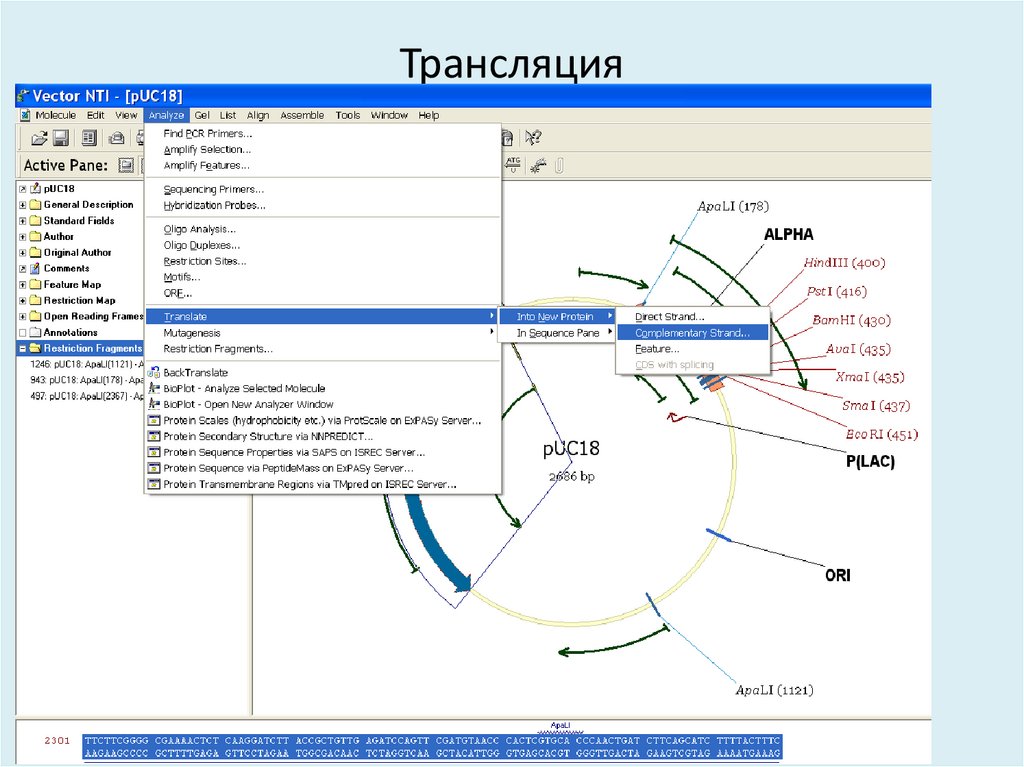
36. Трансляция
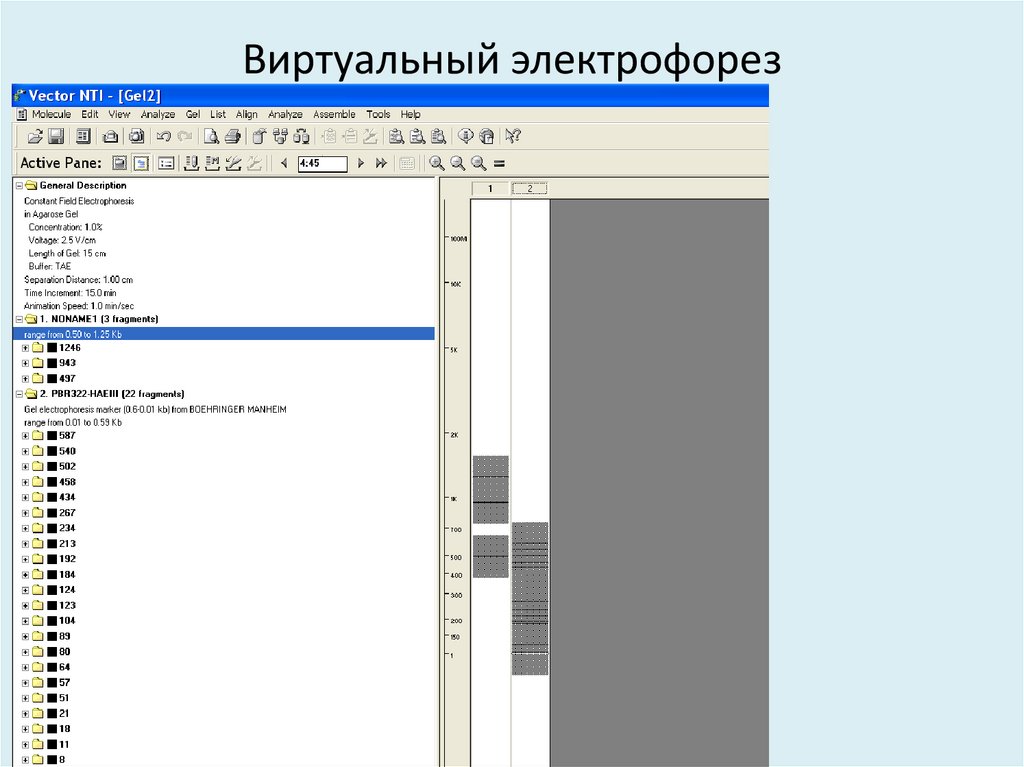
37. Виртуальный электрофорез
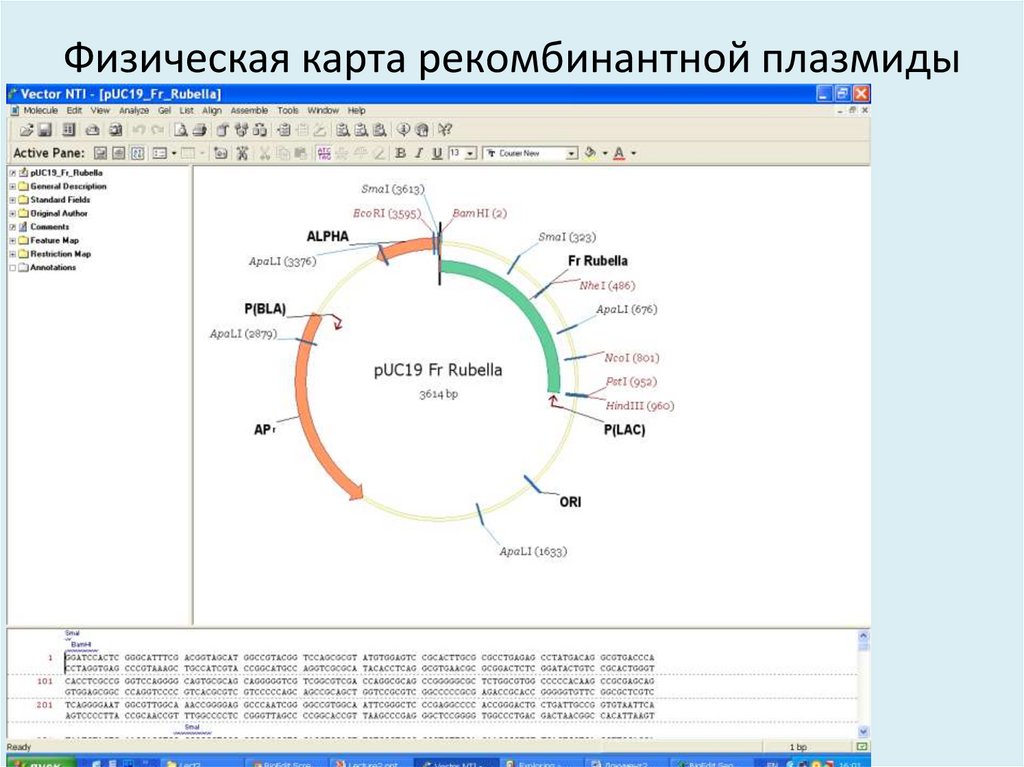
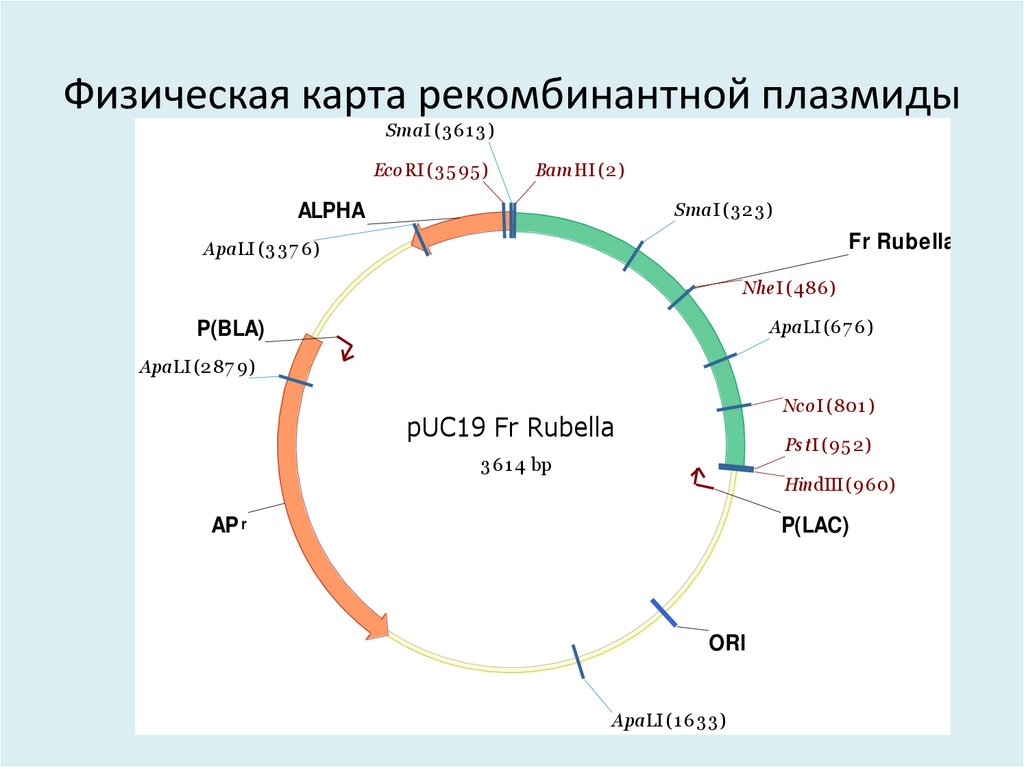
38. Физическая карта рекомбинантной плазмиды
39. Физическая карта рекомбинантной плазмиды
SmaI (3 6 1 3 )Eco RI (3 5 9 5 )
BamHI (2 )
SmaI (3 2 3 )
ALPHA
Fr Rubella
ApaLI (3 3 7 6)
NheI (486 )
ApaLI (6 7 6 )
P(BLA)
ApaLI (2 87 9)
NcoI (801 )
pUC19 Fr Rubella
PstI (95 2 )
3 6 1 4 bp
HindIII (9 6 0)
AP r
P(LAC)
ORI
ApaLI (1 6 3 3 )
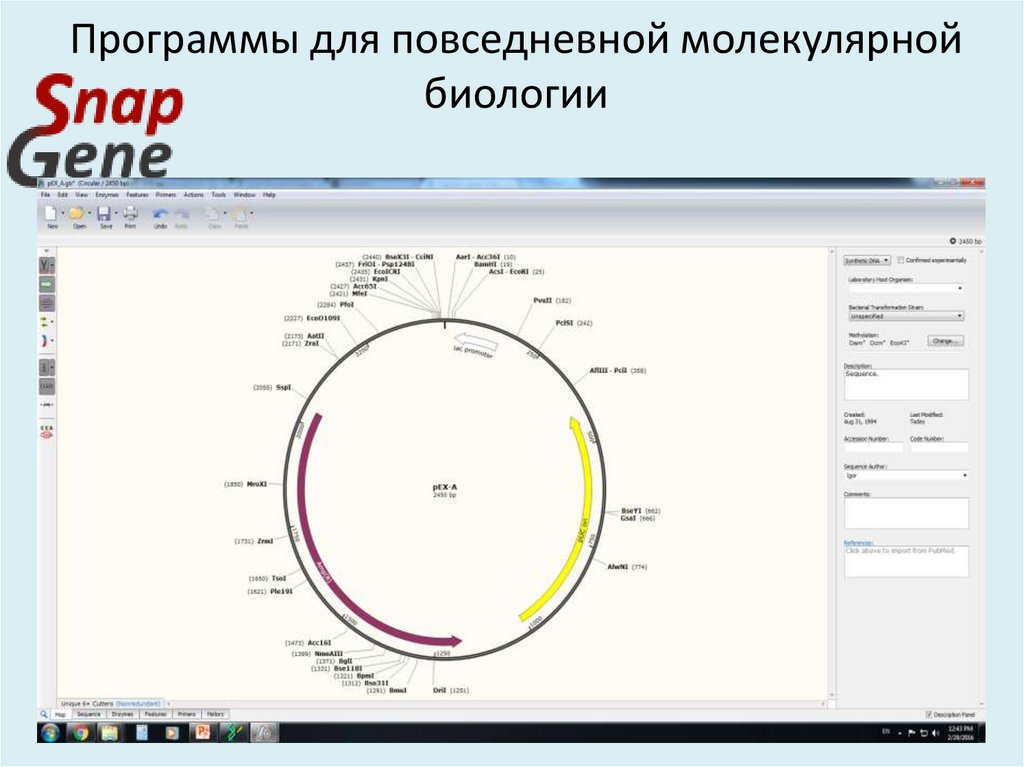
40. Программы для повседневной молекулярной биологии
41. Программы для повседневной молекулярной биологии
SimVector
Geneious
DNASTAR Lasergene
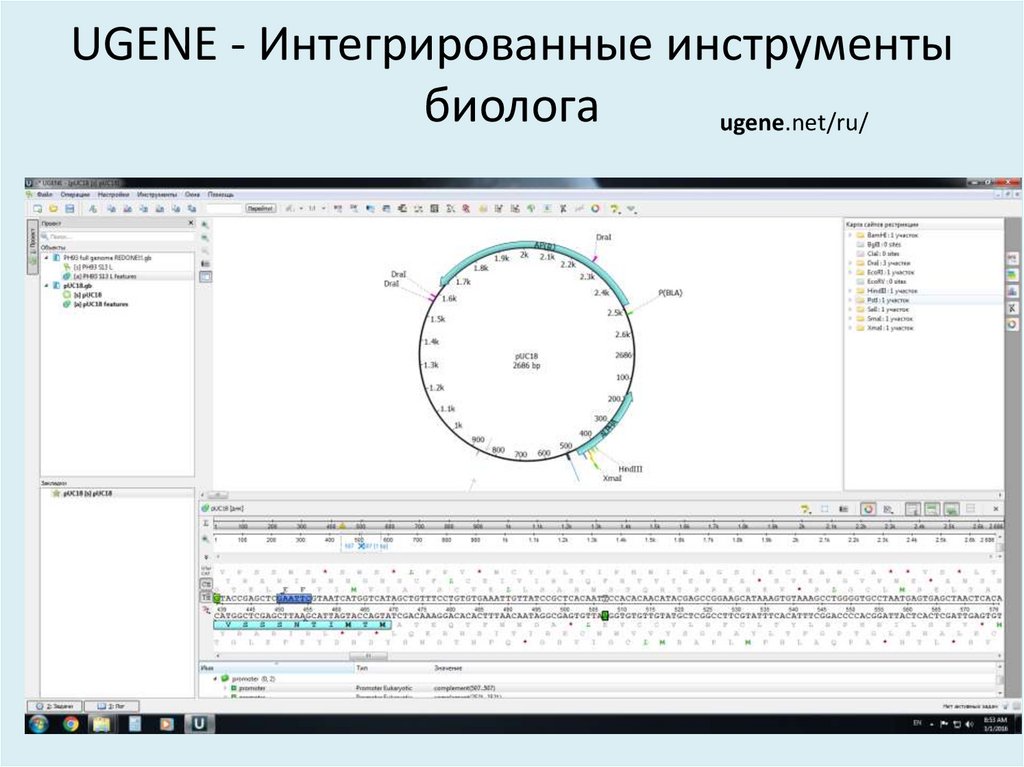
Unipro UGENE (free)
42. UGENE - Интегрированные инструменты биолога
ugene.net/ru/43.
Полезные Интернет-сайты:1. http://www.ncbi.nlm.nih.gov/
2. http://www.ebi.ac.uk/
3. http://web.expasy.org/
4. http://evolution.genetics.washington.edu/phylip/software.html#methods
5. http://blast.ncbi.nlm.nih.gov/Blast.cgi
6. http://mafft.cbrc.jp/alignment/server/index.html
7. http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form
8. http://molbiol.ru/
44. Nexus файл
#NEXUSBegin data;
Dimensions ntax=4 nchar=15;
Format datatype=dna missing=? gap=-;
Matrix
Species1 atgctagctagctcg
Species2 atgcta??tag-tag
Species3 atgttagctag-tgg
Species4 atgttagctag-tag
;
End;