Похожие презентации:
Белки праймосомного комплекса бактерий E
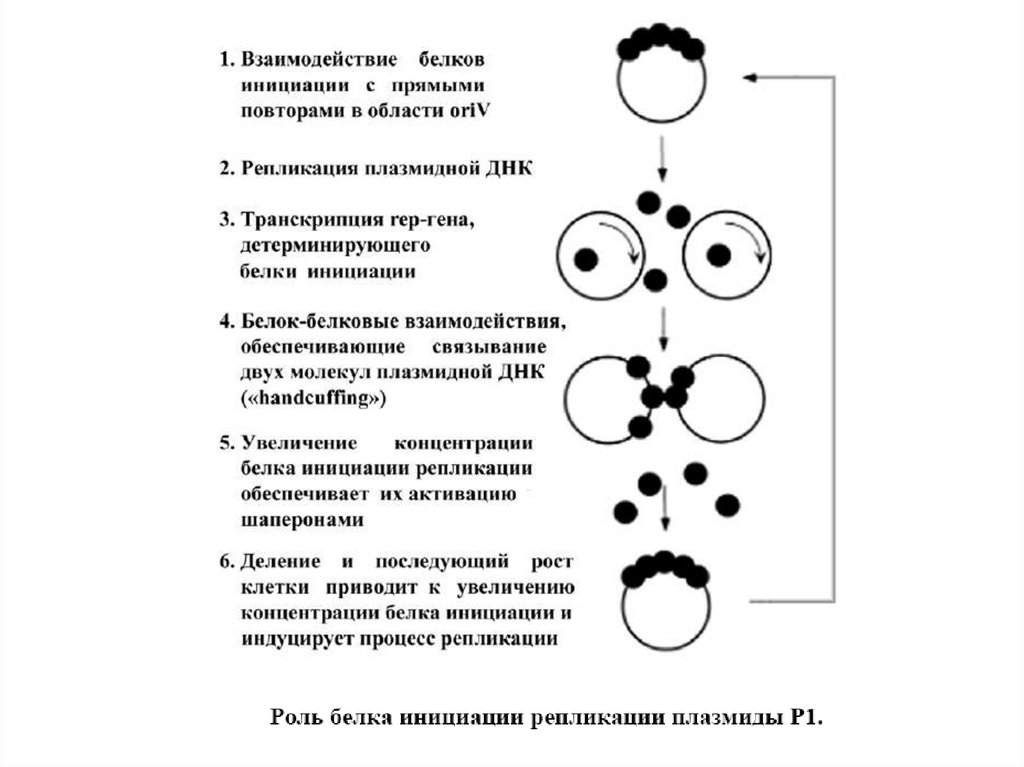
1.
2.
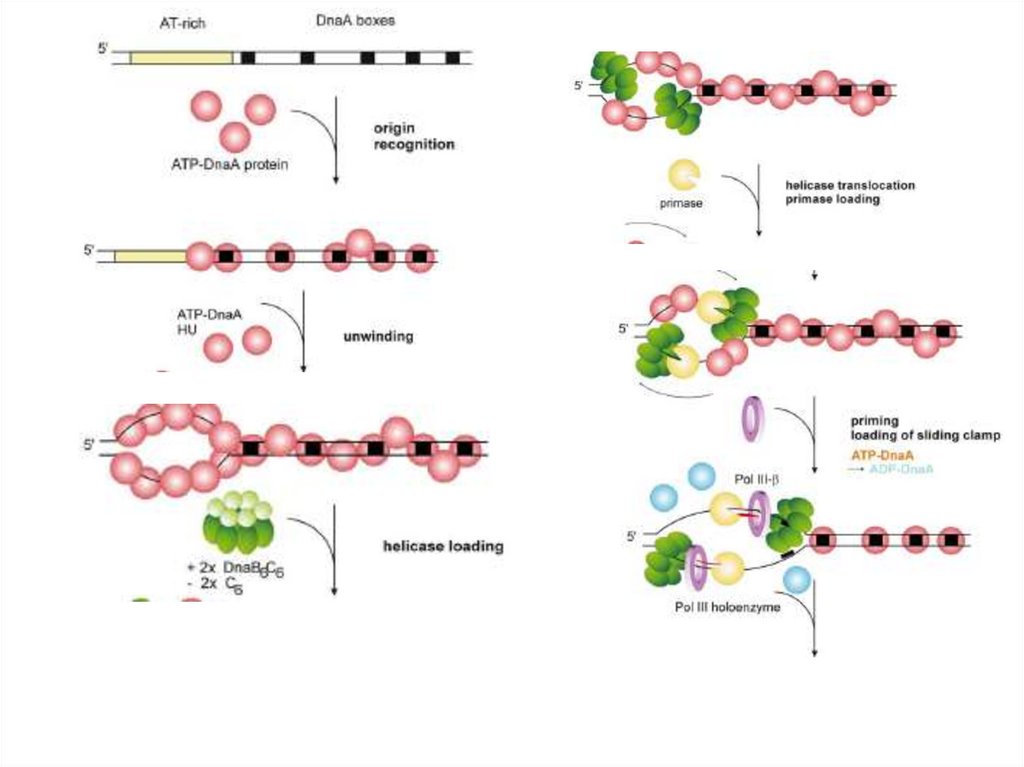
Белки праймосомного комплекса бактерий E. coli и B. subtilis.Репликация ДНК инициируется белком DnaA (либо белком PriA), который при
участии белков PriB, PriC, DnaT (в случае E. coli), либо белков DnaD, DnaB,
DnaI (в случае B. subtilis) обеспечивает связывание DnaB-хеликазы (в случае
E. coli) или DnaC-хеликазы (в случае B. subtilis), а также DnaG-праймазы в
области репликативной вилки. Белки праймосомного комплекса (DnaD, DnaB,
DnaI) бактерий B. subtilis не гомологичны белкам праймосомного комплекса
бактерий E. coli (PriB, PriC, DnaT).
3.
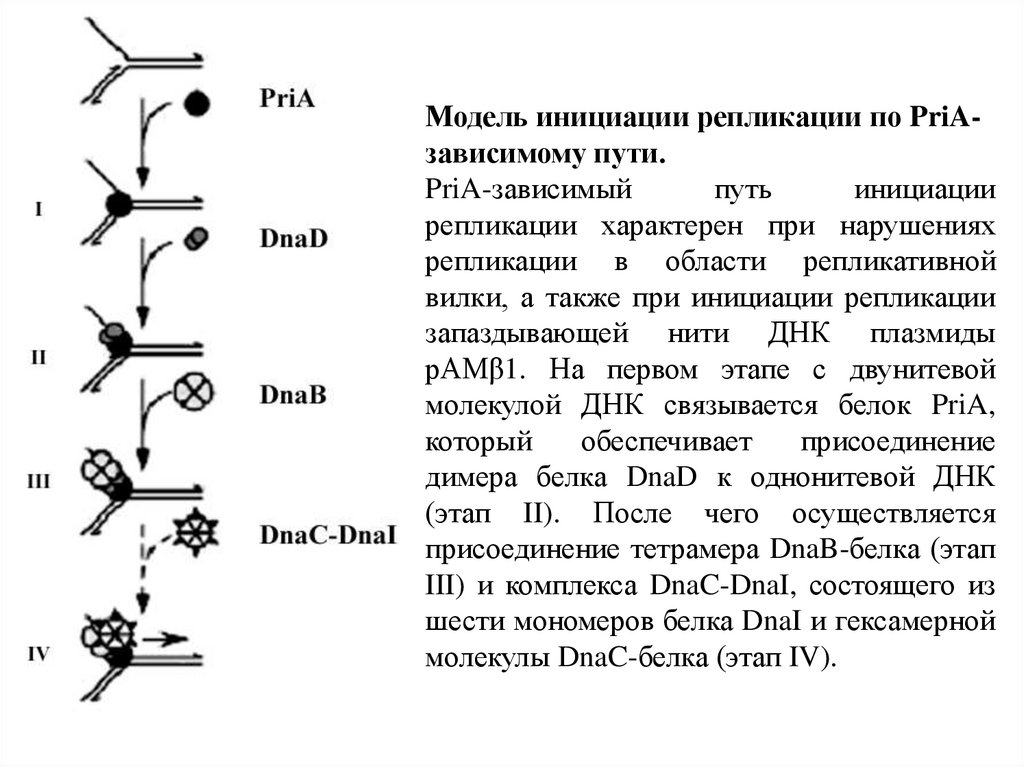
Модель инициации репликации по PriAзависимому пути.PriA-зависимый
путь
инициации
репликации характерен при нарушениях
репликации в области репликативной
вилки, а также при инициации репликации
запаздывающей нити ДНК плазмиды
pAMβ1. На первом этапе с двунитевой
молекулой ДНК связывается белок PriA,
который
обеспечивает
присоединение
димера белка DnaD к однонитевой ДНК
(этап II). После чего осуществляется
присоединение тетрамера DnaB-белка (этап
III) и комплекса DnaC-DnaI, состоящего из
шести мономеров белка DnaI и гексамерной
молекулы DnaC-белка (этап IV).
4.
E. coliB. subtilis
Гомология
Функция
α
PolC
DnaE
30%
40%
Полимеризация
нити ДНК
εθ
PolC,
KapD ?
20%
10%
Экзонуклеазная
активность в
направлении 3'-5'
τ
τ
40%
Объединение
субъединиц
комплекса
β
β
25%
Фиксация нитей
родительской
ДНК
γδδ΄ψχ
τδδ΄
10-40%
Содействие
функции
β-субъединицы
5.
6.
7.
8.
9.
10.
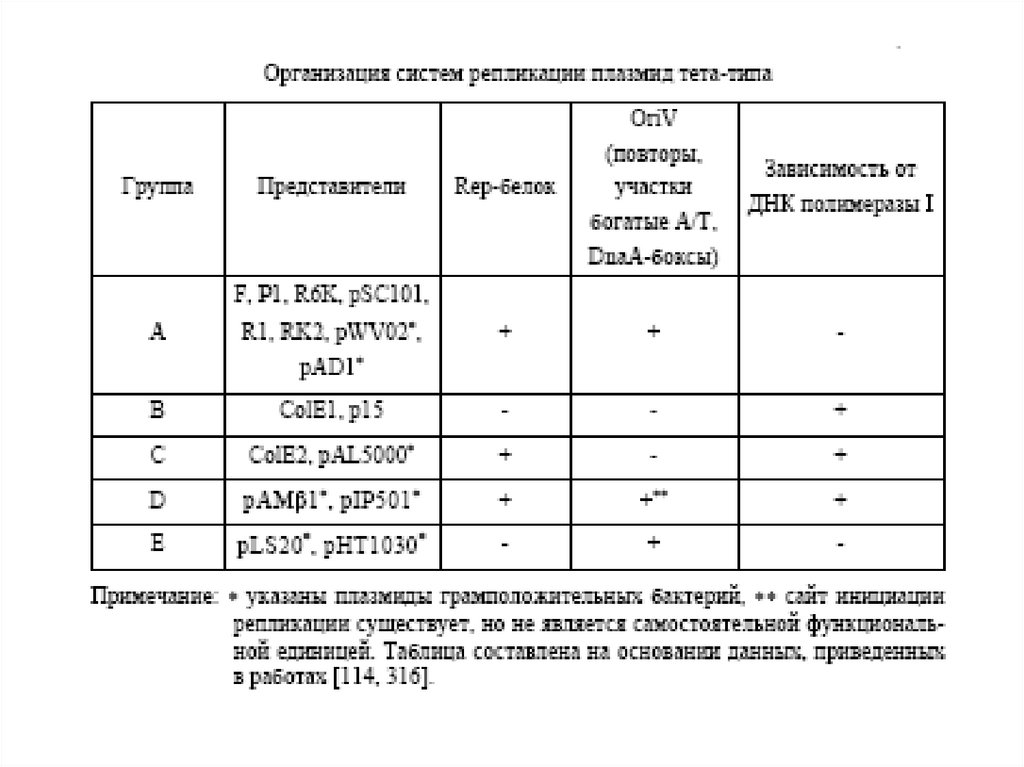
Организация rep-области плазмиды рLS32.Базовый репликон размером 1,5 kb содержит rep-ген (repN), включающий
сайт инициации репликации, содержащий семь прямых (обозначены цифрами
1, 2, 3, 4, 5, 6, 7) и три инвертированных повтора (повтор 2 и 5 представлены в
прямой и обратной ориентации, повтор 8 является инвертированным).
11.
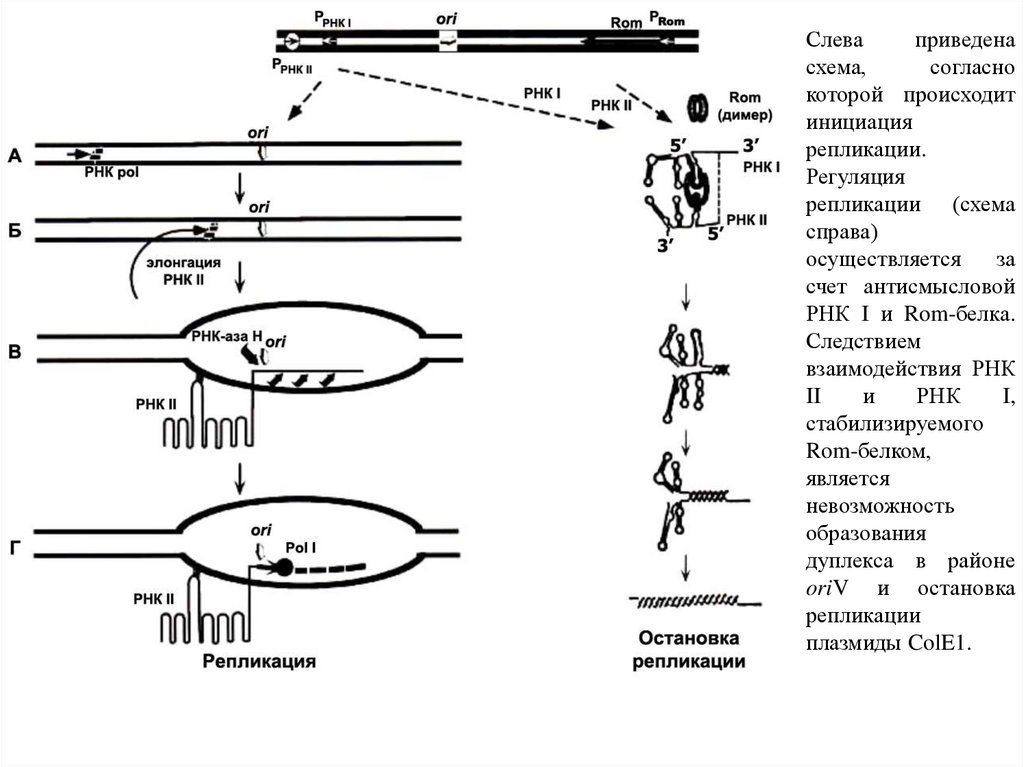
Слеваприведена
схема,
согласно
которой происходит
инициация
репликации.
Регуляция
репликации (схема
справа)
осуществляется
за
счет антисмысловой
РНК I и Rom-белка.
Следствием
взаимодействия РНК
II
и
РНК
I,
стабилизируемого
Rom-белком,
является
невозможность
образования
дуплекса в районе
oriV и остановка
репликации
плазмиды ColE1.
12.
Организация rep-области плазмид группы С.13.
Организация rep-областей плазмид семейства pAMβ114.
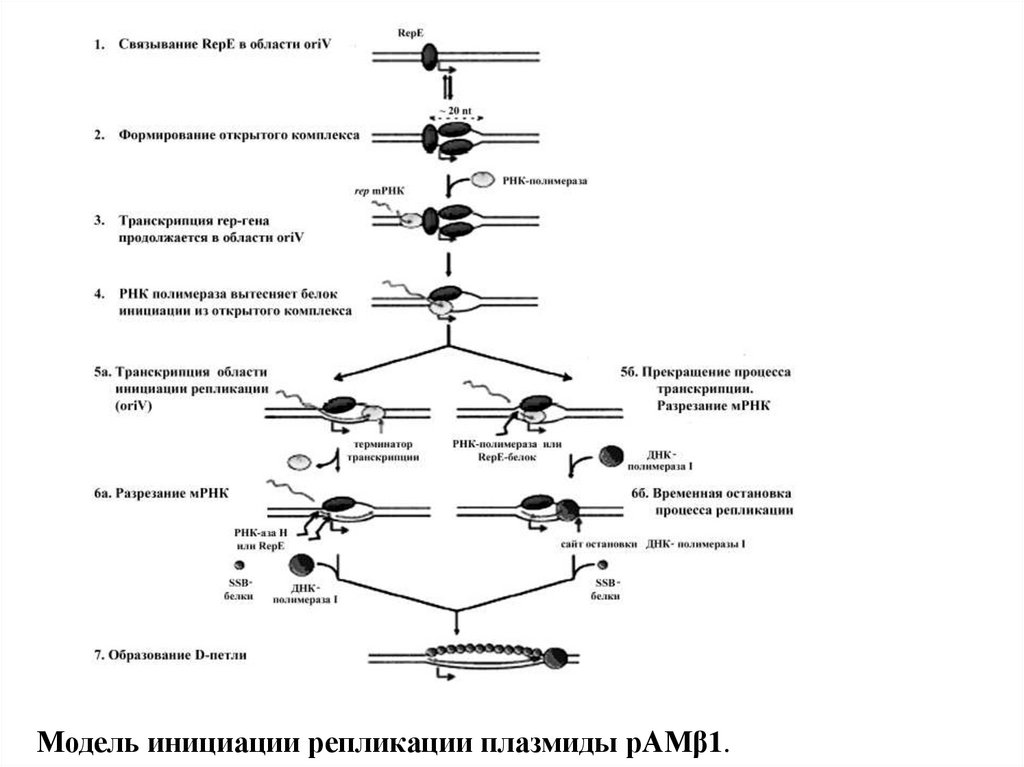
Модель инициации репликации плазмиды pAMβ1.15.
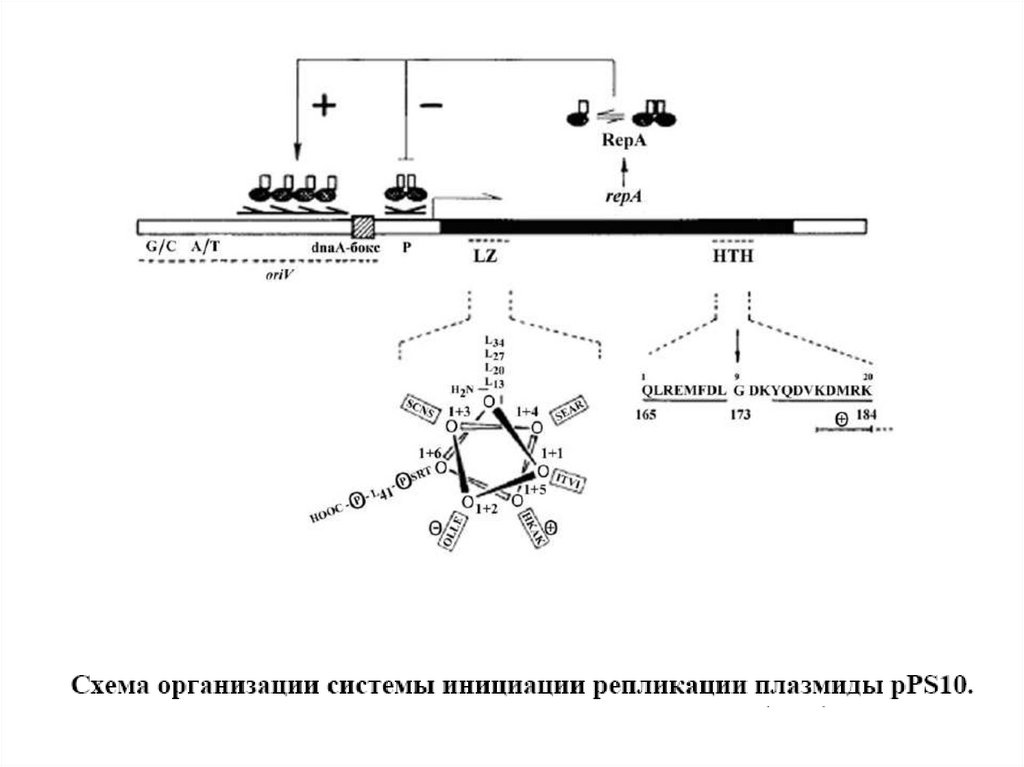
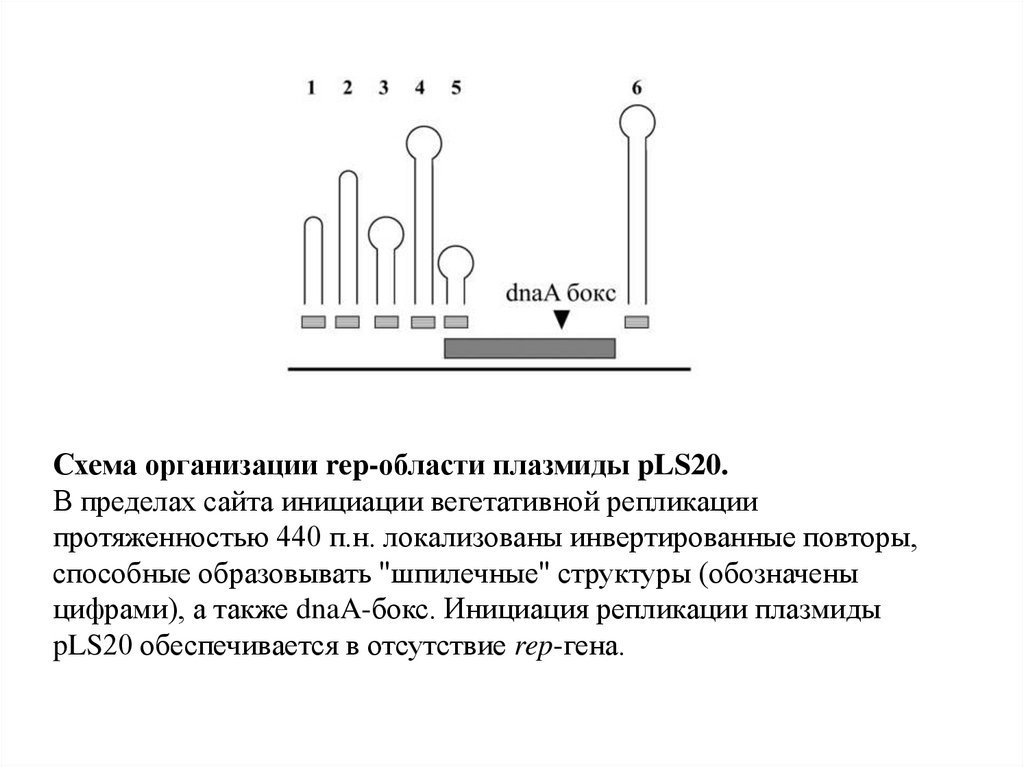
Схема организации rep-области плазмиды pLS20.В пределах сайта инициации вегетативной репликации
протяженностью 440 п.н. локализованы инвертированные повторы,
способные образовывать "шпилечные" структуры (обозначены
цифрами), а также dnaA-бокс. Инициация репликации плазмиды
pLS20 обеспечивается в отсутствие rep-гена.
16.
17.
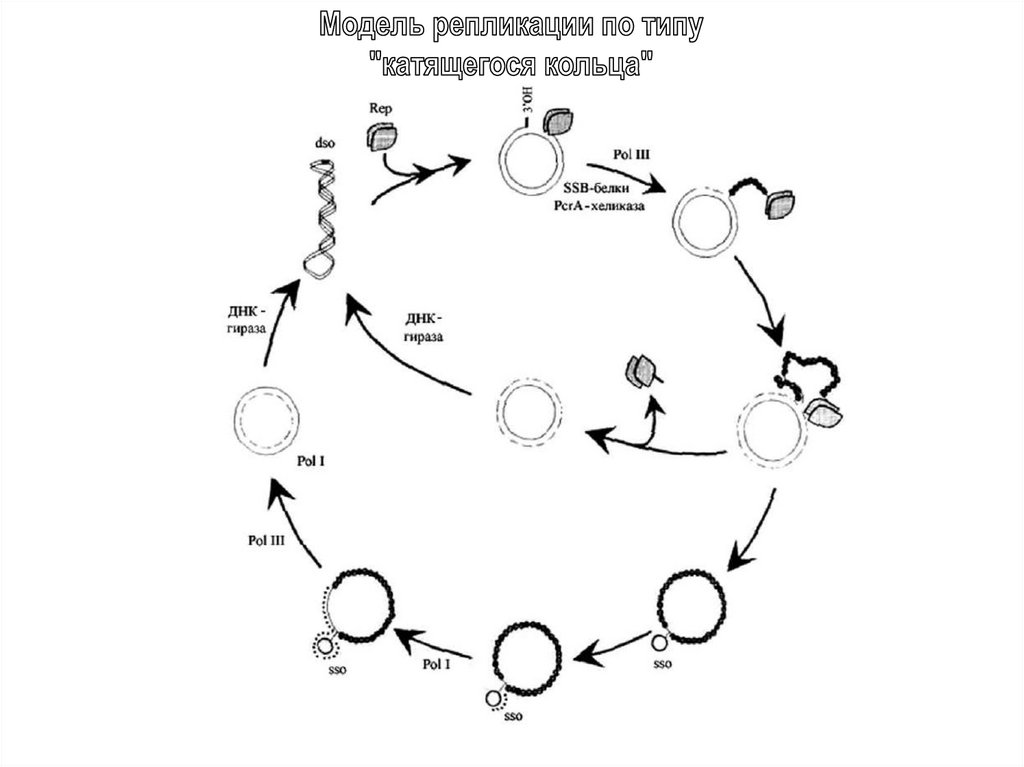
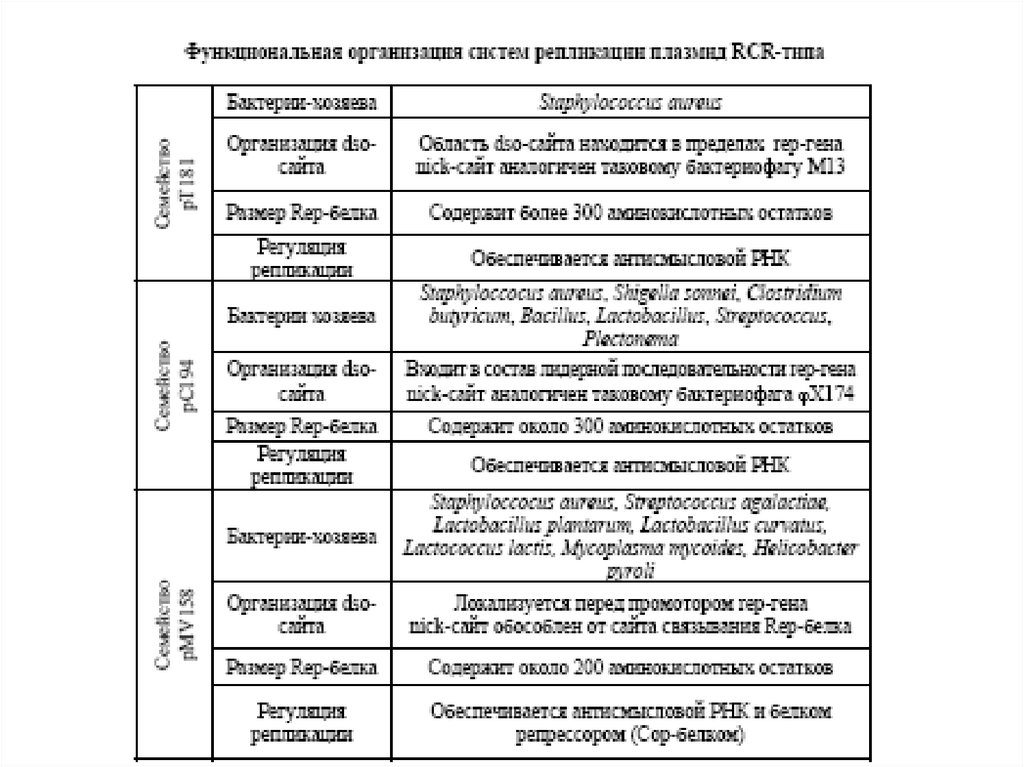
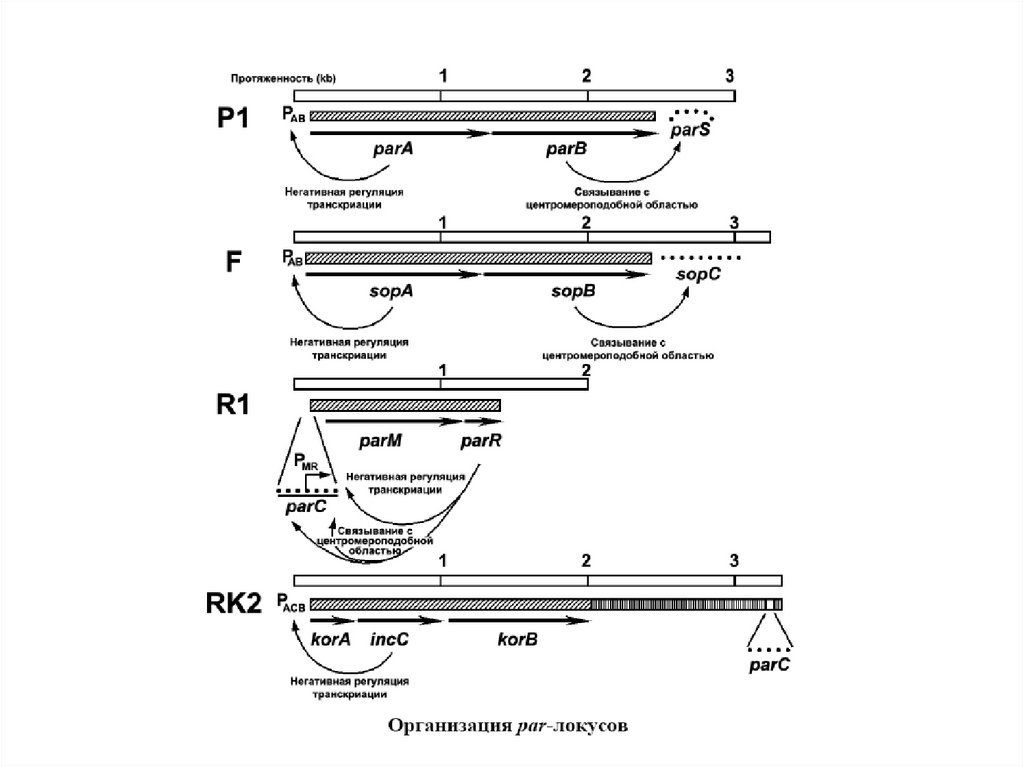
Организация типичных представителей пяти семейств плазмидRCR-типа
18.
19.
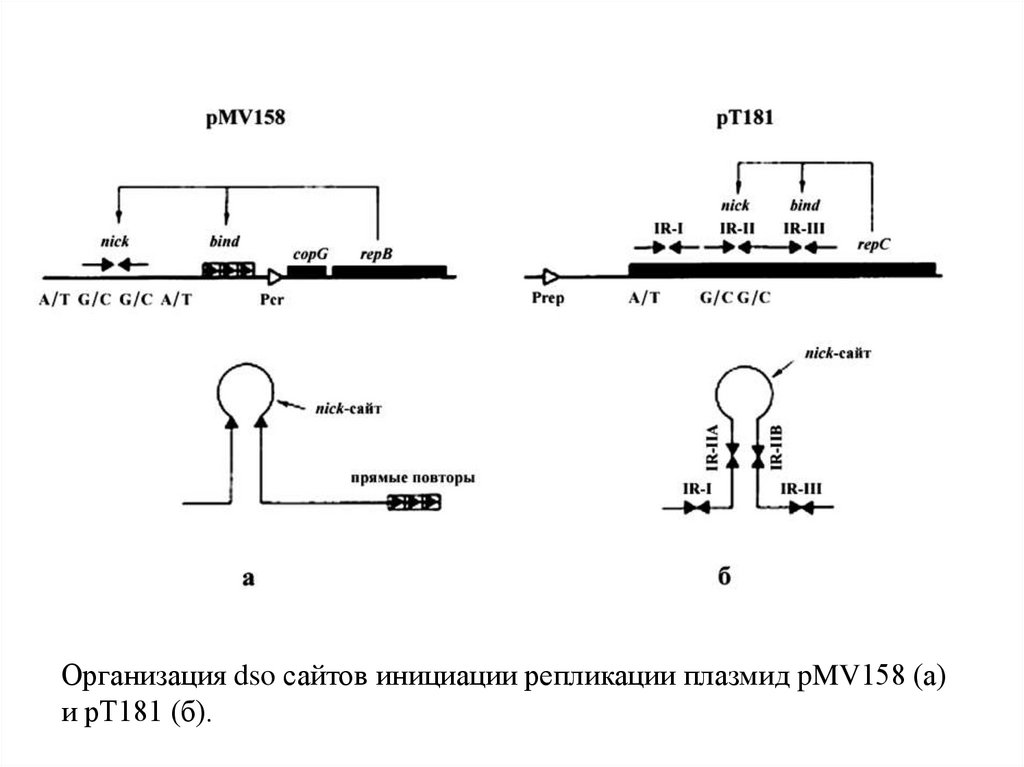
Организация dso сайтов инициации репликации плазмид pMV158 (а)и pT181 (б).
20.
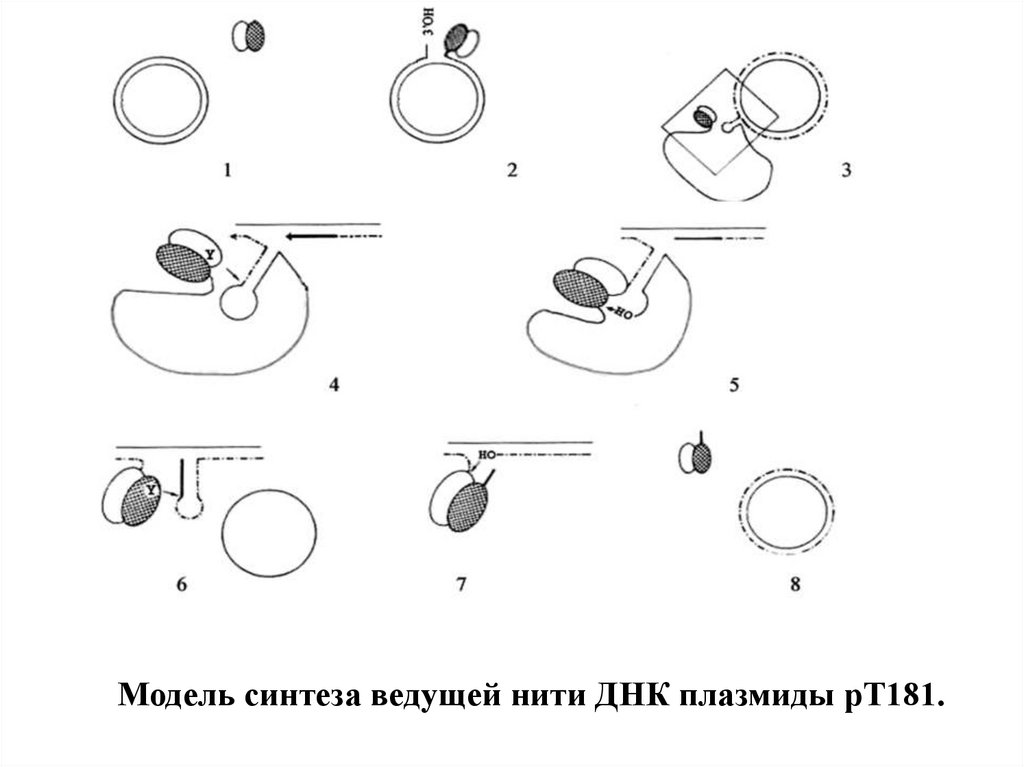
Модель синтеза ведущей нити ДНК плазмиды рТ181.21.
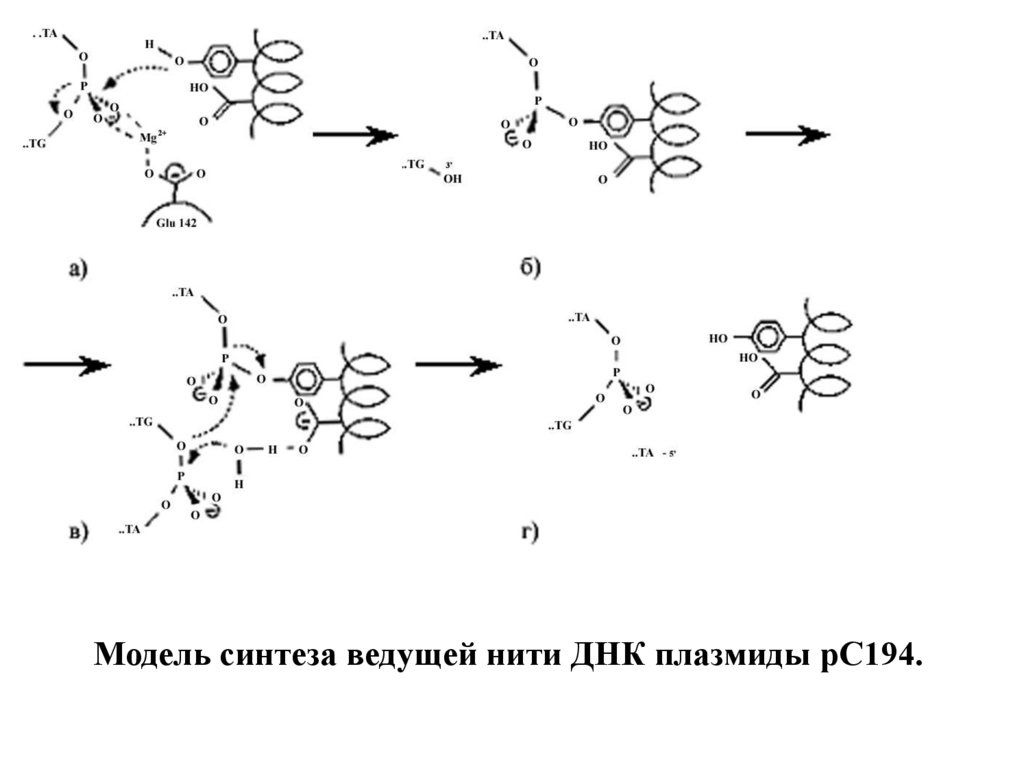
Модель синтеза ведущей нити ДНК плазмиды рС194.22.
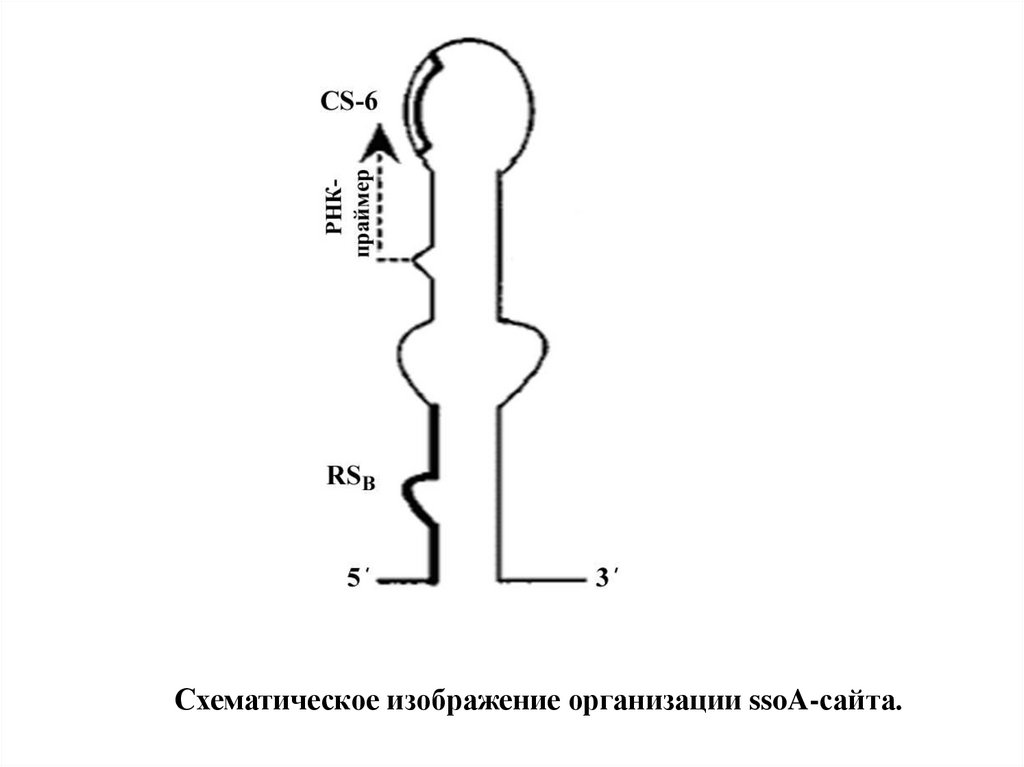
Схематическое изображение организации ssoA-сайта.23.
Схема регуляции копийности плазмиды рТ181.24.
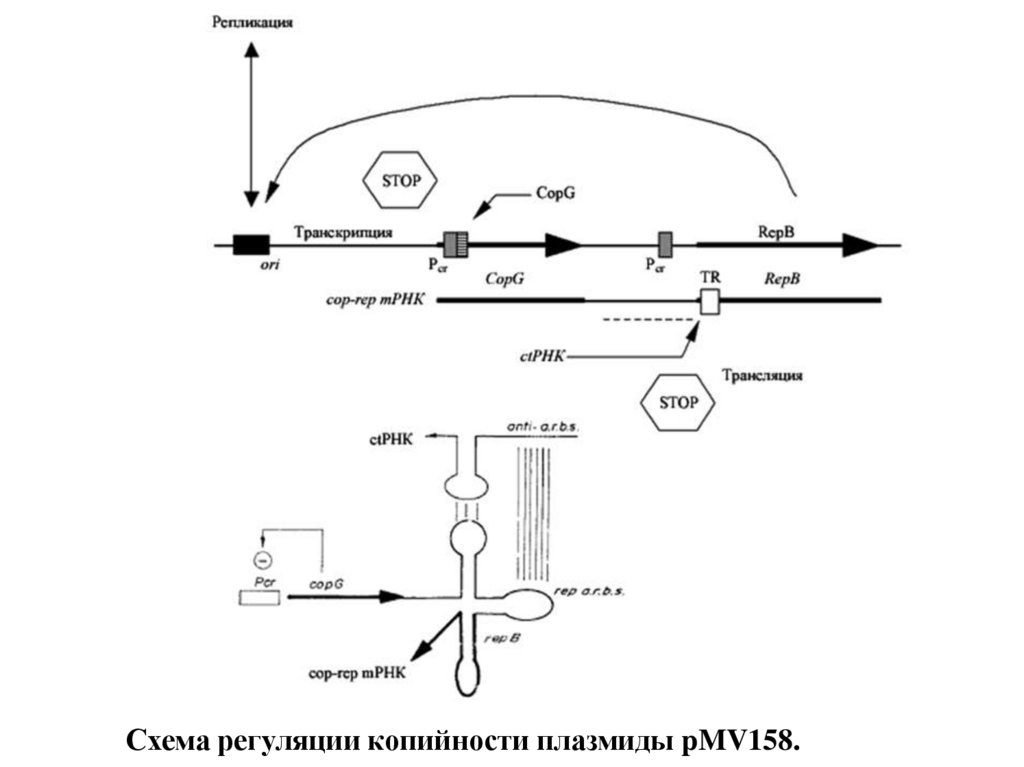
Схема регуляции копийности плазмиды pMV158.25.
26.
27.
28.
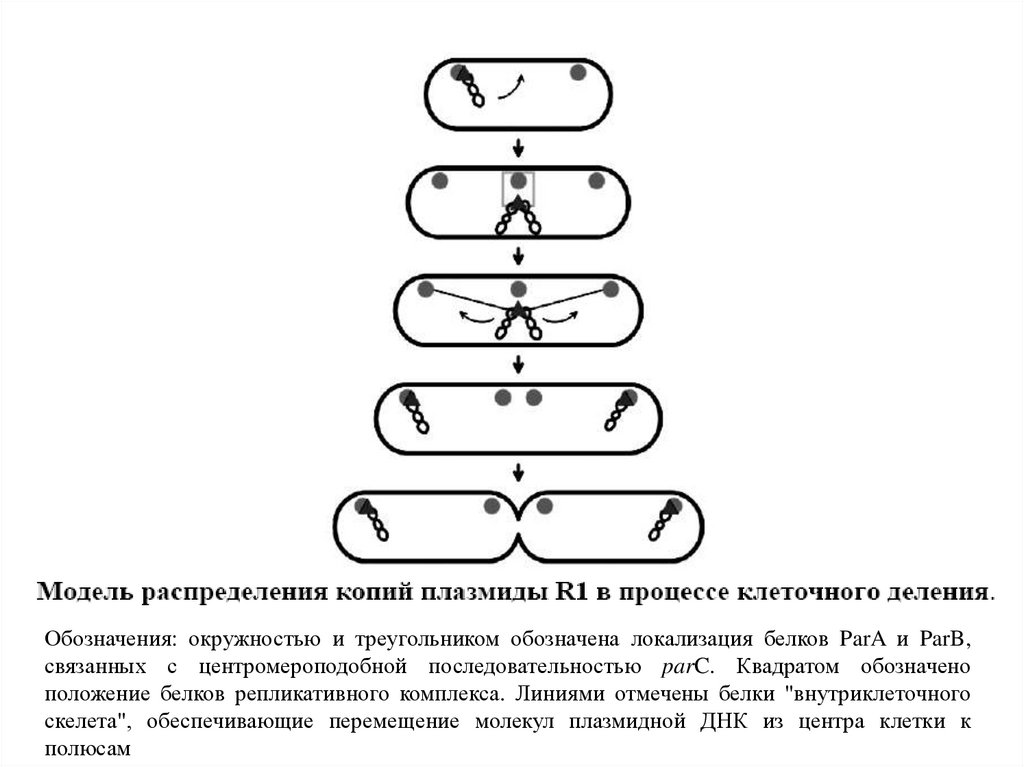
Модель, объясняющая роль par-белков29.
Обозначения: окружностью и треугольником обозначена локализация белков ParA и ParB,связанных с центромероподобной последовательностью parC. Квадратом обозначено
положение белков репликативного комплекса. Линиями отмечены белки "внутриклеточного
скелета", обеспечивающие перемещение молекул плазмидной ДНК из центра клетки к
полюсам
30.
31.
32.
33.
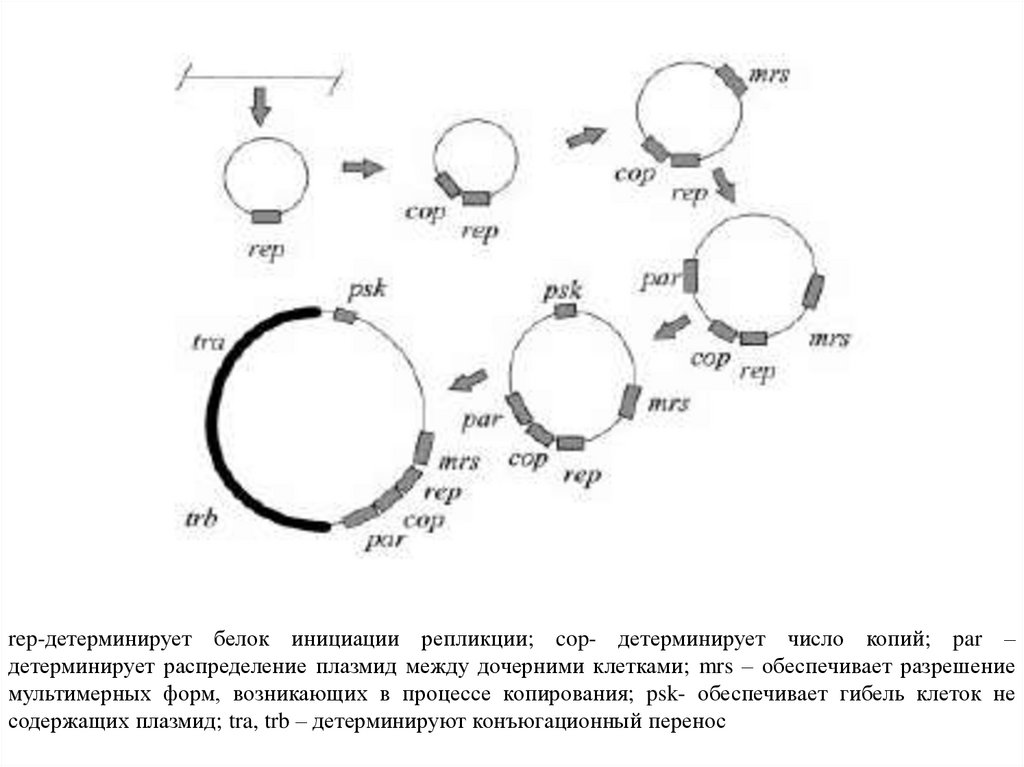
rep-детерминирует белок инициации репликции; cop- детерминирует число копий; par –детерминирует распределение плазмид между дочерними клетками; mrs – обеспечивает разрешение
мультимерных форм, возникающих в процессе копирования; psk- обеспечивает гибель клеток не
содержащих плазмид; tra, trb – детерминируют конъюгационный перенос
34.
35.
Источники выделения метилотрофных бактерий PseudomonasНомера штаммов
Источник выделения
1 – 70
г. Баку (нефтескважины, нефтехранилища)
71 – 73
г. Ленинград (электромеханический завод)
74 – 81
г.п. Малиновка (спиртзавод)
82 – 95
сан. "Криница" (лечебные грязи)
96 – 115
г. Толочин (бензохранилища)
116 – 135
г. Бобруйск (автобусная станция, железнодорожная станция)
136 – 150
г. Осиповичи (железная дорога, автозаправочные станции)
152 – 169
г.Смиловичи
(автозаправочные
бензохранилище)
170 – 189
дер. Косаричи (электростанция, молочная ферма)
190 – 219
г. Минск (автозаправочные станции)
220 – 249
г. Минск (нефтебазы)
250 – 257
г. Минск (завод медпрепаратов)
258 – 335
г. Минск (промышленные предприятия)
20% штаммов содержали плазмиды
станции,
кожфабрика,
36.
Источники выделения нафталинутилизирующих бактерий PseudomonasКол-во
штаммов
Источник выделения
81
Выделены из 205 проб почвы на территории г. Минска (завод
«Кристалл», АвтоВаз, Тракторный завод, Автомобильный
завод, завод Медпрепаратов, «Литмаш», станция аэрации, ж/д
станции,
автобусные
станции,
автобусные
остановки,
заправочные станции, автостоянки) и Минской области (ж/д
станции,
автобусные
станции,
автобусные
остановки,
заправочные станции, автостоянки)
12
Выделены из 29 проб почвы на территории Витебской области
(доломитовый завод, нефтебаза г. Витебск, «Нафтан»,
«Полимир» г. Полоцк, нефтеперерабатывающий завод г.
Новополоцк, автобусные остановки и автостоянки)
7
Выделены из 18 проб почвы на территории Брестской области
(автобусные остановки, заправочные станции, автостоянки)
NL73
1
Выделен из 6 проб почвы на территории Гомельской области
(предприятие «Центролит» г. Гомель)
NL59
1
Выделен из 14 проб, отобранных на территории Могилевской
области (гидролизный завод г. Бобруйск)
Штамм
NL1 – NL3, NL7 – NL14,
NL16 – NL18, NL20,
NL21, NL23, NL24, NL26
– NL28, NL30, NL33,
NL34, NL36 – NL39,
NL55 – NL58, NL61,
NL64, NL65 – NL72, AL1
– AL17 AL24 – AL31
AL33 – AL43
NL40 – NL46, NL52,
NL54, AL23, AL32, AL44
NL47
NL63
–
NL51,
NL62,
37. Электрофореграмма PstI-фрагментов плазмидной ДНК, выделенной из шести различных штаммов Pseudomonas.
12
3
4
5
6
Цифрами обозначены:
1 – штамм 7, 2 – штамм 139,
3 – штамм 180, 4 – штамм 195,
5 – штамм 198, 6 – штамм 253.
Электрофореграмма PstI-фрагментов плазмидной ДНК,
выделенной из шести различных штаммов Pseudomonas.
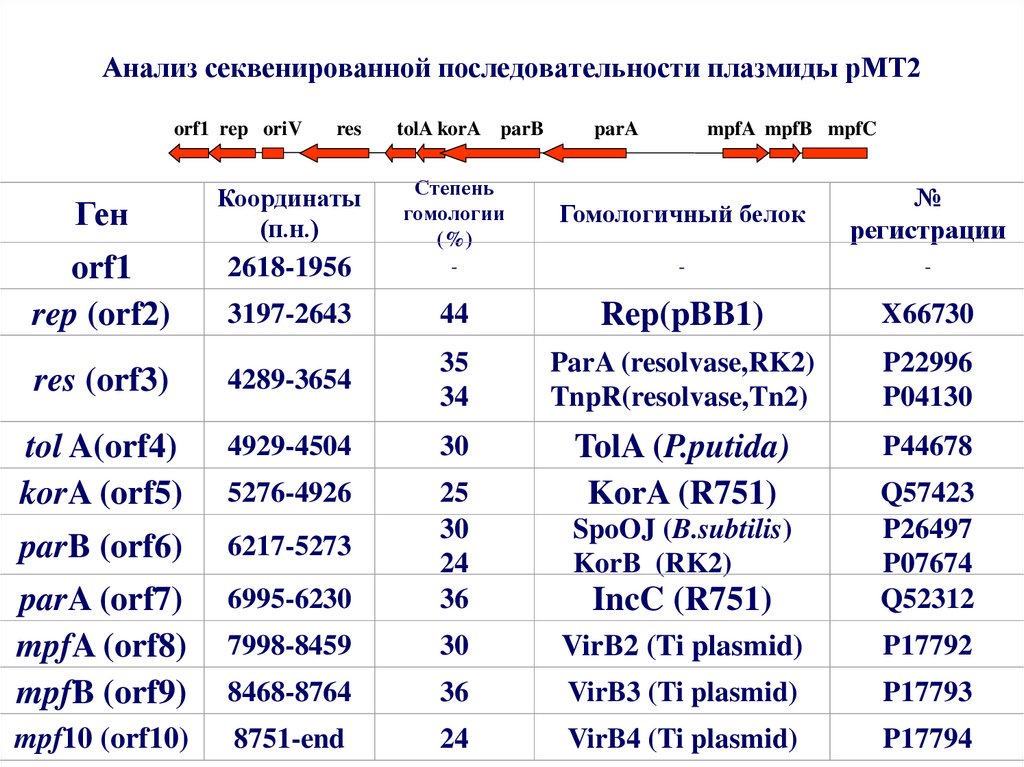
38. Анализ секвенированной последовательности плазмиды рМТ2
orf1 rep oriVres
tolA korA parB
parA
mpfA mpfB mpfC
Ген
Координаты
(п.н.)
Степень
гомологии
(%)
Гомологичный белок
№
регистрации
orf1
rep (orf2)
2618-1956
-
-
-
3197-2643
44
Rep(pBB1)
X66730
res (orf3)
4289-3654
35
34
ParA (resolvase,RK2)
TnpR(resolvase,Tn2)
P22996
P04130
tol A(orf4)
korA (orf5)
4929-4504
30
P44678
5276-4926
parB (orf6)
6217-5273
SpoOJ (B.subtilis)
KorB (RK2)
parA (orf7)
mpfA (orf8)
mpfB (orf9)
6995-6230
25
30
24
36
TolA (P.putida)
KorA (R751)
IncC (R751)
Q57423
P26497
P07674
Q52312
7998-8459
30
VirB2 (Ti plasmid)
P17792
8468-8764
36
VirB3 (Ti plasmid)
P17793
mpf10 (orf10)
8751-end
24
VirB4 (Ti plasmid)
P17794
39. Функциональный анализ rep-области плазмиды рМТ2 IncP-9
StuIПлазмида
pMT2
pBМТ
pBMT-N
pBMT-R
pBМТ-1
pBМТ-2
pBМТ-3
pACМТ
рACМТ-1
рACМТ-2
pACМТ-ΔparA
pACМТ-ΔparB
pACМТ-ΔkorA
рACМТ-ΔtolA
рACМТ-2N
mini-pMТ2
glII
BglII
tra
B
N
A
R
coI AvrII
vrII srII
glII
RsrII BglII
NcoI
parA
S
N
B
heI
NheI
alI
SalI
Частота образования
трансформантов (мкг ДНК)
P. putida
E. coli
parB
korAtolA res oriVrep orf1
Km
4
3
4.8 x 10
1.2 x 10
4
1.9 x 10
3
2.3 x 10
3
1.1x 10
1.5 x 10
4
2.4 x 10
2
2.8 x 10
0
0
0
0
3
3
5.8 x 10
1
2.4 x 10
4
2.3 x 10 *
4.8 x 10
3
2.1 x 10
3
1.3 x 10
3
4.1 x 10
н.о.
н.о.
н.о.
н.о.
н.о.
н.о.
н.о.
0
1.3 x 10
4
3.2 x 10
4
2.4 x 10
4
1.1 x 10
0
2
3
Примечание: н.о.- частота трансформации не определялась; * - образование трансформантов
наблюдалось только в присутствии ParB-белка.
40.
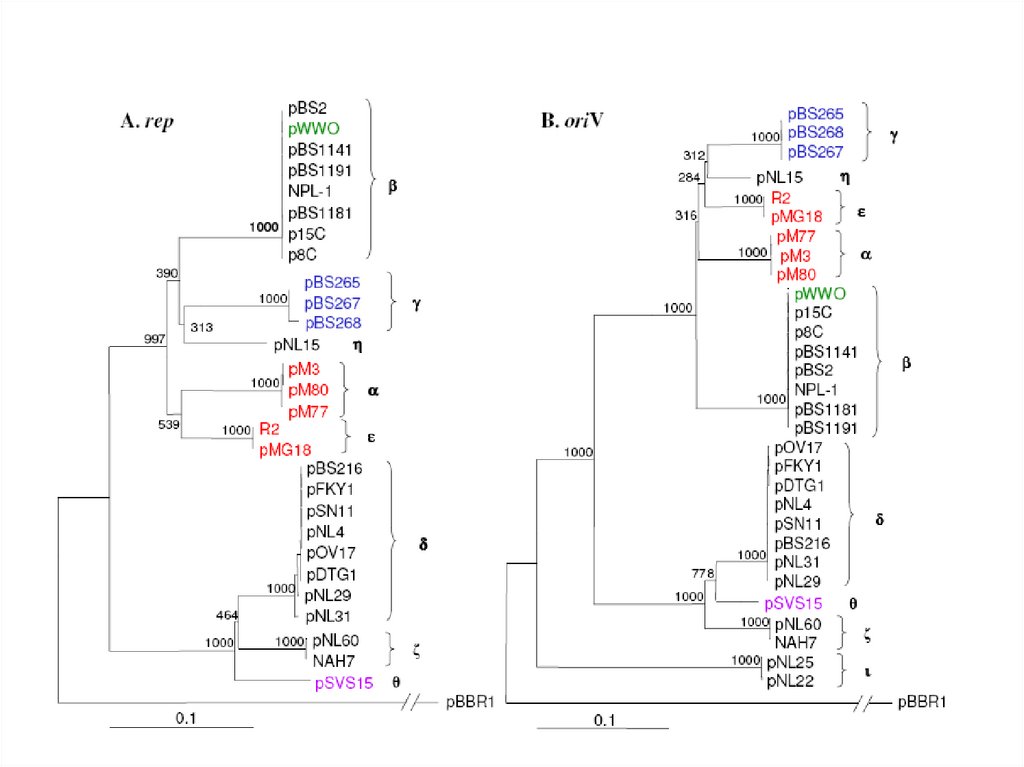
Рестрикционный анализ продуктов амплификацииrep-областей плазмид IncP-9.
Продукты амплификации, обработанные рестриктазой
HaeIII, полученны с помощью праймеров korA3Fa-rep3Rc,
при использовании в качестве матрицы тотальной ДНК
штаммов, содержащих плазмиды IncP-9 группы: 1 –
плазмида R2 (ε-подгуппа); 2 – плазмида pBS267 ( подгруппа); 3 – плазмида pBS101 ( -подгруппа); 4 –
плазмида штамма 15 (η-подгруппа); 5 – плазмида рМ3 ( подгруппа); 6 – 1 kb репер
Результаты рестрикционного анализа продуктов
амплификации rep-гена плазмид IncP-9.
Обработанные
рестриктазой
HaeIII
продукты
амплификации получены с помощью праймеров repF-repR
при использовании в качестве матрицы тотальной ДНК
штаммов, содержащих плазмиды группы IncP-9:1 – штамм
55 (α-подгруппа); 2 – рМ3 (α-подгруппа); 3 – pBS101 (βподгруппа); 4 – pBS267 ( -подгруппа); 5 – pBS216 ( подгруппа); 6 – штамма 4 ( -подгруппа); 7 – штамм 60 (ζподгруппа); 8 – 1 kb репер.
ι-подгруппа
Рестрикционный анализ продуктов амлификации oriV-сайта ферментом Tsp509I.
41.
42.
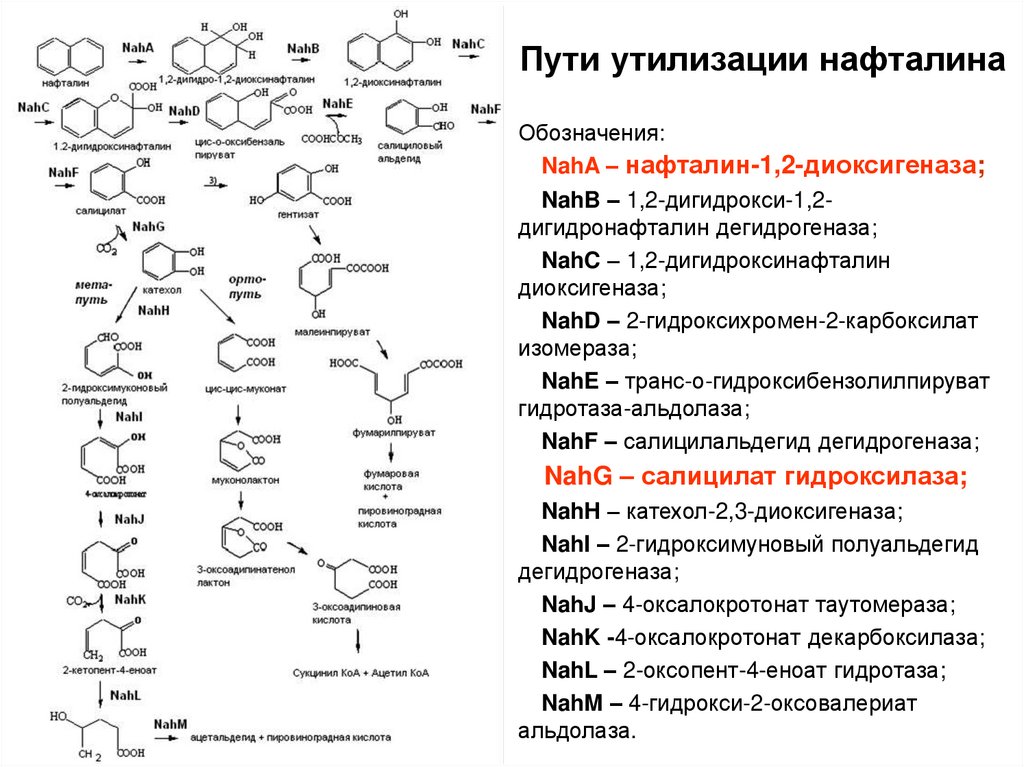
Пути утилизации нафталинаОбозначения:
NahA – нафталин-1,2-диоксигеназа;
NahB – 1,2-дигидрокси-1,2дигидронафталин дегидрогеназа;
NahC – 1,2-дигидроксинафталин
диоксигеназа;
NahD – 2-гидроксихромен-2-карбоксилат
изомераза;
NahE – транс-о-гидроксибензолилпируват
гидротаза-альдолаза;
NahF – салицилальдегид дегидрогеназа;
NahG – салицилат гидроксилаза;
NahH – катехол-2,3-диоксигеназа;
NahI – 2-гидроксимуновый полуальдегид
дегидрогеназа;
NahJ – 4-оксалокротонат таутомераза;
NahK -4-оксалокротонат декарбоксилаза;
NahL – 2-оксопент-4-еноат гидротаза;
NahM – 4-гидрокси-2-оксовалериат
альдолаза.
43.
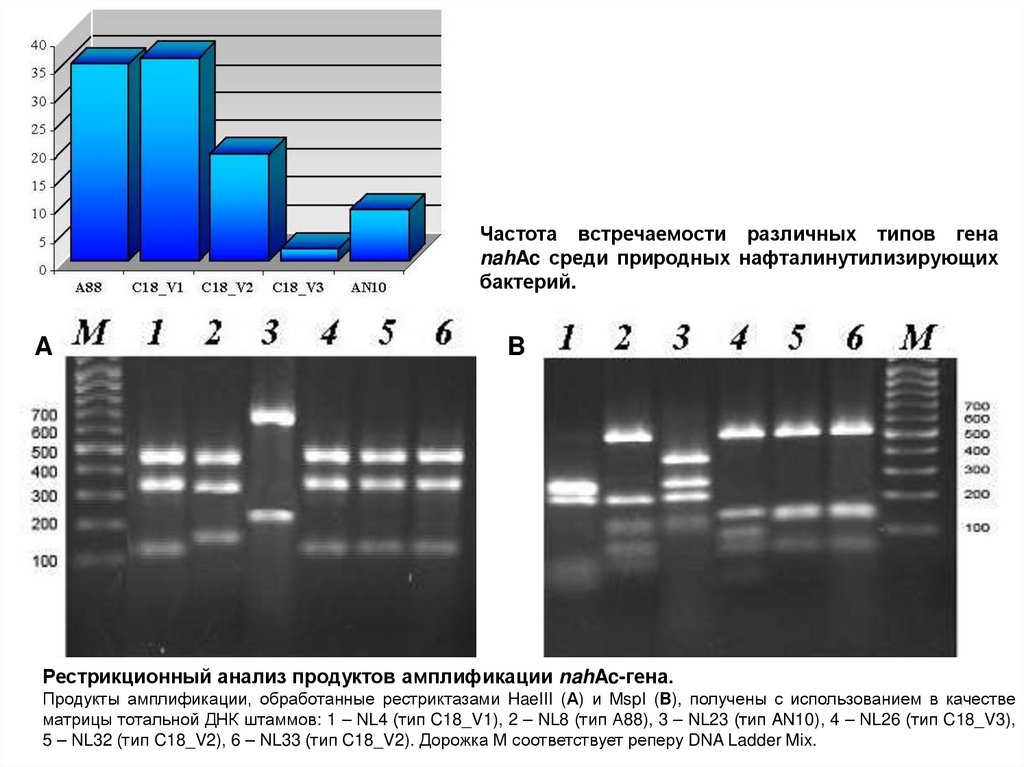
Частота встречаемости различных типов генаnahAc среди природных нафталинутилизирующих
бактерий.
A
B
Рестрикционный анализ продуктов амплификации nahAc-гена.
Продукты амплификации, обработанные рестриктазами HaeIII (А) и MspI (B), получены с использованием в качестве
матрицы тотальной ДНК штаммов: 1 – NL4 (тип C18_V1), 2 – NL8 (тип А88), 3 – NL23 (тип AN10), 4 – NL26 (тип C18_V3),
5 – NL32 (тип C18_V2), 6 – NL33 (тип C18_V2). Дорожка М соответствует реперу DNA Ladder Mix.
44.
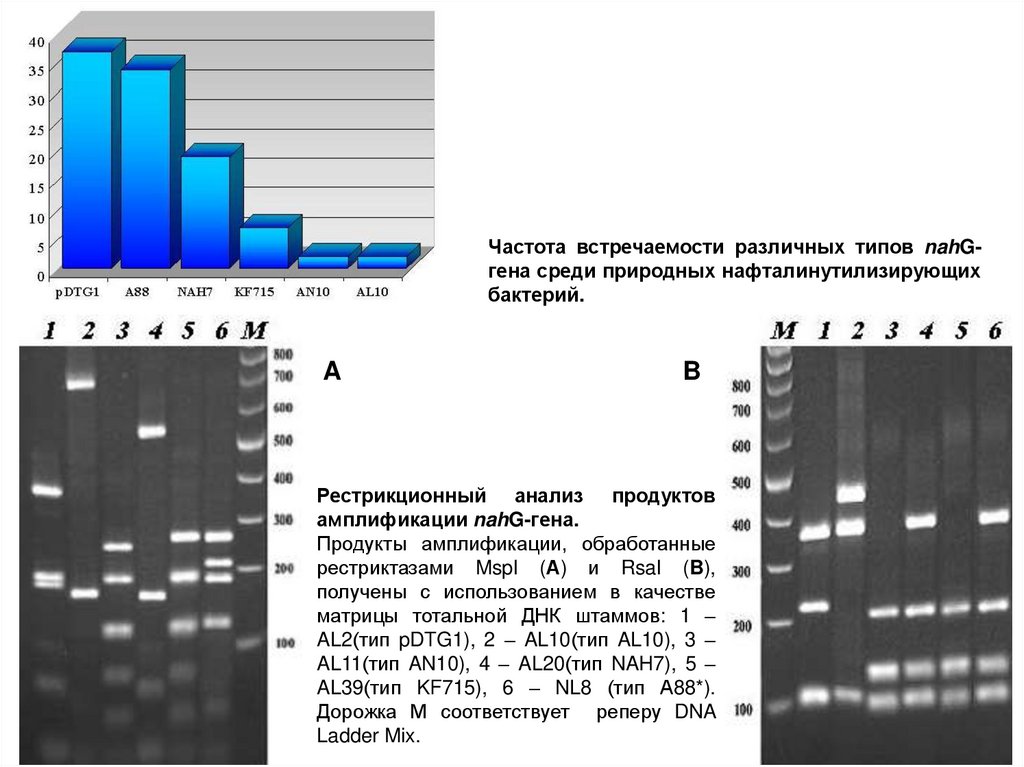
Частота встречаемости различных типов nahGгена среди природных нафталинутилизирующихбактерий.
A
B
Рестрикционный анализ продуктов
амплификации nahG-гена.
Продукты амплификации, обработанные
рестриктазами MspI (А) и RsaI (B),
получены с использованием в качестве
матрицы тотальной ДНК штаммов: 1 –
AL2(тип pDTG1), 2 – AL10(тип AL10), 3 –
AL11(тип AN10), 4 – AL20(тип NAH7), 5 –
AL39(тип KF715), 6 – NL8 (тип А88*).
Дорожка М соответствует реперу DNA
Ladder Mix.
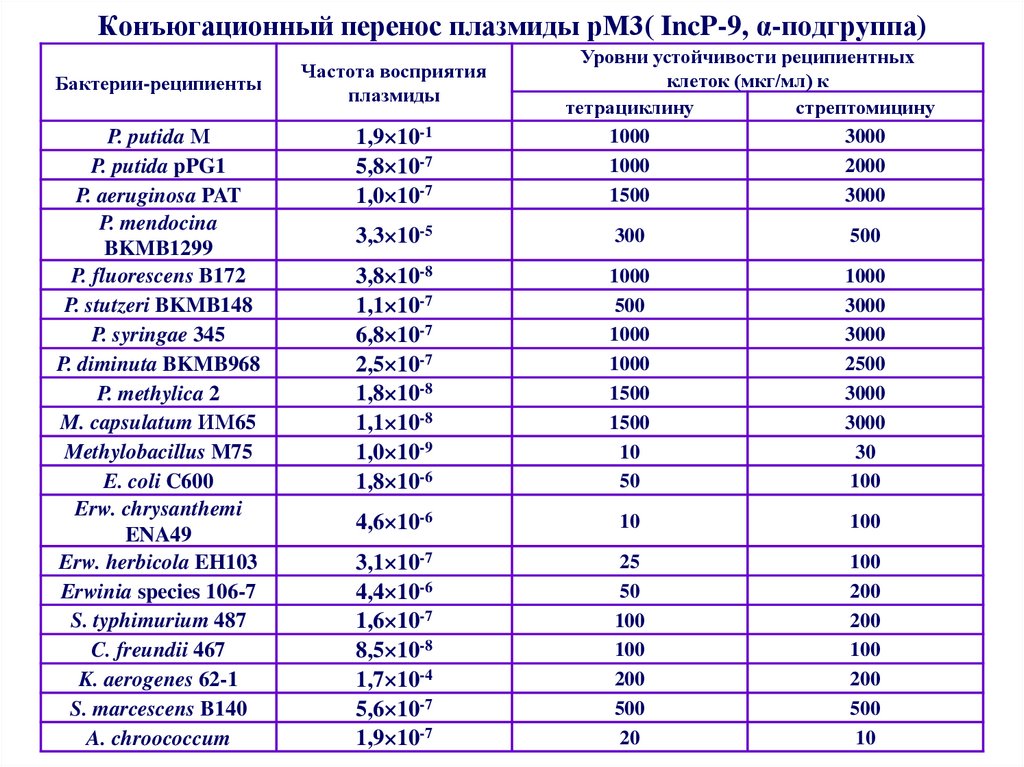
45. Конъюгационный перенос плазмиды рМ3( IncP-9, α-подгруппа)
Бактерии-реципиентыP. putida М
P. putida pPG1
P. aeruginosa PAT
P. mendocina
BKMB1299
P. fluorescens B172
P. stutzeri BKMB148
P. syringae 345
P. diminuta BKMB968
P. methylica 2
M. capsulatum ИМ65
Methylobacillus M75
E. coli C600
Erw. chrysanthemi
ENA49
Erw. herbicola EH103
Erwinia species 106-7
S. typhimurium 487
C. freundii 467
K. aerogenes 62-1
S. marcescens B140
A. chroococcum
Частота восприятия
плазмиды
1,9 10-1
5,8 10-7
1,0 10-7
Уровни устойчивости реципиентных
клеток (мкг/мл) к
тетрациклину
стрептомицину
1000
3000
1000
2000
1500
3000
3,3 10-5
300
500
3,8 10-8
1,1 10-7
6,8 10-7
2,5 10-7
1,8 10-8
1,1 10-8
1,0 10-9
1,8 10-6
1000
500
1000
1000
1500
1500
10
50
1000
3000
3000
2500
3000
3000
30
100
4,6 10-6
10
100
3,1 10-7
4,4 10-6
1,6 10-7
8,5 10-8
1,7 10-4
5,6 10-7
1,9 10-7
25
50
100
100
200
500
20
100
200
200
100
200
500
10
46. Стабильность наследования плазмиды рМ3 (IncP-9, α-подгруппа) различными хозяевами в зависимости от температуры выращивания
Стабильность наследования плазмиды рМ3 (IncP-9, αподгруппа) различными хозяевами в зависимости оттемпературы выращивания
Бактерии-хозяева
P. aeruginosa PAT
P. mendocina BKMB1299
P. stutzeri BKMB148
P. diminuta BKMB968
P. putida pPG1
P. fluorescens B172
P. methylica 2
M. capsulatum ИМ65
A. chroococcum
E. coli C600
Erw. chrysanthemi ENA49
Erw. herbicola EH103
Erwinia species 106-7
S. typhimurium 487
K. aerogenes 62-1
C. freundii 467
S. marcescens B140
Сохранность плазмиды (в %) при
28 С
100
100
100
100
100
100
100
100
100
68
99
89
90
25
99
47
99
37 С
100
100
100
100
0
0
0
0
0
0
0
0
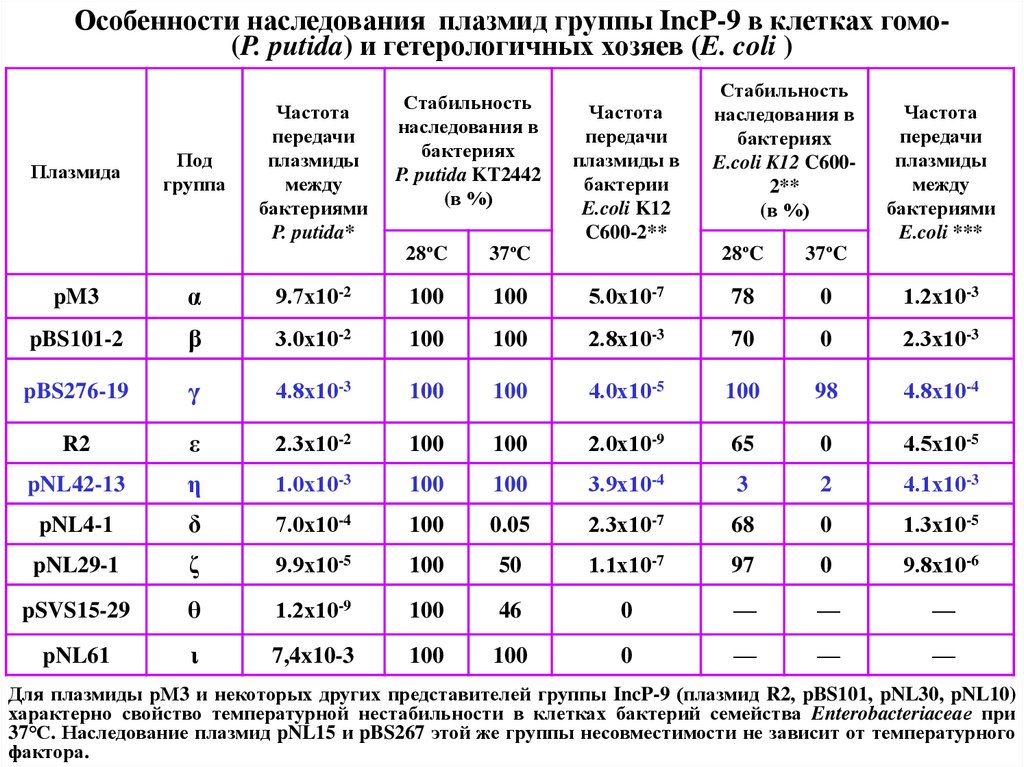
47.
Особенности наследования плазмид группы IncP-9 в клетках гомо(P. putida) и гетерологичных хозяев (E. coli )Плазмида
Под
группа
Частота
передачи
плазмиды
между
бактериями
P. putida*
Стабильность
наследования в
бактериях
P. putida KT2442
(в %)
28ºC
37ºC
Частота
передачи
плазмиды в
бактерии
E.coli K12
C600-2**
Стабильность
наследования в
бактериях
E.coli K12 C6002**
(в %)
28ºC
37ºC
Частота
передачи
плазмиды
между
бактериями
E.coli ***
pM3
α
9.7х10-2
100
100
5.0х10-7
78
0
1.2х10-3
pBS101-2
β
3.0х10-2
100
100
2.8х10-3
70
0
2.3х10-3
pBS276-19
γ
4.8х10-3
100
100
4.0х10-5
100
98
4.8х10-4
R2
ε
2.3х10-2
100
100
2.0х10-9
65
0
4.5х10-5
pNL42-13
η
1.0х10-3
100
100
3.9х10-4
3
2
4.1х10-3
pNL4-1
δ
7.0х10-4
100
0.05
2.3х10-7
68
0
1.3х10-5
pNL29-1
ζ
9.9х10-5
100
50
1.1х10-7
97
0
9.8х10-6
pSVS15-29
θ
1.2х10-9
100
46
0
—
—
—
pNL61
ι
7,4x10-3
100
100
0
—
—
—
Для плазмиды рМ3 и некоторых других представителей группы IncP-9 (плазмид R2, pBS101, pNL30, pNL10)
характерно свойство температурной нестабильности в клетках бактерий семейства Enterobacteriaceaе при
37°С. Наследование плазмид pNL15 и pBS267 этой же группы несовместимости не зависит от температурного
фактора.
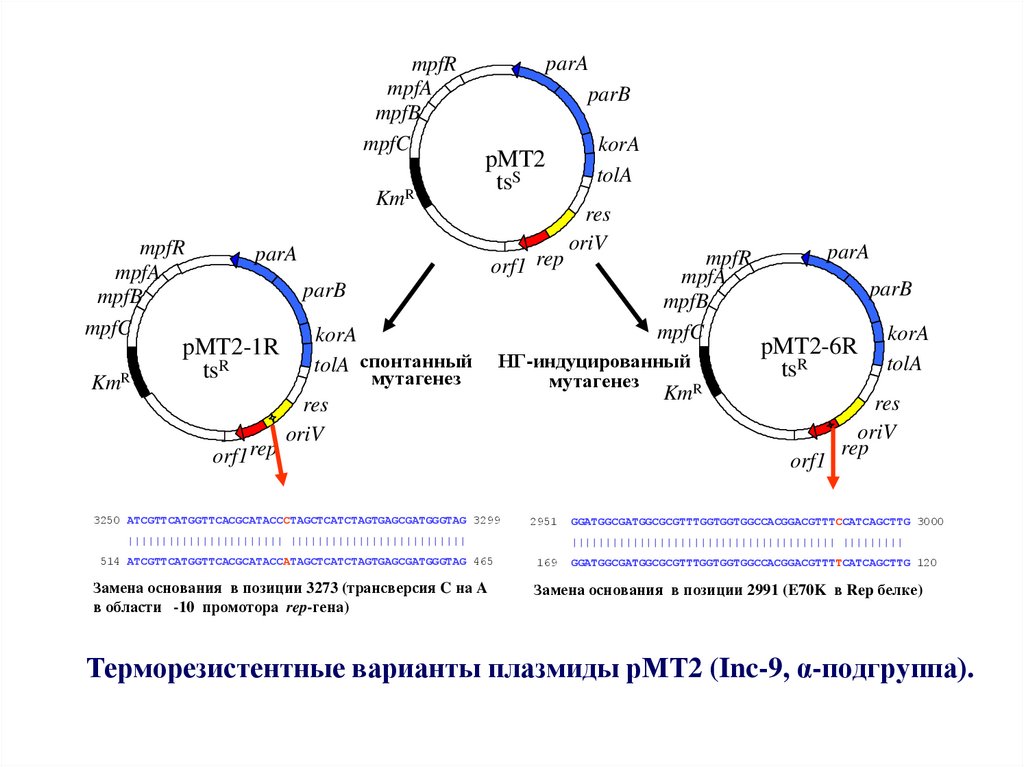
48. Терморезистентные варианты плазмиды рМТ2 (Inc-9, α-подгруппа).
mpfRmpfA
mpfB
mpfC
KmR
mpfR
mpfA
mpfB
mpfC
KmR
parA
parB
pMT2
tsS
parA
orf1 rep
parB
pMT2-1R
tsR
orf1 rep
korA
tolA спонтанный
мутагенез
korA
tolA
res
oriV
mpfR
mpfA
mpfB
mpfC
НГ-индуцированный
мутагенез
R
parB
pMT2-6R
tsR
Km
res
oriV
3250 ATCGTTCATGGTTCACGCATACCCTAGCTCATCTAGTGAGCGATGGGTAG 3299
parA
orf1
2951
||||||||||||||||||||||| ||||||||||||||||||||||||||
korA
tolA
res
oriV
rep
GGATGGCGATGGCGCGTTTGGTGGTGGCCACGGACGTTTCCATCAGCTTG 3000
||||||||||||||||||||||||||||||||||||||| |||||||||
514 ATCGTTCATGGTTCACGCATACCATAGCTCATCTAGTGAGCGATGGGTAG 465
169
Замена основания в позиции 3273 (трансверсия C на A
в области -10 промотора rep-гена)
Замена основания в позиции 2991 (E70K в Rep белке)
GGATGGCGATGGCGCGTTTGGTGGTGGCCACGGACGTTTTCATCAGCTTG 120
Терморезистентные варианты плазмиды рМТ2 (Inc-9, α-подгруппа).
49. Схема клонирования исходного и мутантного промоторов в составе плазмиды pRWX.
xylEEcoRI
pRWX-pr
BamHI
BamHI
pRWX
IncP
xylE
Tc
EcoRI
EcoRI
xylE
pRWX-pm
BamHI
Схема клонирования исходного и мутантного промоторов в составе
плазмиды pRWX.
50.
tac-promoterEcoRI
rep
pOLE1-rep6
SalI
EcoRI
SalI
Sm
pOLE1
tac-promoter
IncQ
tac-promoter
EcoRI
rep
pOLE1-rep
SalI
Cхема клонирования исходного и мутантного rep-генов в составе
плазмиды pOLE1.
51.
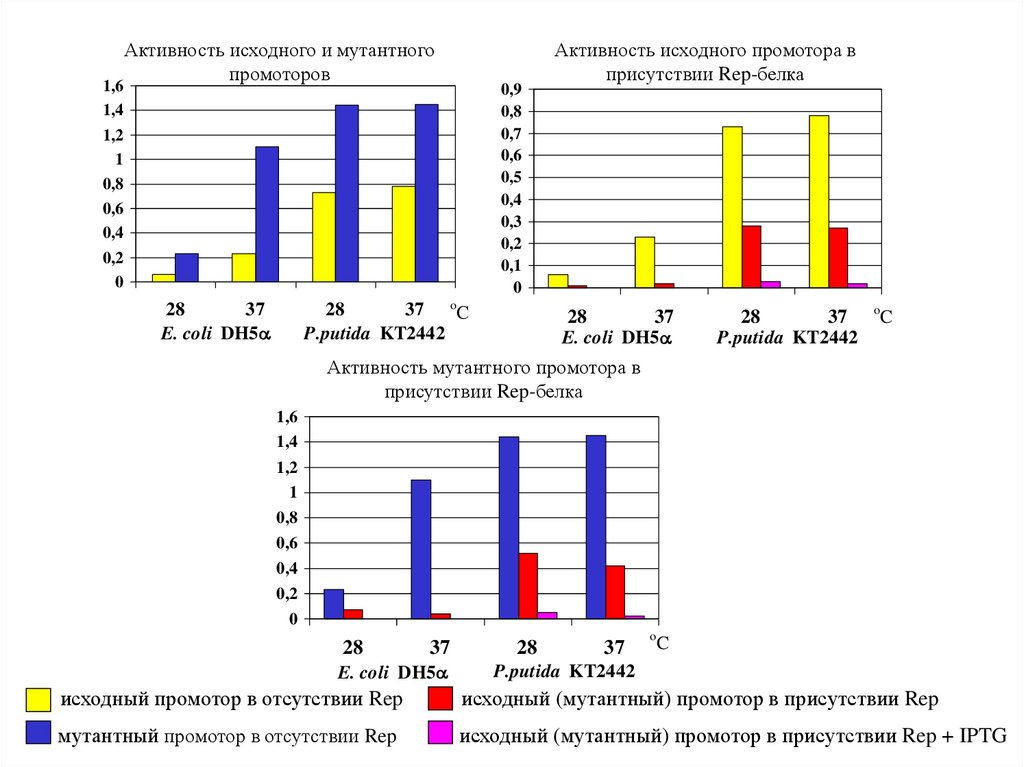
1,61,4
1,2
1
0,8
0,6
0,4
0,2
0
Активность исходного и мутантного
промоторов
0,9
0,8
0,7
0,6
0,5
0,4
0,3
0,2
0,1
0
28
37 oC
P.putida KT2442
28
37
E. coli DH5
Активность исходного промотора в
присутствии Rep-белка
28
37
E. coli DH5
28
37
P.putida KT2442
o
C
Активность мутантного промотора в
присутствии Rep-белка
1,6
1,4
1,2
1
0,8
0,6
0,4
0,2
0
28
37
E. coli DH5
28
37
o
C
P.putida KT2442
исходный промотор в отсутствии Rep
исходный (мутантный) промотор в присутствии Rep
мутантный промотор в отсутствии Rep
исходный (мутантный) промотор в присутствии Rep + IPTG
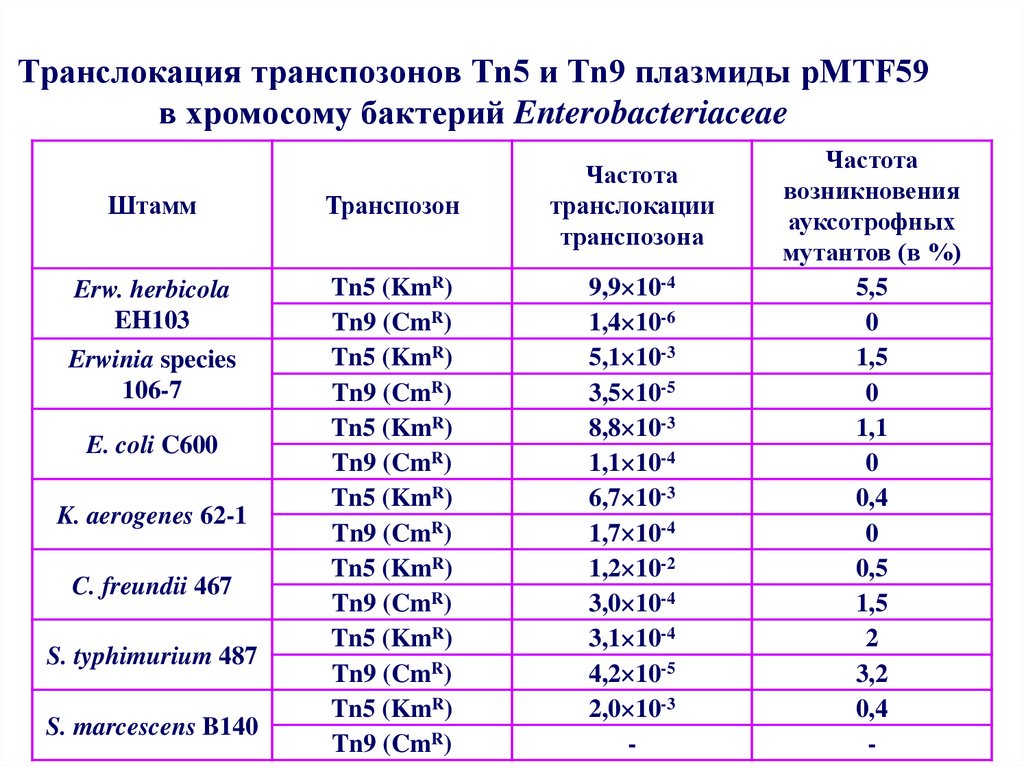
52. Транслокация транспозонов Tn5 и Tn9 плазмиды рМТF59 в хромосому бактерий Enterobacteriaceae
ШтаммТранспозон
Частота
транслокации
транспозона
Erw. herbicola
EH103
Tn5 (KmR)
Tn9 (CmR)
Tn5 (KmR)
Tn9 (CmR)
Tn5 (KmR)
Tn9 (CmR)
Tn5 (KmR)
Tn9 (CmR)
Tn5 (KmR)
Tn9 (CmR)
Tn5 (KmR)
Tn9 (CmR)
Tn5 (KmR)
Tn9 (CmR)
9,9 10-4
1,4 10-6
5,1 10-3
3,5 10-5
8,8 10-3
1,1 10-4
6,7 10-3
1,7 10-4
1,2 10-2
3,0 10-4
3,1 10-4
4,2 10-5
2,0 10-3
-
Erwinia species
106-7
E. coli C600
K. aerogenes 62-1
C. freundii 467
S. typhimurium 487
S. marcescens B140
Частота
возникновения
ауксотрофных
мутантов (в %)
5,5
0
1,5
0
1,1
0
0,4
0
0,5
1,5
2
3,2
0,4
-
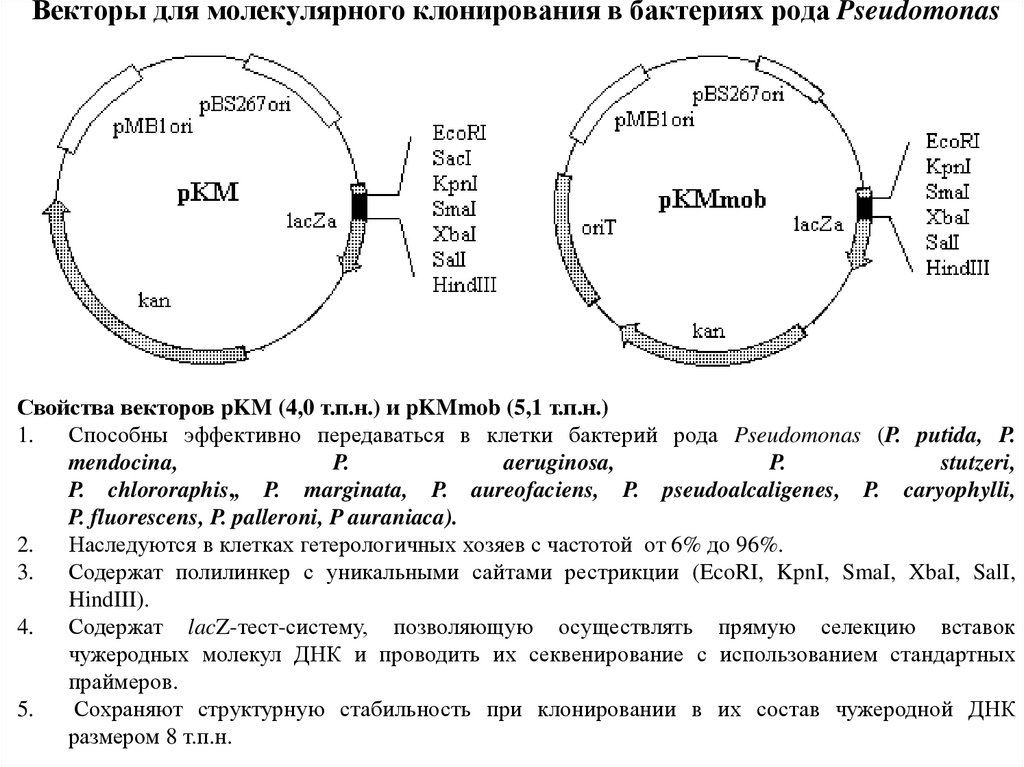
53. Векторы для молекулярного клонирования в бактериях рода Pseudomonas
Свойства векторов pKM (4,0 т.п.н.) и pKMmob (5,1 т.п.н.)1. Способны эффективно передаваться в клетки бактерий рода Pseudomonas (P. putida, P.
mendocina,
P.
aeruginosa,
P.
stutzeri,
P. chlororaphis,, P. marginata, P. aureofaciens, P. pseudoalcaligenes, P. caryophylli,
P. fluorescens, P. palleroni, P auraniaca).
2.
Наследуются в клетках гетерологичных хозяев с частотой от 6% до 96%.
3.
Содержат полилинкер с уникальными сайтами рестрикции (EcoRI, KpnI, SmaI, XbaI, SalI,
HindIII).
4.
Содержат lacZ-тест-систему, позволяющую осуществлять прямую селекцию вставок
чужеродных молекул ДНК и проводить их секвенирование с использованием стандартных
праймеров.
5.
Сохраняют структурную стабильность при клонировании в их состав чужеродной ДНК
размером 8 т.п.н.
54.
55.
Источники выделения спорообразующих бактерий B.subtilisНомера
штаммов
Источник выделения
N1, 2, 4K, 21Z, 31, 34,
37, 40, 43, 45, 72, 124,
Минск и Минская область (луг, поле, клумбы,
127, 190, 195, 200, 240,
лес, оз. Нарочь)
245, 260, 269, 270, 275,
280, 285
57, 60, 63, 64, 68, 102, Брест и Брестская область (огород, клумбы, р.
109
Бобрик)
1, 4, 15, 50, 52, 54, 57, 59
Гомельская область (поле, луг, оз. Рисловское,
оз. Святское)
8, 19, 22, 23, 25, 29
Гродненская область (луг, лес, клумбы)
110, 114, 160, 165, 170
Могилевская область (огород, клумбы, поле)
105, 205, 209, 226, 230
Витебская область (огород, луг, лес)
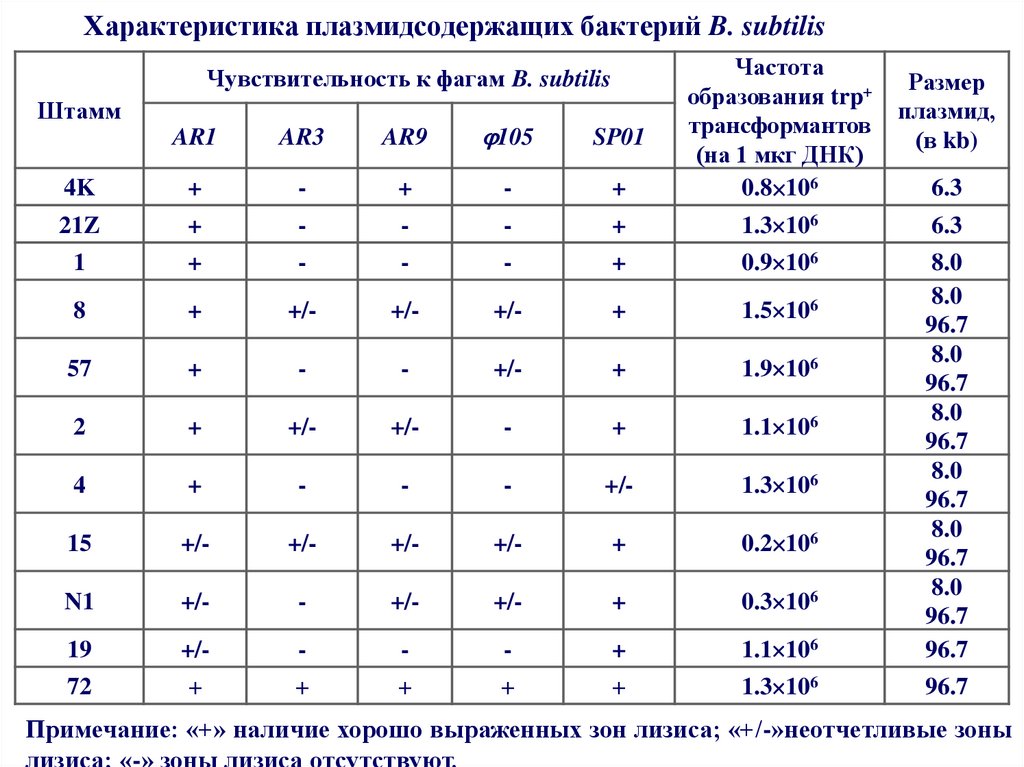
56. Характеристика плазмидсодержащих бактерий B. subtilis
Характеристика плазмидсодержащих бактерий B. subtilisAR1
AR3
AR9
105
SP01
4K
+
-
+
-
+
Частота
образования trp+
трансформантов
(на 1 мкг ДНК)
0.8 106
21Z
+
-
-
-
+
1.3 106
6.3
1
+
-
-
-
+
0.9 106
8
+
+/-
+/-
+/-
+
1.5 106
57
+
-
-
+/-
+
1.9 106
2
+
+/-
+/-
-
+
1.1 106
4
+
-
-
-
+/-
1.3 106
15
+/-
+/-
+/-
+/-
+
0.2 106
N1
+/-
-
+/-
+/-
+
0.3 106
19
+/-
-
-
-
+
1.1 106
8.0
8.0
96.7
8.0
96.7
8.0
96.7
8.0
96.7
8.0
96.7
8.0
96.7
96.7
72
+
+
+
+
+
1.3 106
96.7
Чувствительность к фагам B. subtilis
Штамм
Размер
плазмид,
(в kb)
6.3
Примечание: «+» наличие хорошо выраженных зон лизиса; «+/-»неотчетливые зоны
лизиса; «-» зоны лизиса отсутствуют.
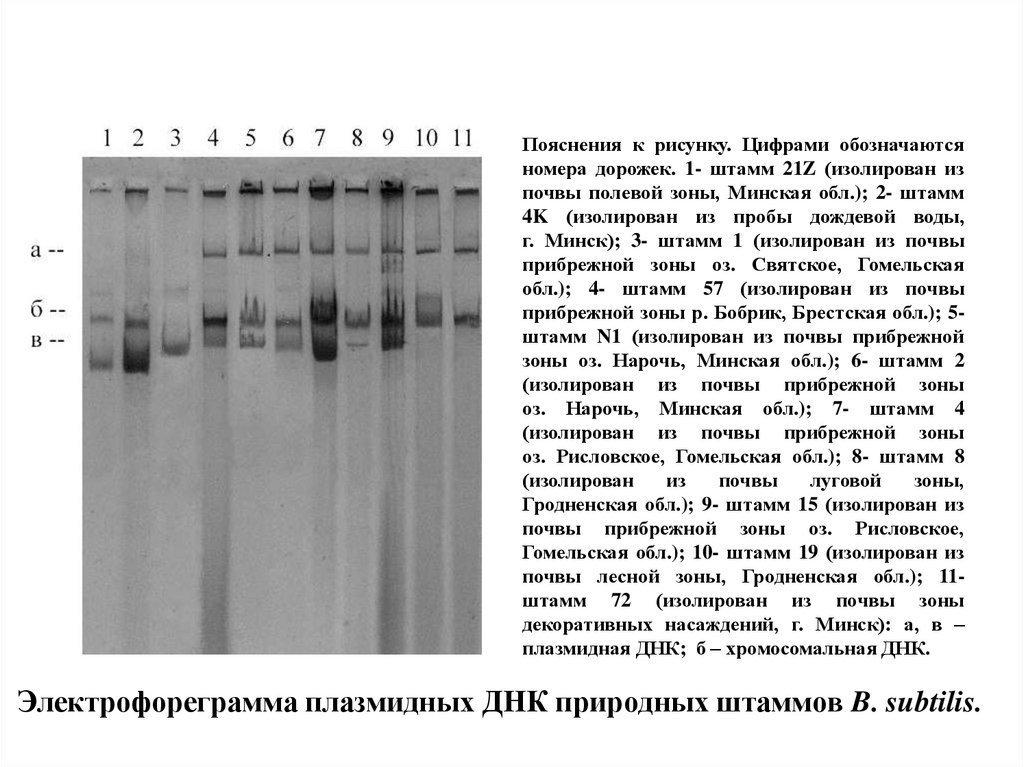
57. Электрофореграмма плазмидных ДНК природных штаммов B. subtilis.
Пояснения к рисунку. Цифрами обозначаютсяномера дорожек. 1- штамм 21Z (изолирован из
почвы полевой зоны, Минская обл.); 2- штамм
4K (изолирован из пробы дождевой воды,
г. Минск); 3- штамм 1 (изолирован из почвы
прибрежной зоны оз. Святское, Гомельская
обл.); 4- штамм 57 (изолирован из почвы
прибрежной зоны р. Бобрик, Брестская обл.); 5штамм N1 (изолирован из почвы прибрежной
зоны оз. Нарочь, Минская обл.); 6- штамм 2
(изолирован из почвы прибрежной зоны
оз. Нарочь, Минская обл.); 7- штамм 4
(изолирован из почвы прибрежной зоны
оз. Рисловское, Гомельская обл.); 8- штамм 8
(изолирован
из
почвы
луговой
зоны,
Гродненская обл.); 9- штамм 15 (изолирован из
почвы прибрежной зоны оз. Рисловское,
Гомельская обл.); 10- штамм 19 (изолирован из
почвы лесной зоны, Гродненская обл.); 11штамм 72 (изолирован из почвы зоны
декоративных насаждений, г. Минск): а, в –
плазмидная ДНК; б – хромосомальная ДНК.
Электрофореграмма плазмидных ДНК природных штаммов B. subtilis.
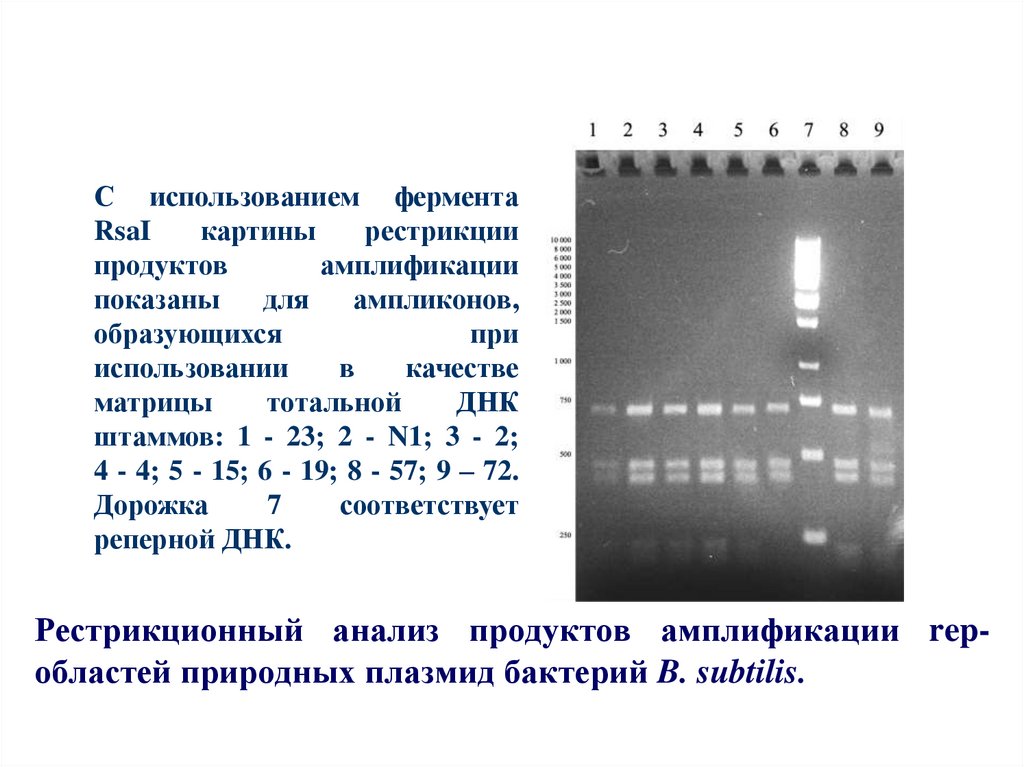
58. Рестрикционный анализ продуктов амплификации rep-областей природных плазмид бактерий B. subtilis.
С использованием ферментаRsaI
картины
рестрикции
продуктов
амплификации
показаны
для
ампликонов,
образующихся
при
использовании
в
качестве
матрицы
тотальной
ДНК
штаммов: 1 - 23; 2 - N1; 3 - 2;
4 - 4; 5 - 15; 6 - 19; 8 - 57; 9 – 72.
Дорожка
7
соответствует
реперной ДНК.
Рестрикционный анализ продуктов амплификации repобластей природных плазмид бактерий B. subtilis.
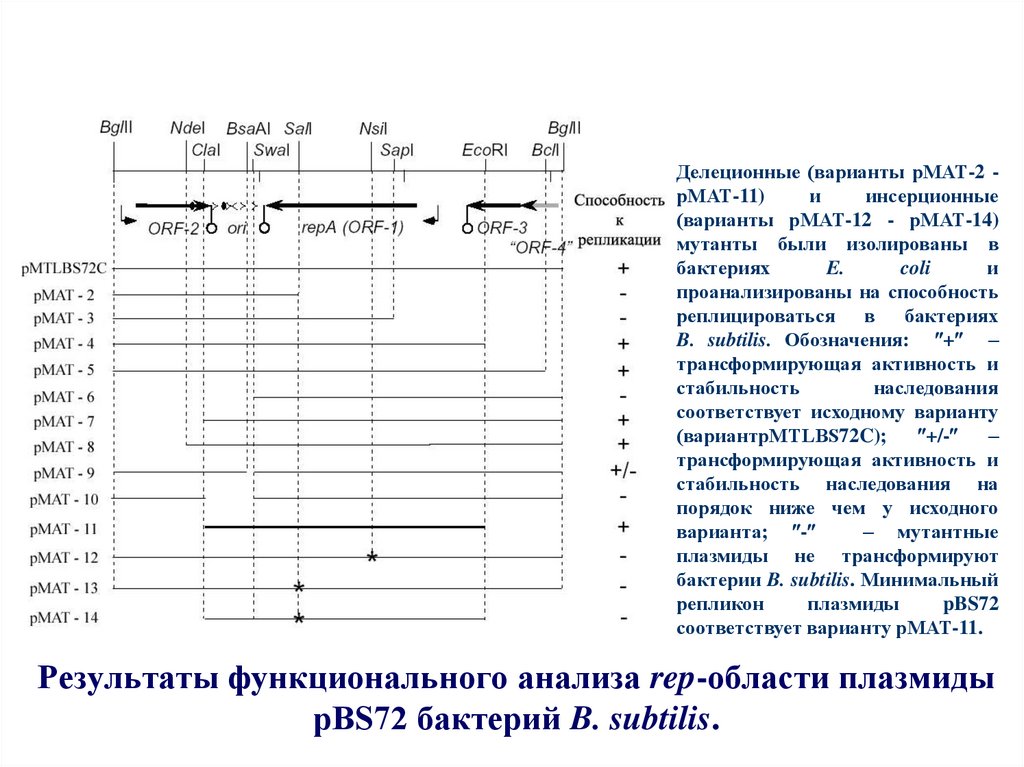
59. Результаты функционального анализа rep-области плазмиды pBS72 бактерий B. subtilis.
Делеционные (варианты рМАТ-2 рМАТ-11)и
инсерционные
(варианты рМАТ-12 - рМАТ-14)
мутанты были изолированы в
бактериях
E.
coli
и
проанализированы на способность
реплицироваться в бактериях
B. subtilis. Обозначения: + –
трансформирующая активность и
стабильность
наследования
соответствует исходному варианту
(вариантpMTLBS72C);
+/-
–
трансформирующая активность и
стабильность наследования на
порядок ниже чем у исходного
варианта; -
– мутантные
плазмиды не трансформируют
бактерии B. subtilis. Минимальный
репликон
плазмиды
pBS72
соответствует варианту рМАТ-11.
Результаты функционального анализа rep-области плазмиды
pBS72 бактерий B. subtilis.
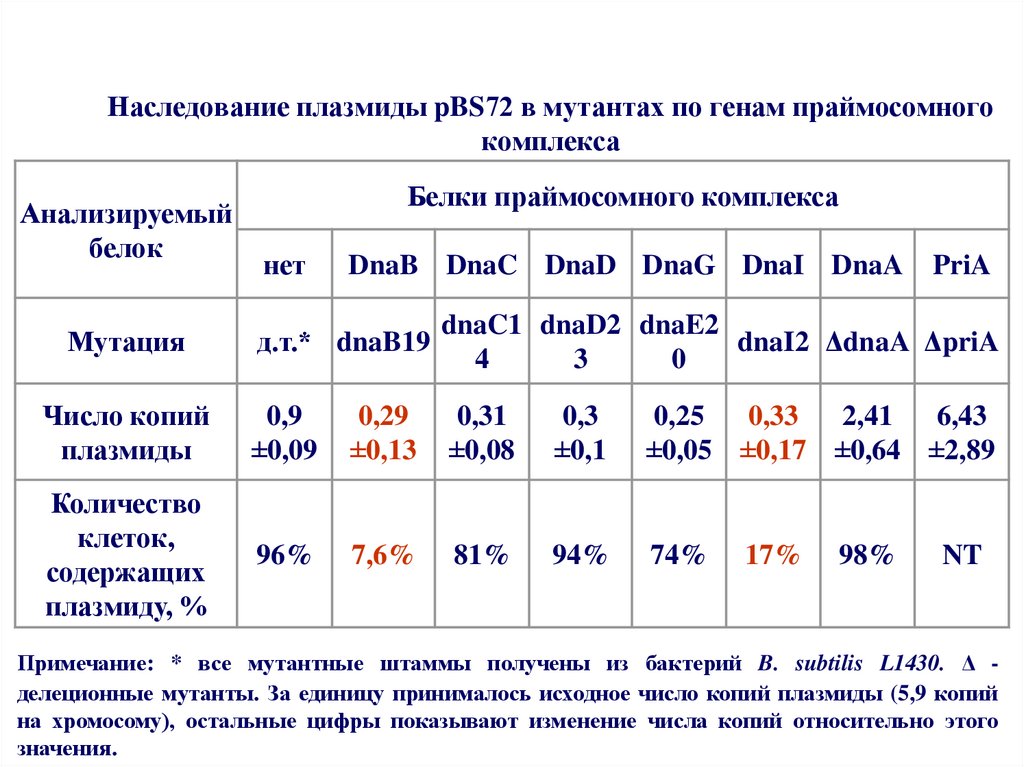
60. Наследование плазмиды pBS72 в мутантах по генам праймосомного комплекса
Анализируемыйбелок
Белки праймосомного комплекса
нет
DnaB DnaC DnaD DnaG DnaI DnaA
PriA
д.т.* dnaB19
dnaC1 dnaD2 dnaE2
dnaI2 ΔdnaA ΔpriA
4
3
0
Число копий
плазмиды
0,9
±0,09
0,29
±0,13
0,31
±0,08
0,3
±0,1
0,25
0,33
2,41
6,43
±0,05 ±0,17 ±0,64 ±2,89
Количество
клеток,
содержащих
плазмиду, %
96%
7,6%
81%
94%
74%
Мутация
17%
98%
NT
Примечание: * все мутантные штаммы получены из бактерий B. subtilis L1430. Δ делеционные мутанты. За единицу принималось исходное число копий плазмиды (5,9 копий
на хромосому), остальные цифры показывают изменение числа копий относительно этого
значения.
61. Векторы для молекулярного клонирования в бактериях B.subtilis
На основании rep-области плазмиды рBS72 были сконструированы двурепликонныевекторные системы, предназначенные для клонирования (pAL2) и экспрессии (pAL1)
генетического материала в клетках бактерий B. subtilis . Созданные конструкции
способны эффективно трансформироваться в клетки B. subtilis и характеризуются
структурной и сегрегационной стабильностью при введении в их состав чужеродных
фрагментов размером до 10 kb.
62.
INTAS97-1464 «Function analysis of genes on plasmids in Bacillus» (1999-2001),INTAS-99-01487 «The Pseudomonas horizontal gene pool: the role of IncP-9 plasmids in the
diversity of this genus» (2000-2003),
3.
INTAS 01-2383 «Characterisation and exploitation of plasmids controlling biodegradation of
naphthalene by Pseudomonas species» (2002-2005).
4.
Проект Минобразования «Молекулярно-генетический анализ систем репликации и
стабильного поддержания плазмид тета-типа грамположительных и грамотрицательных
бактерий (2003-2005).
5.
Задание 17.17 государственной программы «Генетическая инженерия»: «Создать систему
хозяин-вектор для клонирования и экспрессии про- и эукариотических генов в бактериях
Bacillus subtilis» (2003-2006).
6.
Задание 13 государственной программы «Биотехнология» «Выделить из природных
источников штаммы бактерий Bacillus subtilis, наследующие плазмиды различных типов.
Охарактеризовать основные свойства плазмид и создать на их основе векторы для
молекулярного клонирования» (2004-2006).
7. Задание 1.08 «Изучение генетического разнообразия и оценка биодеградативного
потенциала бактерий-деструкторов полициклических ароматических углеводородов,
выделенных из природных источников на территории Беларуси» ГППИ «Новые
биотехнологии» (2006-2010).
8.
Проект Белорусского республиканского фонда фундаментальных исследований «Изучение
плазмид с тета-типом репликации, содержащихся в штаммах Bacillus subtilis, выделенных
из почв Беларуси и России» (2004-2006).
9.
Проект Белорусского республиканского фонда фундаментальных исследований
«Особенности наследования плазмид группы IncP-9 в клетках гомо- и гетерологичных
хозяев» (2006-2008).
10. Проект Белорусского республиканского фонда фундаментальных исследований «Роль
внехромосомных и хромосомных генетических детерминант в микробной деградации
полициклических ароматических углеродов» (2008-2009).
1.
2.
63.
Статьи:1.
Greated A., Titok M.A., Krasowiak R., Fairclough R.J., Thomas C.M. The replication and stable-inheritance
functions of IncP-9 plasmid рM3 // Microbiology. – 2000. – Vol. 146. – P. 2249-2258.
2.
Krasowiak R., Smalla K., Sokolov S., Kosheleva I.A., Sevastianovich Y.R., Titok M.A., Thomas C.M. PCR
primers for detection and characterization of IncP-9 plasmids // FEMS Microbiol. Ecol. – 2002. – Vol. 42, № 2. – P.
217-225.
3.
Titok M.A., Chapuis J., Selezneva Y.V., Lagodich, A.V., Prokulevich V.A., Ehrlich S.D., Janniere L. Bacillus
subtilis soil isolates: plasmid replicon analysis and construction of a new theta-replicating vector // Plasmid. – 2003. –
Vol. 49, № 1. – С. 53-62.
4.
Титок М.А. Использование плазмиды широкого круга хозяев рМ3 (IncP-9) для генетического анализа
бактерий семейства Enterobacteriaceae // Генетика. – 2003. – Т. 39, № 12. – С. 1606-1611.
5.
Лагодич А.В., Штанюк Я.В., Прозоров А.А., Титок М.А. Характеристика систем репликации плазмид
природных штаммов Bacillus subtilis // Мол. Биол. – 2004. – Т. 38, № 3. – с. 1-5.
6.
Левчук А.А., Василенко С.Л., Булыга И.М., Титок М.А. Томас К.М. Характеристика штаммов
нафталинутилизирующих бактерий рода Pseudomonas // Известия РАН– 2005.–– №2.– p.162-167.
7.
Лагодич А.В., Черва Е.А., Штанюк Я. В., Прокулевич В.А., Фомичев Ю.К., Прозоров А.А. Титок М.А.
Создание векторной системы для молекулярного клонирования в клетках Bаcillus subtilis и Escherichia coli //
Мол.биол. – 2005. – Т. 39.–№2. – с.345-348
8.
Sevastsyanovich, Y.R., Titok, M.A., Krasowiak, R., Bingle, L.E.H., Thomas, C.M. (2005). Ability of IncP-9
plasmid pM3 to replicate in Escherichia coli is dependent on both rep and par functions. Mol. Microbiol., 57 (3), 819833.
9.
Titok M., Suski C., Dalmais B., Ehrlich S. D., Jannie L.The replicative polymerases PolC and DnaE
arerequired for theta replication of the Bacillus subtilis plasmid pBS72//Microbiology (2006), 152, 1471–1478
10.
Левчук А.А., Булыга И.М., Измалкова Т.Ю., Севастьянович Я.Р., Кошелева И.А., Thomas C.M., Титок
М.А. Nah-плазмиды группы IncP-9 природных штаммов Pseudomonas// Мол.биол. 2006,Т.40 №5,С. 835–843..
11.
Василенко С. Л. Титок М. А. Особенности наследования плазмид биодеградации в клетках гомо- и
гетерологичных хозяев. // Микробиология.-2008.-Т.77, №1.- с. 16-22.
12.
Sevastsyanovich Y. R., Krasowiak R., Bingle L. E. H., Haines A. S., Sokolov S. L., Kosheleva I. A., Leuchuk A.
A., Titok M. A., Smalla K., Thomas C. M. Diversity of IncP-9 plasmids of Pseudomonas// Microbiology . 2008 V.154,
№10, р. 2929-41.



























 Биология
Биология








