Похожие презентации:
Эволюция ферментов биосинтеза фенилпропаноидов у растений
1. Эволюция ферментов биосинтеза фенилпропаноидов у растений
Автор: Чеснокова Анна Сергеевна, ученица10Б класса МБОУ «Лицей №22 «НадеждаСибири»
Руководители: Дорошков Алексей Владимирович, кандидат биологических наук;
Сильванович Елизавета Константиновна
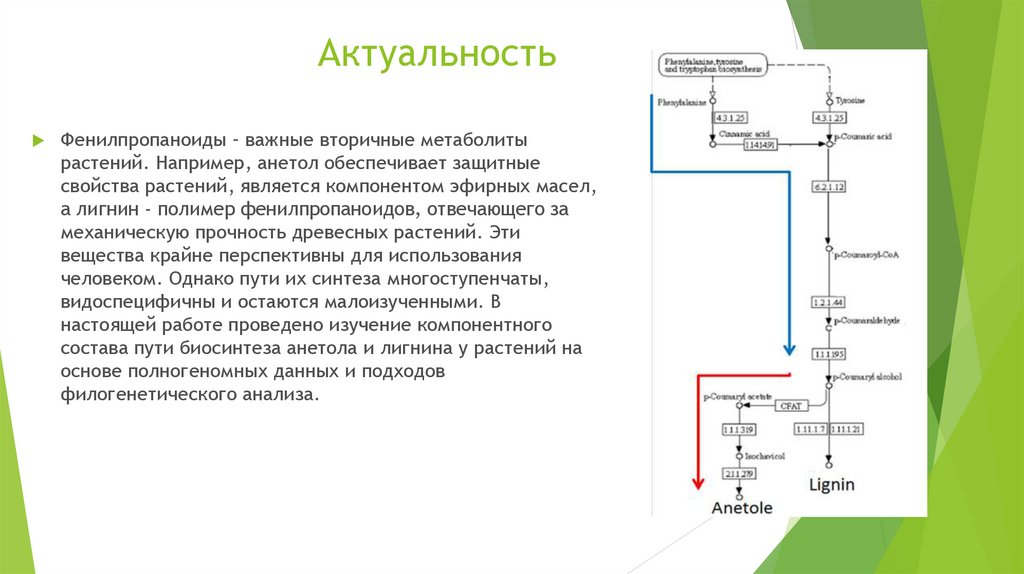
2. Актуальность
Фенилпропаноиды – важные вторичные метаболитырастений. Например, анетол обеспечивает защитные
свойства растений, является компонентом эфирных масел,
а лигнин - полимер фенилпропаноидов, отвечающего за
механическую прочность древесных растений. Эти
вещества крайне перспективны для использования
человеком. Однако пути их синтеза многоступенчаты,
видоспецифичны и остаются малоизученными. В
настоящей работе проведено изучение компонентного
состава пути биосинтеза анетола и лигнина у растений на
основе полногеномных данных и подходов
филогенетического анализа.
3. Цели и задачи
*Цель: провести изучение ферментов пути биосинтеза анетола илигнина у растений на основе полногеномных данных и подходов
филогенетического анализа.
*Задачи:
Изучить путь биосинтеза анетола и лигнина.
Сделать выборку аннотированных белков и их гомологичных
последовательностей.
Построить филогенетические деревья, оценить эволюцию ферментов
и их изменчивость в ортогруппах.
Провести анализ полногеномных данных компонетнов пути
биосинтеза анетола и лигнина и сделать предположение о их
взаимодействии.
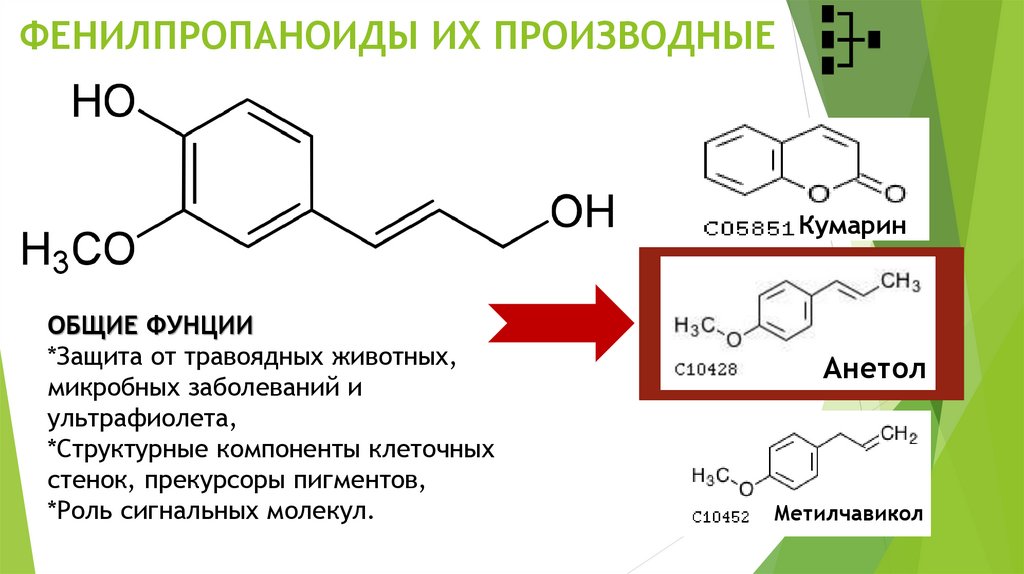
4. ФЕНИЛПРОПАНОИДЫ ИХ ПРОИЗВОДНЫЕ
КумаринОБЩИЕ ФУНЦИИ
*Защита от травоядных животных,
микробных заболеваний и
ультрафиолета,
*Структурные компоненты клеточных
стенок, прекурсоры пигментов,
*Роль сигнальных молекул.
Анетол
Метилчавикол
5. АНЕТОЛ
ПУТЬ БИОСИНТЕЗААНЕТОЛ
Обладает мощными
антимикробными
свойствами
6. МЕТОДЫ
Было произведено извлечение белковыхпоследовательностей и поиск их ортологов.
Далее проведено множественное выравнивание
последовательностей и удаление
неконсервативных участков, также определение
белковых доменов. Из полученных данных
рассчитали филогенетические отношения.
Далее была оценена экспрессии генов на основе
полнотранскриптомных данных.
7.
Выбор пути биосинтеза одного из производных фенилпропаноидов - АНЕТОЛА8.
Поиск последовательности аннотированных белков и ихгомологов
BLAST (алгоритм поиска гомологичных последовательностей)
9.
Выравнивание гомологичных последовательностейMAFFT (средство выравнивания гомологичных последовательностей)
УДАЛЕНИЕ ГЭПОВ
РЕЗУЛЬТАТ: ГОТОВАЯ ВЫРАВНЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ БЕЗ ГЭПОВ
10.
*Деревья строятся по методу максимального правдоподобия*Archaeopteryx: Visualization, Analysis, and Editing of
Phylogenetic Trees
FastTree
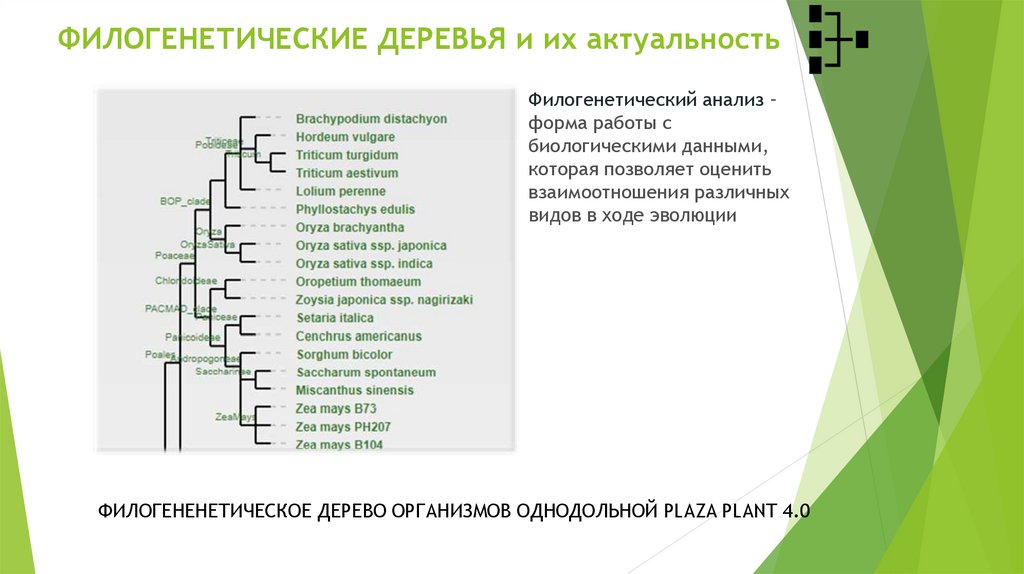
11. ФИЛОГЕНЕТИЧЕСКИЕ ДЕРЕВЬЯ и их актуальность
Филогенетический анализ –форма работы с
биологическими данными,
которая позволяет оценить
взаимоотношения различных
видов в ходе эволюции
ФИЛОГЕНЕНЕТИЧЕСКОЕ ДЕРЕВО ОРГАНИЗМОВ ОДНОДОЛЬНОЙ PLAZA PLANT 4.0
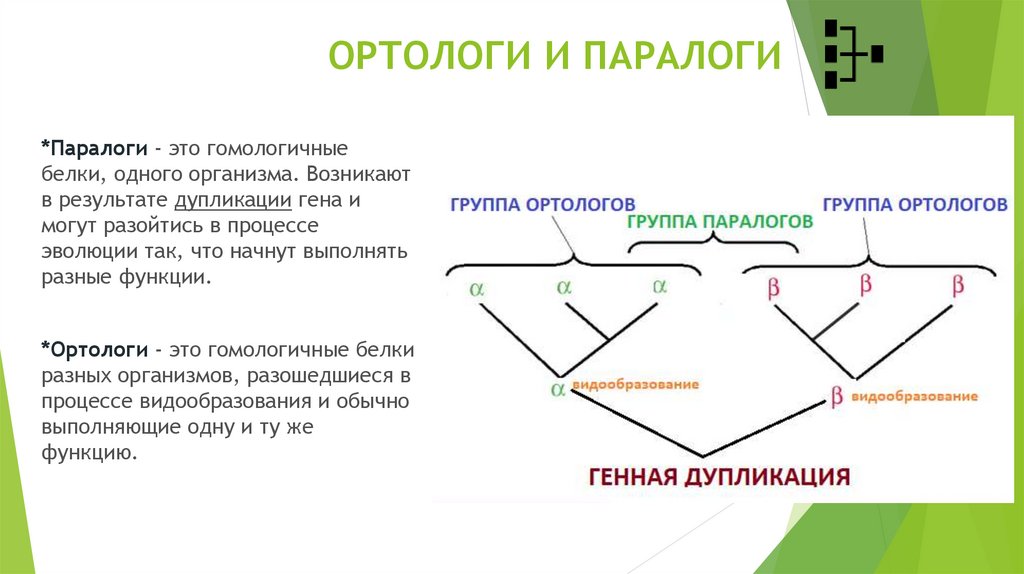
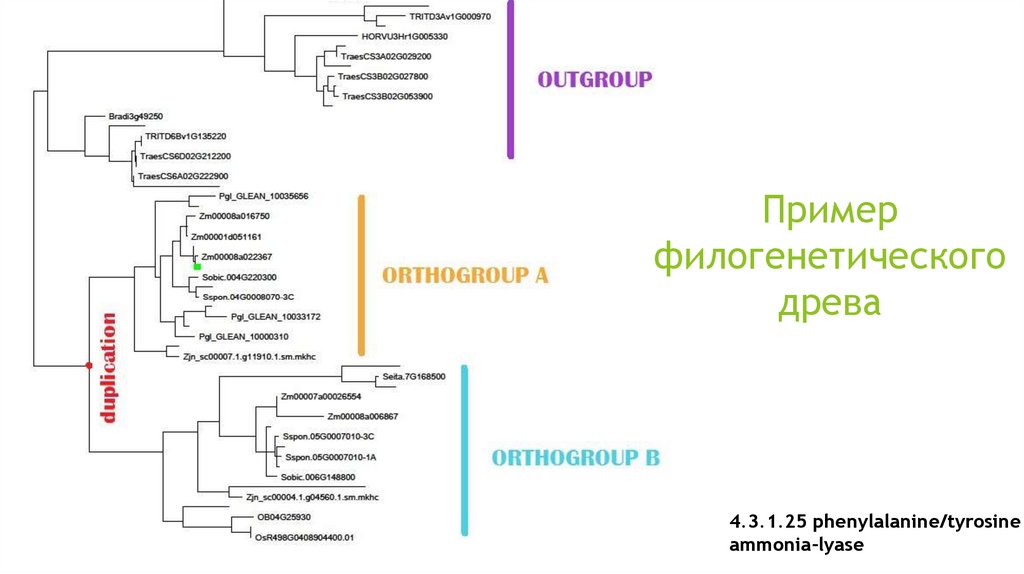
12. ОРТОЛОГИ И ПАРАЛОГИ
*Паралоги - это гомологичныебелки, одного организма. Возникают
в результате дупликации гена и
могут разойтись в процессе
эволюции так, что начнут выполнять
разные функции.
*Ортологи - это гомологичные белки
разных организмов, разошедшиеся в
процессе видообразования и обычно
выполняющие одну и ту же
функцию.
13. Пример филогенетического древа
4.3.1.25 phenylalanine/tyrosineammonia-lyase
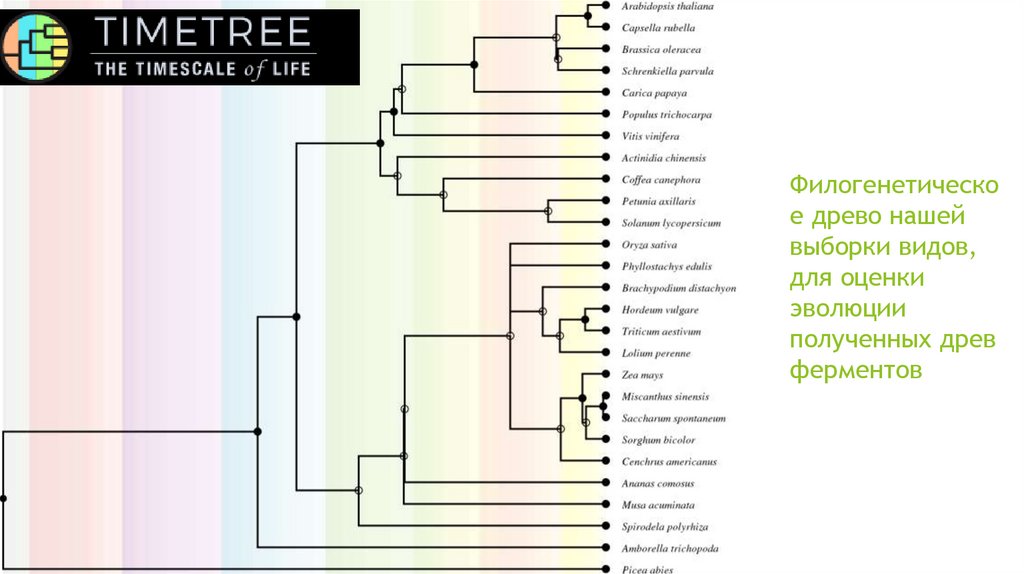
14. Филогенетическое древо нашей выборки видов, для оценки эволюции полученных древ ферментов
Филогенетическое древо нашей
выборки видов,
для оценки
эволюции
полученных древ
ферментов
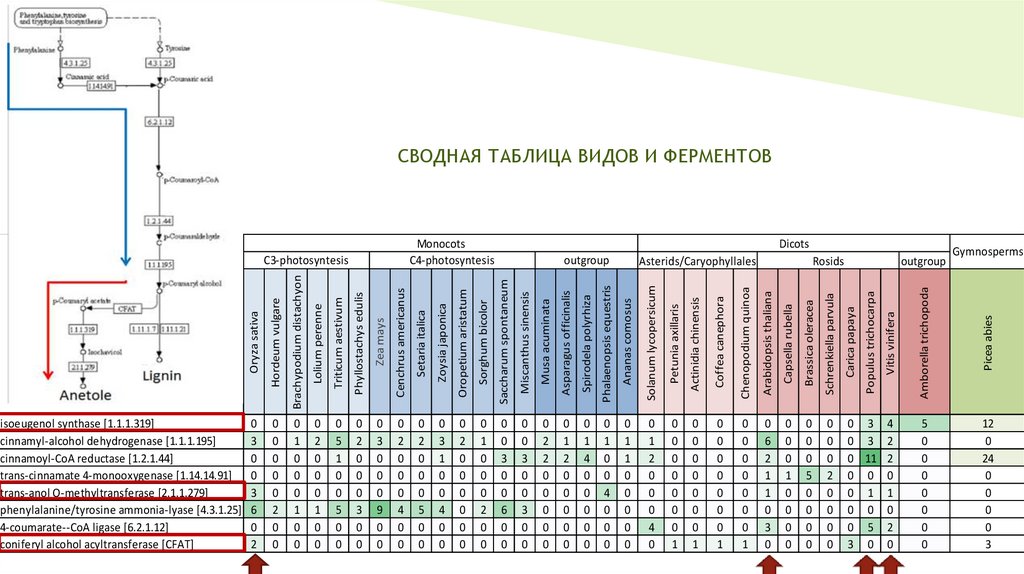
15.
СВОДНАЯ ТАБЛИЦА ВИДОВ И ФЕРМЕНТОВMonocots
С4-photosyntesis
Zea mays
Cenchrus americanus
Setaria italica
Zoysia japonica
Oropetium aristatum
Sorghum bicolor
Saccharum spontaneum
Miscanthus sinensis
Musa acuminata
Asparagus officinalis
Spirodela polyrhiza
Phalaenopsis equestris
Ananas comosus
Solanum lycopersicum
Petunia axillaris
Actinidia chinensis
Coffea canephora
Chenopodium quinoa
Arabidopsis thaliana
Capsella rubella
Brassica oleracea
Schrenkiella parvula
Carica papaya
Vitis vinifera
Amborella trichopoda
Picea abies
0
3
0
0
3
6
0
2
0
0
0
0
0
2
0
0
0
1
0
0
0
1
0
0
0
2
0
0
0
1
0
0
0
5
1
0
0
5
0
0
0
2
0
0
0
3
0
0
0
3
0
0
0
9
0
0
0
2
0
0
0
4
0
0
0
2
0
0
0
5
0
0
0
3
1
0
0
4
0
0
0
2
0
0
0
0
0
0
0
1
0
0
0
2
0
0
0
0
3
0
0
6
0
0
0
0
3
0
0
3
0
0
0
2
2
0
0
0
0
0
0
1
2
0
0
0
0
0
0
1
4
0
0
0
0
0
0
1
0
0
4
0
0
0
0
1
1
0
0
0
0
0
0
1
2
0
0
0
4
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
1
0
6
2
1
1
0
3
0
0
0
0
1
0
0
0
0
0
0
0
5
0
0
0
0
0
0
0
2
0
0
0
0
0 3 4
0 3 2
0 11 2
0 0 0
0 1 1
0 0 0
0 5 2
3 0 0
5
0
0
0
0
0
0
0
12
0
24
0
0
0
0
3
Populus trichocarpa
Phyllostachys edulis
Gymnosperms
Triticum aestivum
outgroup
Lolium perenne
Rosids
Brachypodium distachyon
Asterids/Caryophyllales
Hordeum vulgare
isoeugenol synthase [1.1.1.319]
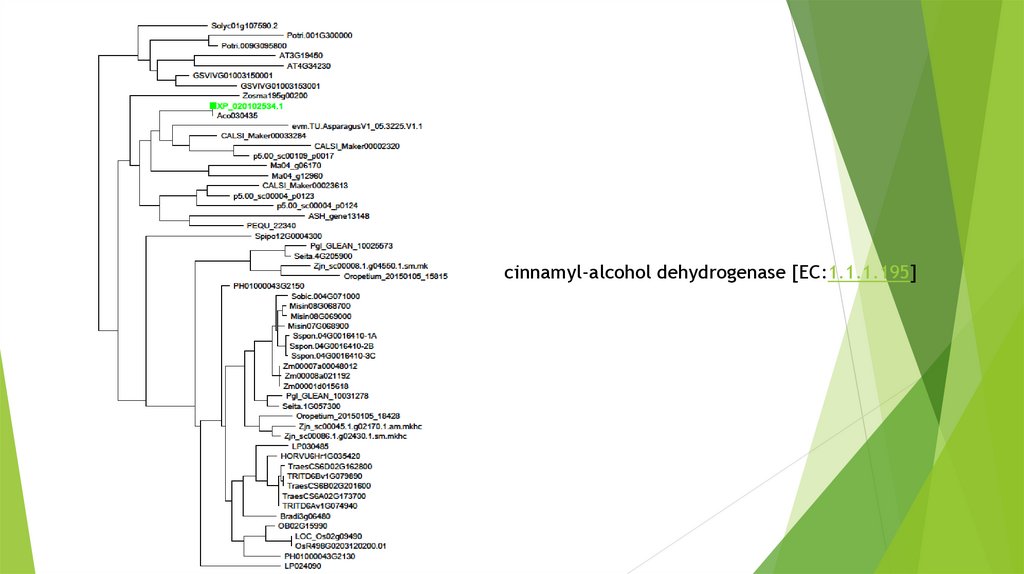
cinnamyl-alcohol dehydrogenase [1.1.1.195]
cinnamoyl-CoA reductase [1.2.1.44]
trans-cinnamate 4-monooxygenase [1.14.14.91]
trans-anol O-methyltransferase [2.1.1.279]
phenylalanine/tyrosine ammonia-lyase [4.3.1.25]
4-coumarate--CoA ligase [6.2.1.12]
coniferyl alcohol acyltransferase [CFAT]
outgroup
Oryza sativa
С3-photosyntesis
Dicots
16.
МЕСТА ЛОКАЛИЗАИЙ ФЕРМЕНТОВ И ЭКСПРЕССИИ ГЕНАМесто экспрессии гена
phenylalanine/tyrosine ammonia-lyase [4.3.1.25]
Корень
trans-cinnamate 4-monooxygenase [1.14.14.91]
Корень
4-coumarate--CoA ligase [6.2.1.12]
Корень
cinnamoyl-CoA reductase [1.2.1.44]
Корень
cinnamyl-alcohol dehydrogenase [1.1.1.195]
Ксилема
coniferyl alcohol acyltransferase [CFAT]
Лист
expression atlas
isoeugenol synthase [1.1.1.319]
Плод
trans-anol O-methyltransferase [2.1.1.279]
Ксилема/корень
Еxpression atlas
Локализация фермента
Корень
Корень
Корень
Корень
Ксилема
Лист
Лист/плод
Лист/плод
17. Результаты
Построено 8 филогенетических деревьев по ферментам пути биосинтезаанетола. Базальная топология соответствует современной систематике
организмов. Все белки были найдены как у двудольных и однодольных
растений, у голосеменных. Выделены ортогруппы аннотированных белков.
Проведён анализ всех выделенных ортогрупп по ортологам и паралогам, а
также по видовому разнообразию.
Было найдено 222 последовательности различных видов.
Создано дерево видов, просчитано количество последовательностей для
каждого белка и сделана сводная таблица.
Проведён анализ эволюции белков.
18. Выводы
По данным таблицы, полный комплект белков пути биосинтеза анетола неимеет ни один вид. Наибольшее количество белков имеют виды: Oruza
sativa, Arabidopsis thaliana, Populus trichocarpa, Vitis vinifera.
У некоторых видов часть белков отсутствует в большом количестве.
Предположительная причина – субстрат-специфичные ферменты, которые
характерны только для этого пути, и при их отсутствии вещество не
синтезируется, но также эти белки могут находиться в организме для
других процессов.
У видов, у которых отсутствует небольшое количество белков - возможно,
вещество синтезируется, но белки не вошли в ортогрпуппу, так как
накопили достаточное количество замен чтобы отличаться от изначально
аннотированного белка.
Белки быстро накапливают замены и не являются консервативными.
У древесных растений (Picea abies, Populus trichocarpa) в большей мере
синтезируется анетол, полагаясь на его свойства, можно предположить,
что антимикробные свойства сильнее, чем у растений без синтеза лигнина.
19. Источники и литература
http://bioinformatics.ru/downloads/pdf/bioinfo9_09.pdfhttps://bmcevolbiol.biomedcentral.com/articles/10.1186/s12862-017-0891-5
http://www.plantphysiol.org/content/149/1/384
https://febs.onlinelibrary.wiley.com/doi/epdf/10.1111/j.14321033.1979.tb13138.x
https://doi.org/10.1111/j.1432-1033.1984.tb08002.x
https://febs.onlinelibrary.wiley.com/doi/full/10.1111/j.14321033.1974.tb03359.x
https://doi.org/10.1016/0005-2744(73)90174-5
https://www.sciencedirect.com/science/article/pii/S1074552106003875?via%3D
ihub
20. Эволюция ферментов биосинтеза фенилпропаноидов у растений
Авторы: Гандзюк Валентина Юрьевна, Чеснокова Анна Сергеевна, ученицы 10Бкласса МБОУ «Лицей №22 «Надежда Сибири»
Руководитель: Дорошков Алексей Владимирович, кандидат биологических наук,
старший научный сотрудник ИЦиГ СО РАН




















 Биология
Биология








