Похожие презентации:
Методы изучения регуляторных районов генов
1.
Методы изучения регуляторныхрайонов генов
Лекция III
Меркулова Татьяна Ивановна
Институт цитологии и генетики СО РАН
2.
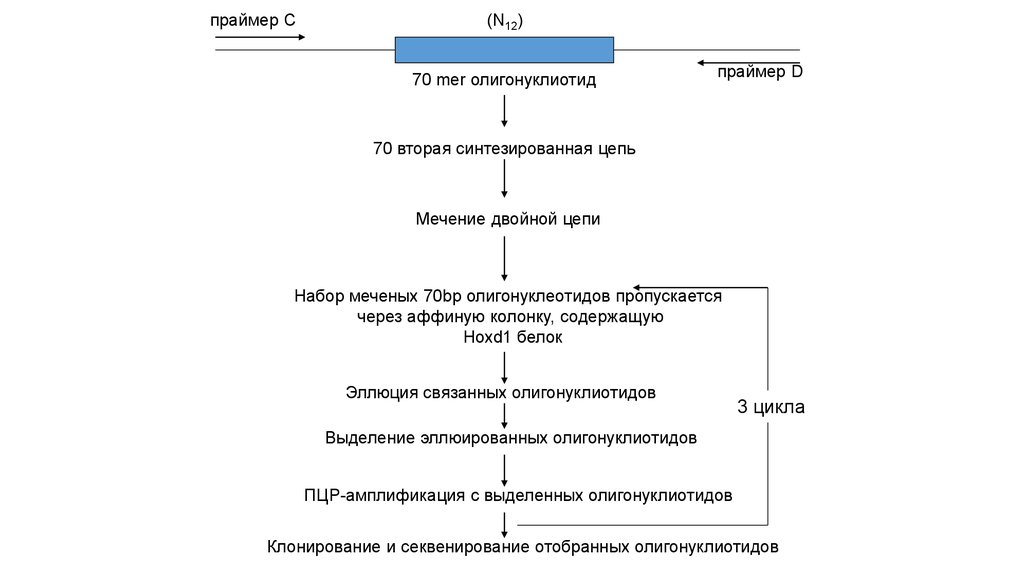
праймер C(N12)
70 mer олигонуклиотид
праймер D
70 вторая синтезированная цепь
Мечение двойной цепи
Набор меченых 70bp олигонуклеотидов пропускается
через аффиную колонку, содержащую
Hoxd1 белок
Эллюция связанных олигонуклиотидов
3 цикла
Выделение эллюированных олигонуклиотидов
ПЦР-амплификация с выделенных олигонуклиотидов
Клонирование и секвенирование отобранных олигонуклиотидов
3.
4.
5.
6.
7.
8.
0Н
0
К
25
МТдт
К
МТ27
В
МТдт
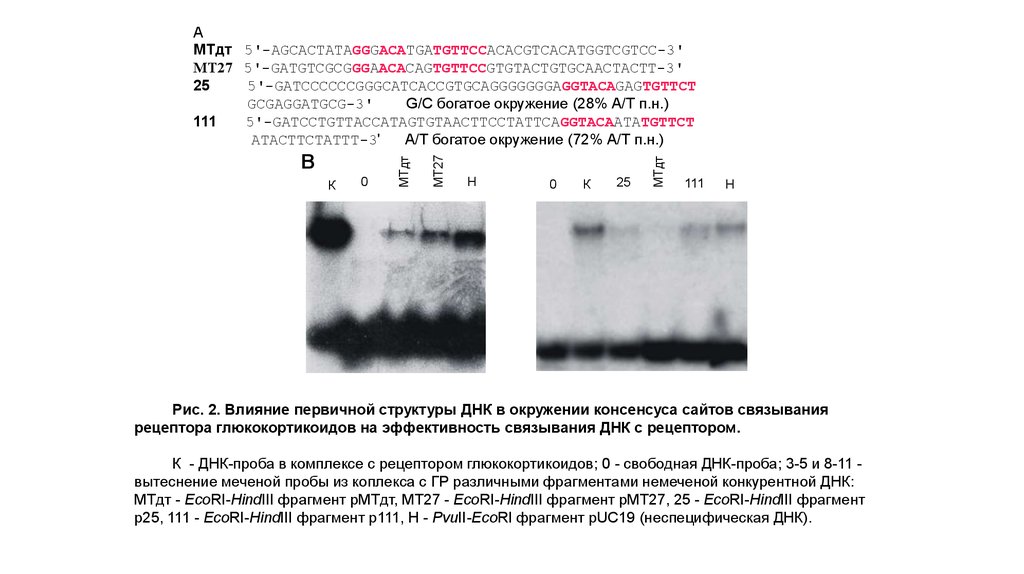
A
МТдт 5'-AGCACTATAGGGACATGATGTTCCACACGTCACATGGTCGTCC-3'
МТ27 5'-GATGTCGCGGGAACACAGTGTTCCGTGTACTGTGCAACTACTT-3'
25
5'-GATCCCCCCGGGCATCACCGTGCAGGGGGGGAGGTACAGAGTGTTCT
GCGAGGATGCG-3'
G/C богатое окружение (28% А/Т п.н.)
111
5'-GATCCTGTTACCATAGTGTAACTTCCTATTCAGGTACAATATGTTCT
ATACTTCTATTT-3'
А/Т богатое окружение (72% А/Т п.н.)
111
Н
Рис. 2. Влияние первичной структуры ДНК в окружении консенсуса сайтов связывания
рецептора глюкокортикоидов на эффективность связывания ДНК с рецептором.
К - ДНК-проба в комплексе с рецептором глюкокортикоидов; 0 - свободная ДНК-проба; 3-5 и 8-11 вытеснение меченой пробы из коплекса с ГР различными фрагментами немеченой конкурентной ДНК:
МТдт - EcoRI-HindIII фрагмент pMTдт, МТ27 - EcoRI-HindIII фрагмент pMT27, 25 - EcoRI-HindIII фрагмент
p25, 111 - EcoRI-HindIII фрагмент p111, Н - PvuII-EcoRI фрагмент рUC19 (неспецифическая ДНК).
9.
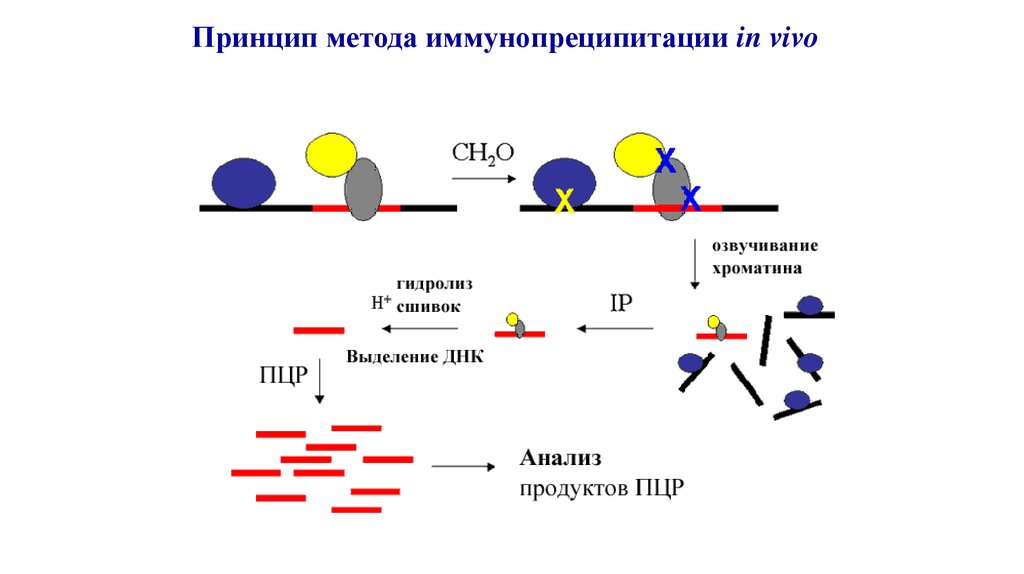
Принцип метода иммунопреципитации in vivo10.
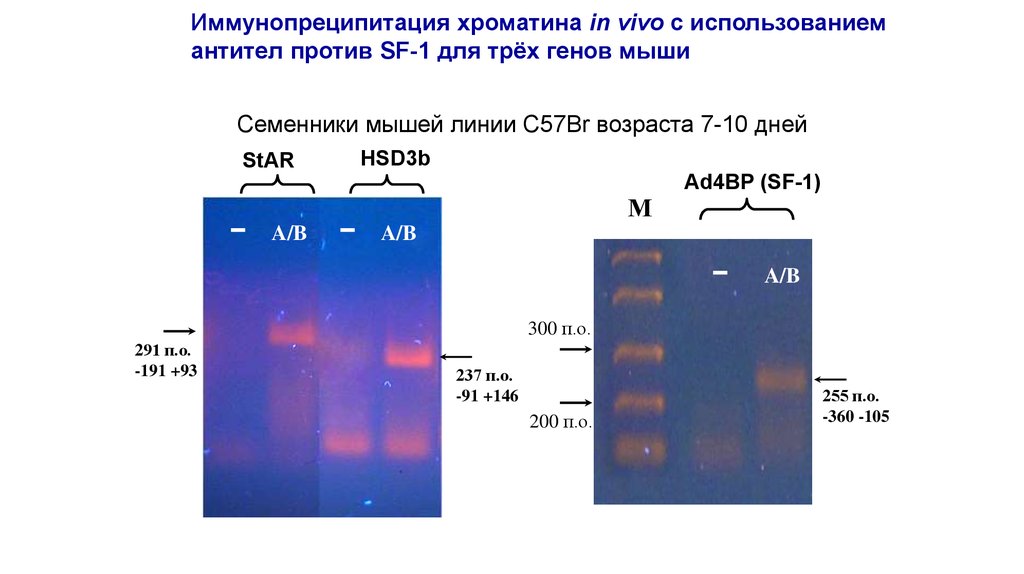
Иммунопреципитация хроматина in vivo с использованиемантител против SF-1 для трёх генов мыши
Семенники мышей линии C57Br возраста 7-10 дней
HSD3b
StAR
Ad4BP (SF-1)
-
A/B
-
М
A/B
-
A/B
300 п.о.
291 п.о.
-191 +93
237 п.о.
-91 +146
200 п.о.
255 п.о.
-360 -105
11.
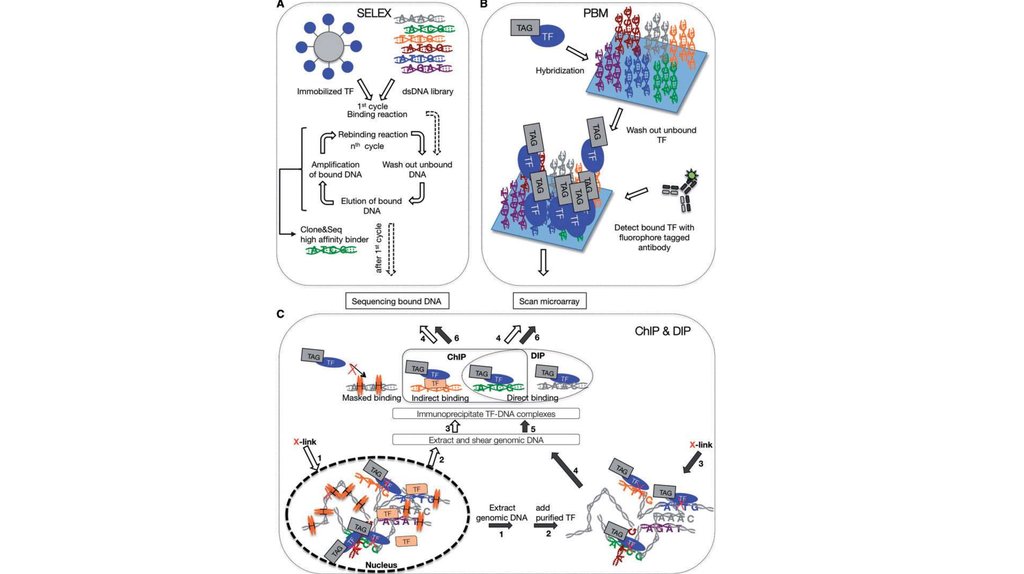
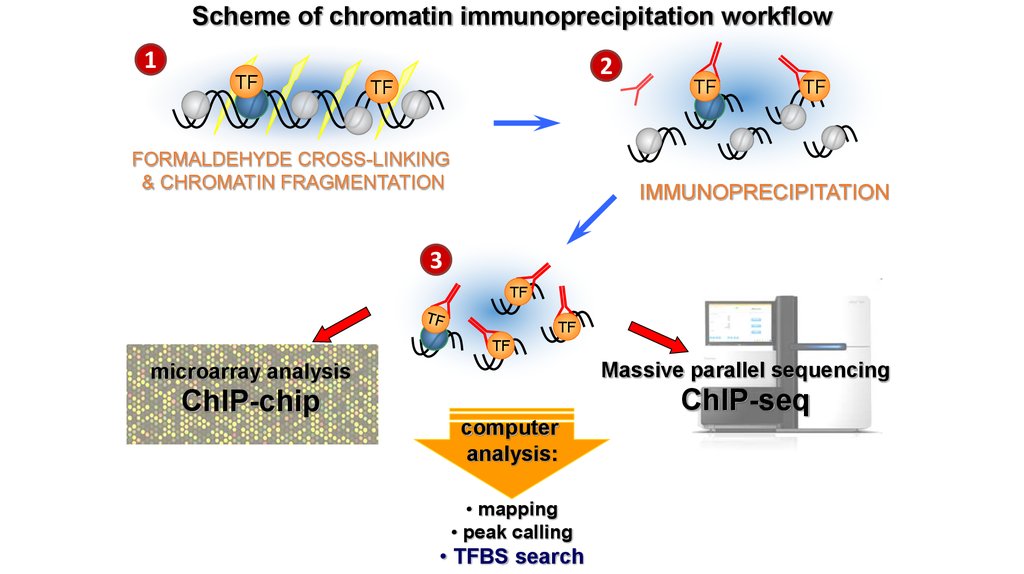
Scheme of chromatin immunoprecipitation workflow1
TF
2
TF
FORMALDEHYDE CROSS-LINKING
& CHROMATIN FRAGMENTATION
TF
TF
IMMUNOPRECIPITATION
3
TF
TF
TF
microarray analysis
Massive parallel sequencing
ChIP-chip
ChIP-seq
computer
analysis:
• mapping
• peak calling
• TFBS search
12.
Features of p65-binding sites on chromosome 22(A) p65 binding relative to Sangerannotated genes and hybridizing
regions on chromosome 22 is
illustrated.
(B) Distribution of NF-B consensus
sequences in p65-binding sites.
The sequences of p65-bound
fragments on the microarray were
searched for NF-B consensus sites
by using both an in-house
chromosome annotation system
and the TFSEARCH database.
www.cbrc.jpresearchdbTFSEARCH.html
R. Martone, G. Euskirchen, P. Bertone, S. Hartman et al. Distribution of NF-B-binding sites across human chromosome 22
PNAS, October 14, 2003, vol. 100 no. 21, 12247–12252
13.
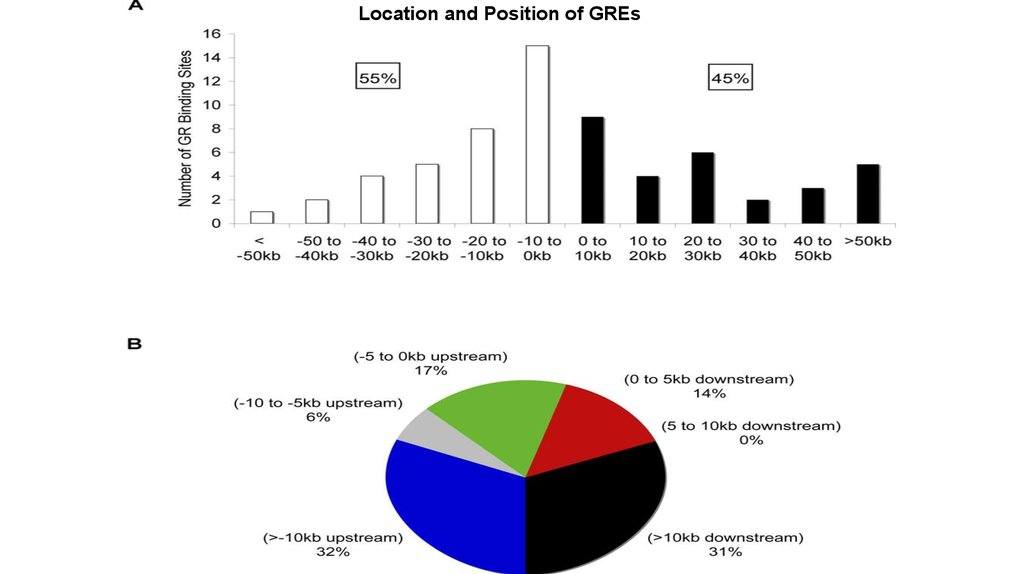
Location and Position of GREs14.
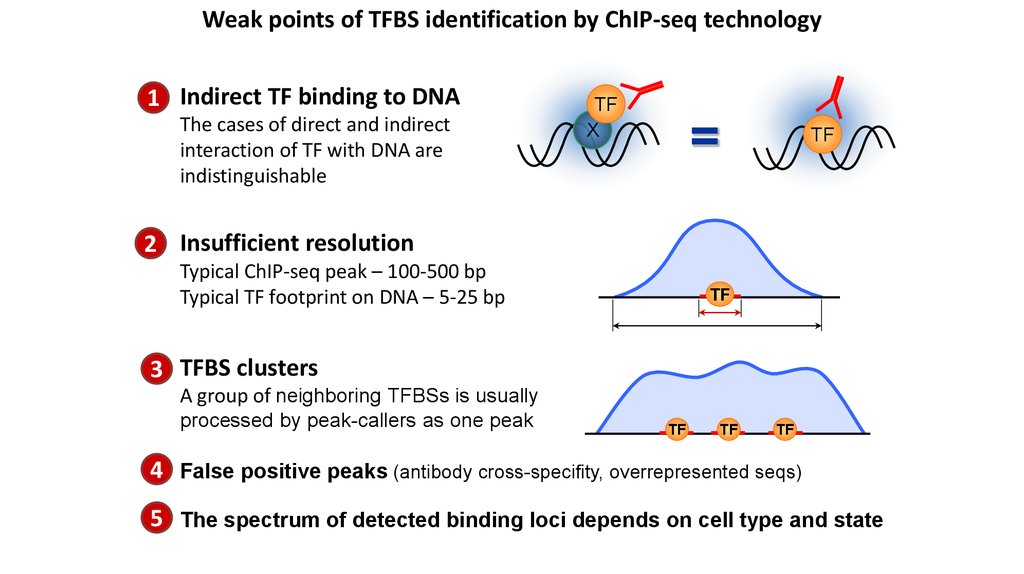
Weak points of TFBS identification by ChIP-seq technology1 Indirect TF binding to DNA
The cases of direct and indirect
interaction of TF with DNA are
indistinguishable
TF
X
=
TF
2 Insufficient resolution
Typical ChIP-seq peak – 100-500 bp
Typical TF footprint on DNA – 5-25 bp
TF
3 TFBS clusters
A group of neighboring TFBSs is usually
processed by peak-callers as one peak
TF
TF
TF
4 False positive peaks (antibody cross-specifity, overrepresented seqs)
5 The spectrum of detected binding loci depends on cell type and state
15.
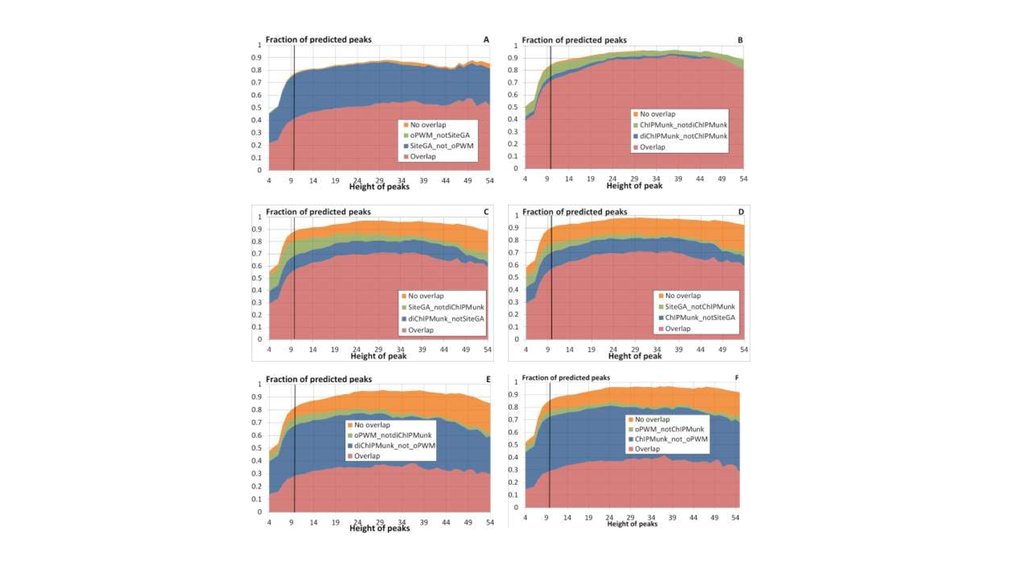
Используемые для интерпретации данных СhIP-seqбиоинформационные методы
Методы предсказания ССТФ
Методы, основанные на
использовании обучающих выборок
ССТФ
De novo методы поиска ССТФ
• методы поиска наиболее
представленных мотивов в
участках связывания ТФ с
хроматином (пиках СhIP-seq).
(без предварительной информации
о ССТФ)
monoCHiPMunk
Kulakovskiy I.V., et al.,
Bioinformatics 2010, 26(20):2622-3
diCHiPMunk
• Консенсус, весовые матрицы (PWM)
и пр.
FoxA oPWM
SiteGA
[Levitsky V.G., et al., BMC Bioinformatics 2007, 8:481]
[Levitsky V.G., et al., Dokl Biochem Biophys. 2011, 436:12-5]
16.
Объект исследования: cайты связывания транскрипционныхфакторов FoxA
ТФ FoxA : FoxA1, FoxA2, FoxA3
ТА
NLS
FKH
NLS
ТА
FoxAs имеют высококонсервативный ДСД типа wingedhelix (FKH).
FoxA обладают способностью вытеснять линкерные
гистоны и открывать компактизованный хроматин, т.е.
могут служить как pioneer transcription factors.
FoxAs играют важную роль в раннем эмбриогенезе,
органогенезе и поддержании метаболического гомеостаза
организма.
Все три ТФ FoxA остаются активными в печени
взрослого, играя активную роль в регуляции метаболизма
Консенсус FoxA :
5'-RYMAAYA-3'
[Overdier et al., 1994; Roux et al., 1995]
FoxA2 ChIP-seq в печени мыши [Wederell et al., 2008]
Число пиков– 11 475
Средняя высота пика– 16
Средняя длина пика– 727 bp
•Wederell E.D. et al. Nucleic Acids Res. 2008 36: 4549–4564
17.
PWM и SiteGAДля создания обучающей выборки из 81 известных ССТФ FoxA
использовали данные из базы TRRD и литературных источников.
Критерием для отбора сайтов служило наличие доказательств
взаимодействия FoxA с соответствующим районом ДНК, полученных
одним из следующих методов:
- футпринтинг ДНКазой I с использованием очищенного белка;
-
EMSA с использованием очищенного белка;
EMSA с использованием ядерного экстракта белков и
специфических антител (EMSA supershift).
Выравнивание последовательностей проводилось относительно
наиболее представленного мотива (в кодировке IUPAC) [Levitsky et al.,
2011]. В итоге, для построения методов PWM и SiteGA использовалось
выравнивание 53 последовательностей, содержащих мотив TRTTTRYH
(R=A/G, Y=C/T, H=A/C/G).
18.
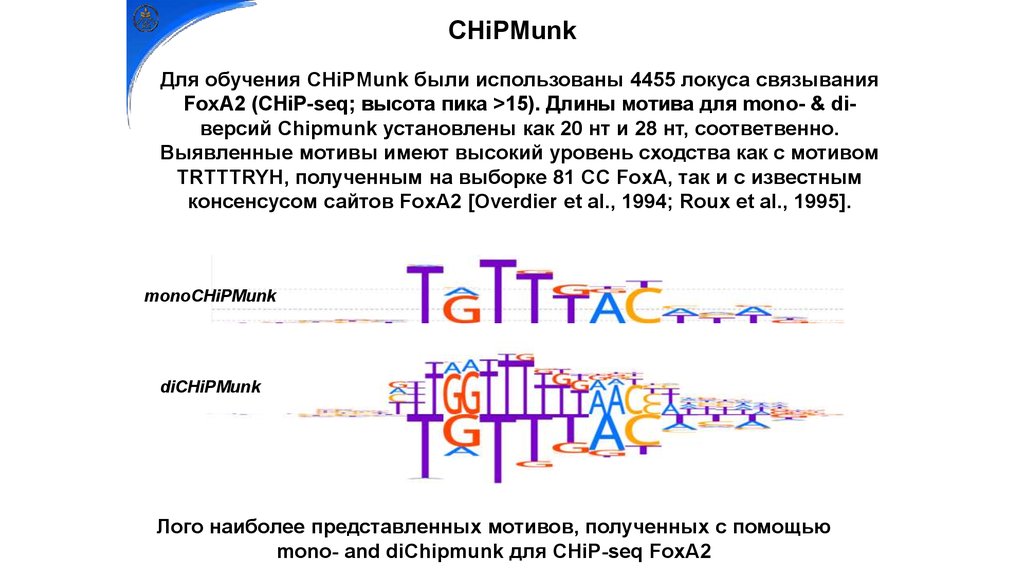
CHiPMunkДля обучения CHiPMunk были использованы 4455 локуса связывания
FoxA2 (CHiP-seq; высота пика >15). Длины мотива для mono- & diверсий Chipmunk установлены как 20 нт и 28 нт, соответвенно.
Выявленные мотивы имеют высокий уровень сходства как с мотивом
TRTTTRYH, полученным на выборке 81 СС FoxA, так и с известным
консенсусом сайтов FoxA2 [Overdier et al., 1994; Roux et al., 1995].
monoCHiPMunk
diCHiPMunk
Лого наиболее представленных мотивов, полученных с помощью
mono- and diChipmunk для CHiP-seq FoxA2
19.
20.
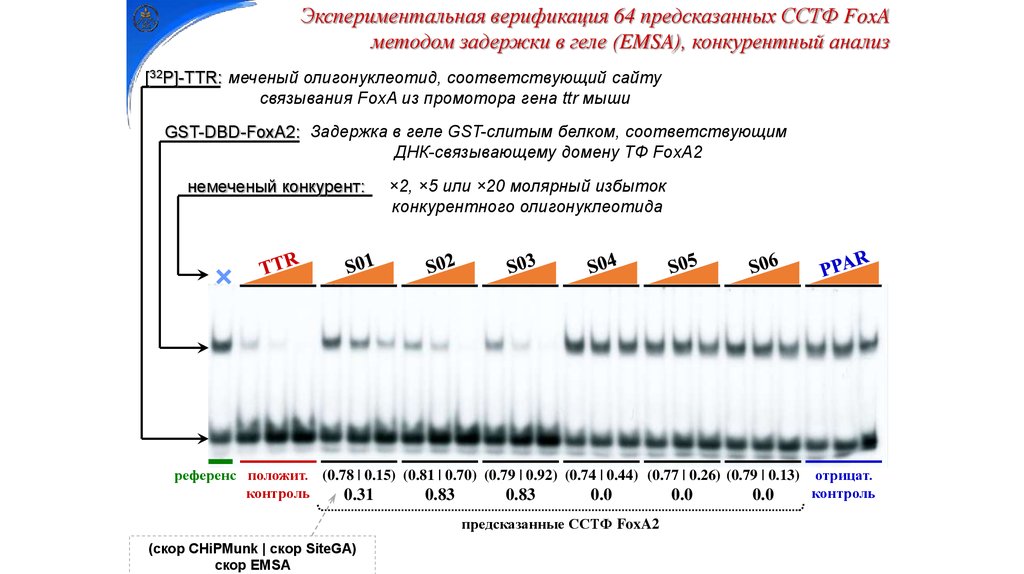
Экспериментальная верификация 64 предсказанных ССТФ FoxAметодом задержки в геле (EMSA), конкурентный анализ
[32P]-TTR: меченый олигонуклеотид, соответствующий сайту
связывания FoxA из промотора гена ttr мыши
GST-DBD-FoxA2: Задержка в геле GST-слитым белком, соответствующим
ДНК-связывающему домену ТФ FoxA2
немеченый конкурент:
×2, ×5 или ×20 молярный избыток
конкурентного олигонуклеотида
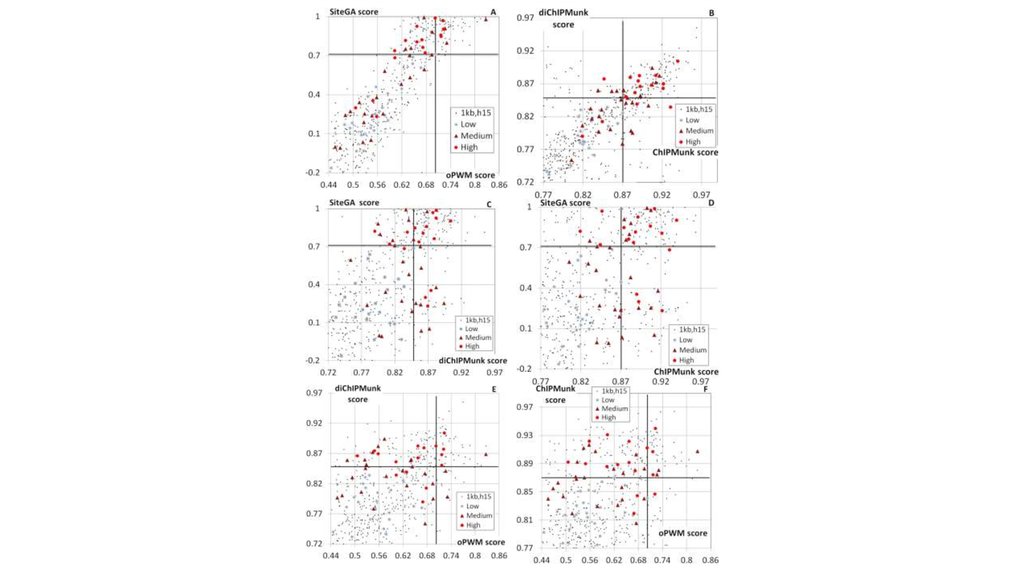
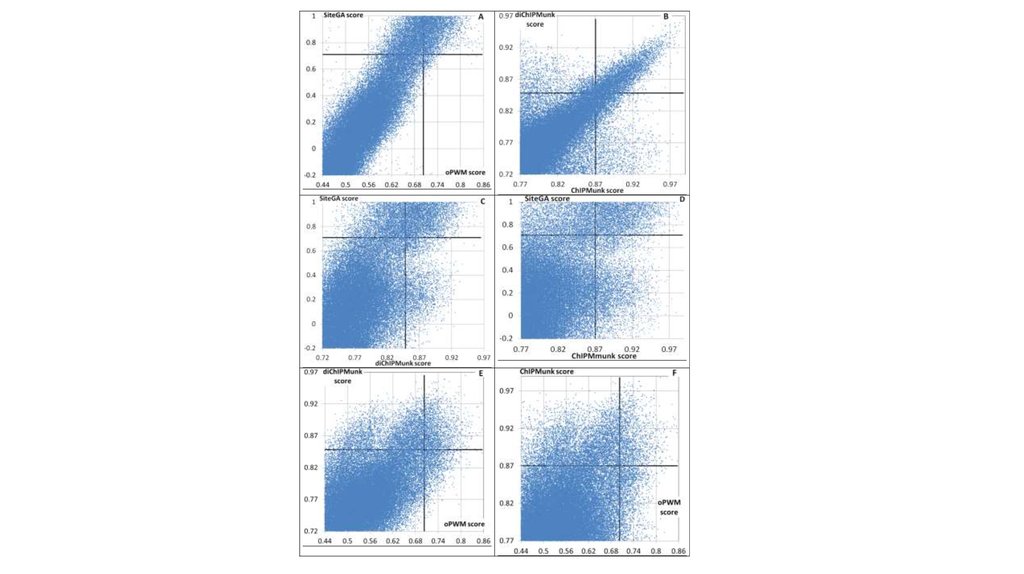
референс положит. (0.78 | 0.15) (0.81 | 0.70) (0.79 | 0.92) (0.74 | 0.44) (0.77 | 0.26) (0.79 | 0.13) отрицат.
контроль
контроль
0.31
0.83
0.83
0.0
0.0
0.0
предсказанные ССТФ FoxA2
(скор CHiPMunk | скор SiteGA)
cкор EMSA
21.
Wdr45 (-359)Mon1a (-245)
H1_015780
F4_018742
G2_026179
F5_138681
Aldh6a1 (-356)
C9_019742
E4_001037937
C7_010747
F1_010821
S03_183028
S02_175329
F2_197986
B5_001045528
E1_001039533
Slc22a7 (-316)
Vwa2 (-538)
S08_181849
S18_001003949
E3_133661_2
A4_183270
B4_009483
H2_197986
S16_011787
Apob (-117)
B2_001042655
B3_001080969
Upp2 (-343)
D2_011255
C5_021319
F3_030703
S09_023750
C1_008558
G1_030016
C4_029562
Sec16b (-184)
C8_178380
S01_029562
D3_011281
C3_144549
S12_172739
A3_023403
E2_177756_2
E3_133661
A5_212442
Cib4 (-110)
A7_026238
D1_001038703
E2_177756
B1_033314
S07_172503
S17_173010
S11_010172
E5_134042
S13_011281
S10_013465
Ahsg (-99)
S15_008558
S14_011994
C6_026417
S06_018742
S04_013743
S05_011787
C2_023887
A6_010172
A2_010189
A1_001101467
скор EMSA
1.5
1.25
1
0.75
0.5
подтвержденные сайты
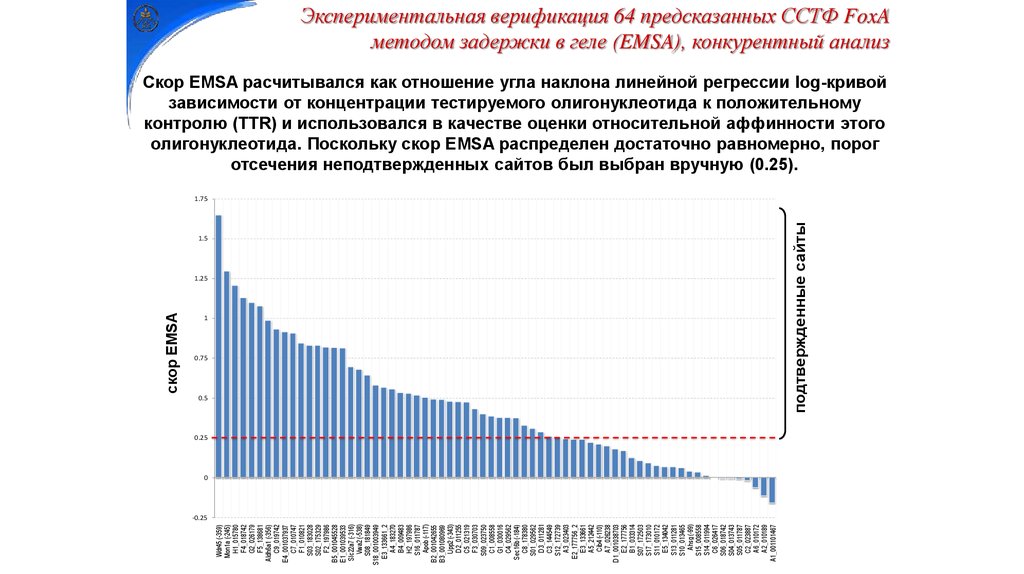
Экспериментальная верификация 64 предсказанных ССТФ FoxA
методом задержки в геле (EMSA), конкурентный анализ
Скор EMSA расчитывался как отношение угла наклона линейной регрессии log-кривой
зависимости от концентрации тестируемого олигонуклеотида к положительному
контролю (TTR) и использовался в качестве оценки относительной аффинности этого
олигонуклеотида. Поскольку скор EMSA распределен достаточно равномерно, порог
отсечения неподтвержденных сайтов был выбран вручную (0.25).
1.75
0.25
0
-0.25
22.
23.
24.
Figure 7. Context patterns of the SF-1 BSs recognized by sequence analysis of the top-scoringChIP-seq peaks (peak height of 15 or higher). The frequency matrices were visualized by the
WebLogo tool (Crooks et al., 2004) for the samples of sites predicted by oPWM, Sitecon, and
SiteGA. The X axis shows nucleotide positions and Y axis, bits.







 Биология
Биология








