Похожие презентации:
Биотехнология растений. Трансгенные растения (часть 4)
1.
Биотехнология растений2.
Лекция 6: Трансгенные растения-41. Получение инсерционных мутантов растений на основе
агробактериальной трансформации
a)
Клонирование генов
b)
Изучение с помощью трансгенных растений
экспрессии генов и структуры растительных промоторов
c)
Получение растений со сверхэкспрессией гена с целью
изучения функции гена
d)
Использование методов «генетической хирургии» для
изучения взаимодействия клеток и тканей в процессах
развития
e)
Использование трансформации для изучения роли
гормонов в развитиии растений.
3.
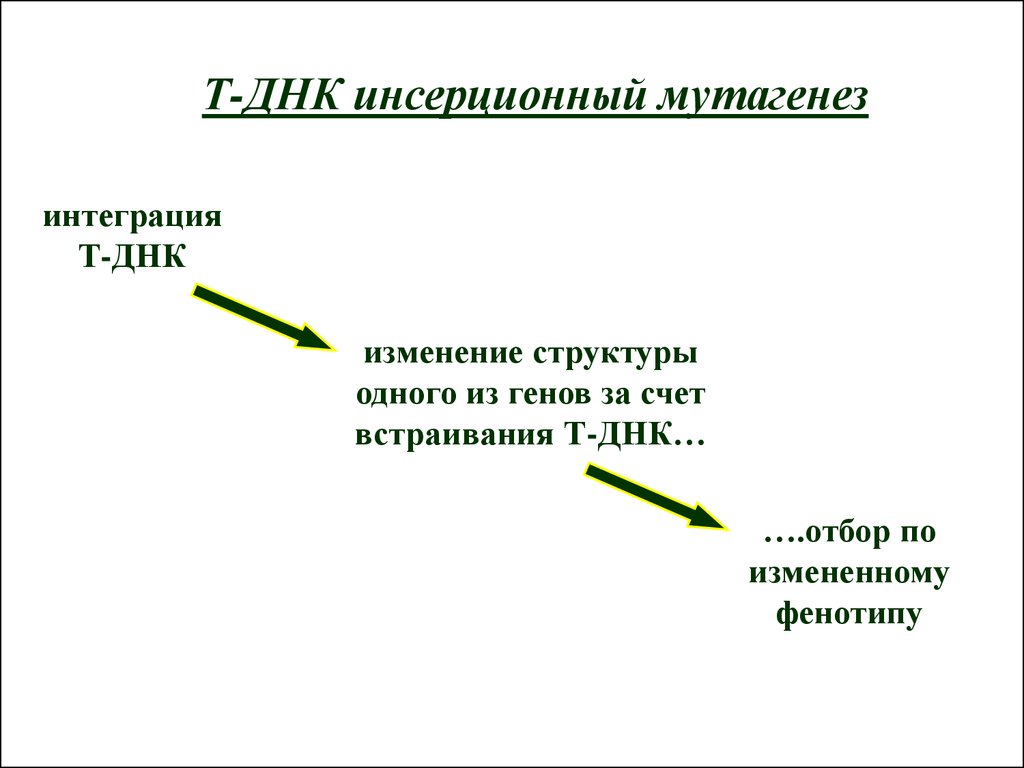
Т-ДНК инсерционный мутагенезинтеграция
Т-ДНК
изменение структуры
одного из генов за счет
встраивания Т-ДНК…
….отбор по
измененному
фенотипу
4.
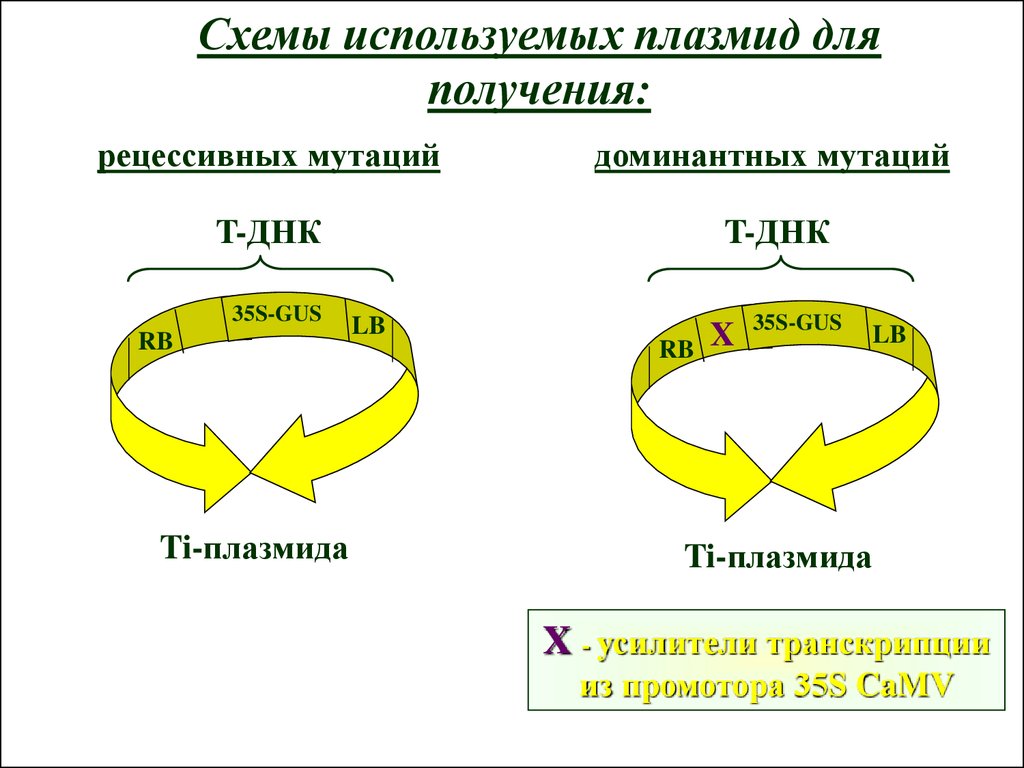
Схемы используемых плазмид дляполучения:
рецессивных мутаций
доминантных мутаций
T-ДНК
T-ДНК
35S-GUS
RB
Ti-плазмида
LB
RB
X
35S-GUS
LB
Ti-плазмида
X - усилители транскрипции
из промотора 35S CaMV
5.
Клонирование генов6.
Клонирование гена agamous с помощьюТ-ДНК инсерционного мутагенеза
NPTII
pBR322
ЭМСмутагенез
мутант
ag-1
RB
LB
wt
10 лепестков,
4 чашелистика
агробактериальная
трансформация семян
отбор KmR
растений с
фенотипом
agamous
клонирование
последовательности в
E. coli
мутант
ag-2
pBR322
AmpR
T-ДНК дизрупированный ген AG
S
LB
S
RB S
RB
трансформированное
растение с цветками
дикого типа
трансформация
мутанта ag-2
7.
Ген, маркированный транспозоном (taggedgene), можно клонировать с помощью
«вытягивания за транспозон»
8.
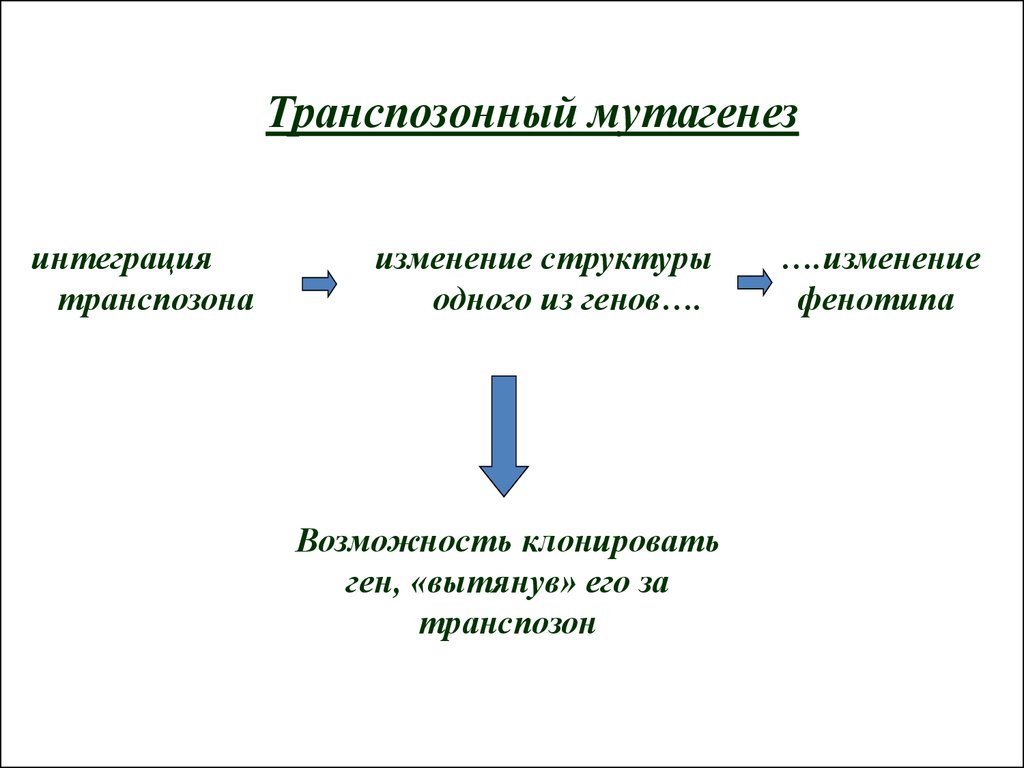
Транспозонный мутагенезинтеграция
транспозона
изменение структуры
одного из генов….
Возможность клонировать
ген, «вытянув» его за
транспозон
….изменение
фенотипа
9.
При встраивании транспозона вкодирующую последовательность гена,
наблюдается мутантный фенотип.
При вырезании фенотип
восстанавливается
10.
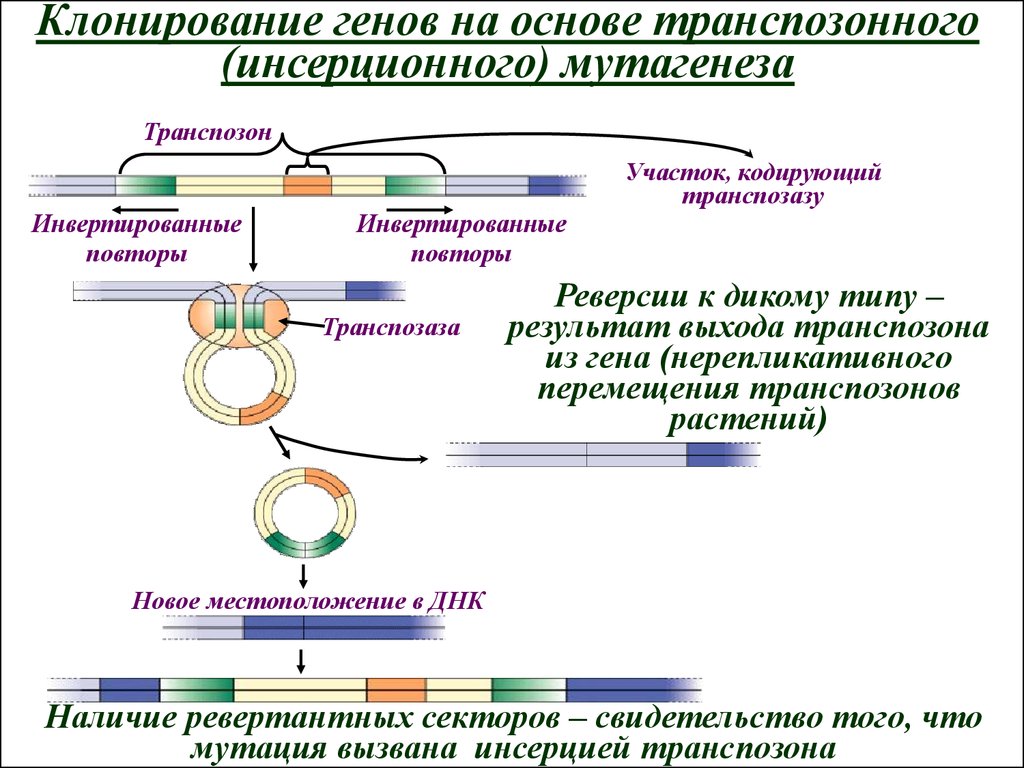
Клонирование генов на основе транспозонного(инсерционного) мутагенеза
Транспозон
Участок, кодирующий
транспозазу
Инвертированные
повторы
Инвертированные
повторы
Транспозаза
Реверсии к дикому типу –
результат выхода транспозона
из гена (нерепликативного
перемещения транспозонов
растений)
Новое местоположение в ДНК
Наличие ревертантных секторов – свидетельство того, что
мутация вызвана инсерцией транспозона
11.
Клонирование генов на основе транспозонного(инсерционного) мутагенеза
Antirrhinum majus (L.)
wild type
floricaula
у мутанта floricaula (flo)
вместо цветков
развиваются побеги
12.
Получение мутации flo613 и клонирование генаFLORICAULA львиного зева
(Coen et al., 1990)
1. Выращивание 26 000 растений львиного зева с активным
мобильным элементом Tam3 (Transposone of Antirrhinum
majus) при tº=15ºC, вызывающей наибольшую частоту
транспозиций (растения М1).
2. Самоопыление растений М1 с целью выявления
рецессивных мутаций, возникших в М1 в результате
перемещения Tam3.
3. Выявление 15 разных гомеозисных мутаций среди 80 000
растений М2, в том числе и flo613.
4. Укоренение и вегетативное размножение веточек мутанта
flo613 при tº=15ºC, для выявления реверсий. Генетический
анализ.
13.
• Рестрикционный анализ ДНК из выделенныхклонов, необходимый для выявления участков
растительной ДНК, фланкирующих транспозон
• Субклонирование растительной ДНК с целью
дальнейшего использования в качестве пробы
• Создание геномной библиотеки из растений
дикого типа
• Скрининг библиотеки с меченой пробой
растительной ДНК, фланкирующей транспозон,
для выявления клона, содержащего ген FLO
• Доказательство того, что выявленные клоны
содержат ген FLO
14.
Создание геномной библиотеки из мутантаflo 613, выращиваемого при tº=25ºC (Тam3 не
перемещается)
Вставка
растительной
ДНК
Ген для цветной
селекции
Изоляция
ДНК
Изоляция
векторной
ДНК
Рестрикция;
Комбинирование
растительной и
векторной ДНК;
Лигирование
Вставка
растительной
ДНК, содержащей
ген интереса
AmpR (ген
устойчивости к
антибиотику)
Рост
бактерии
Трансформация
бактерии
15.
Скрининг библиотеки с меченой пробой из Tam3для выявления клонов, содержащих транспозон
Блот с
нитроцеллюлозным
фильтром
Вектор с инсерцией
растительной ДНК
Вектор пустышка
Реплика
Лизис бактерий,
денатурацияДНК
Экспозиция на
чашке с
селективной средой
Вектор с инсерцией
растительной ДНК,
содержащей ген
интереса
Амплификация
бактерий,
содержащих вектор
с геном интереса
Использованеи
авторадиографии для
локализации
бактериальных колоний,
содержащих ген интереса
Гибридизация с
радиоктивной
меткой для
выяления гена
интереса
Получение отпечатков клонов библиотеки на нитроцеллюлозных фильтрах
Гибридизация фильтров с меченой пробой (чаще – кДНК меченая Р32)
Авторадиография
16.
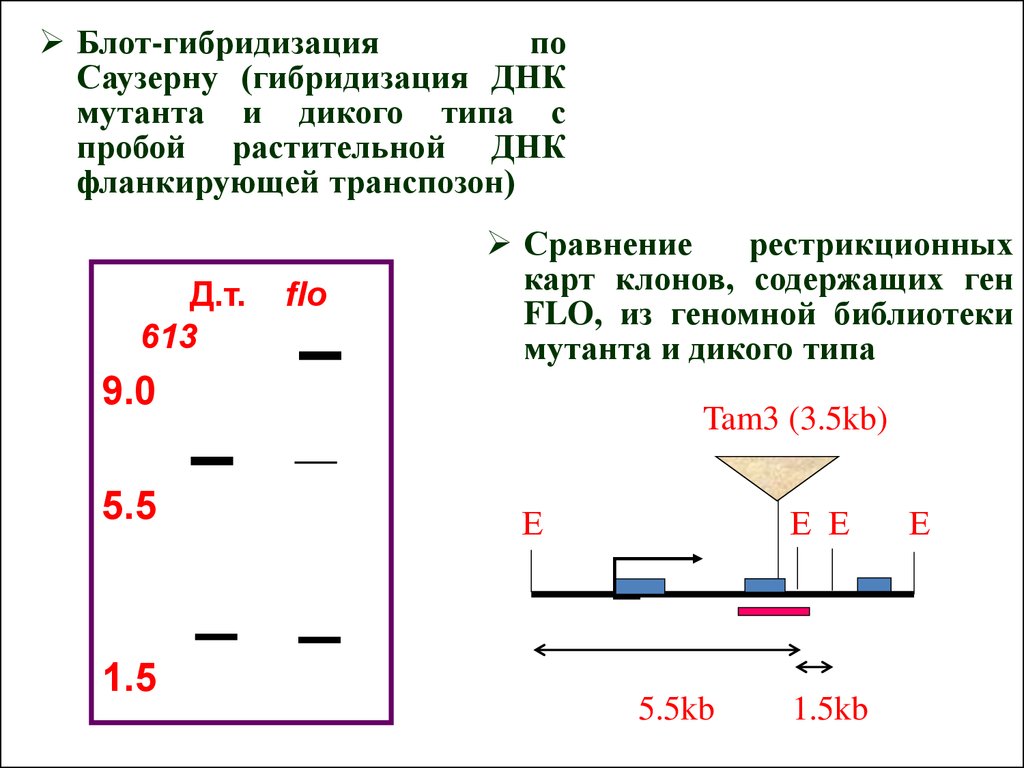
Блот-гибридизацияпо
Саузерну (гибридизация ДНК
мутанта и дикого типа с
пробой растительной ДНК
фланкирующей транспозон)
Д.т.
613
flo
Сравнение
рестрикционных
карт клонов, содержащих ген
FLO, из геномной библиотеки
мутанта и дикого типа
9.0
5.5
1.5
Tam3 (3.5kb)
Е
Е Е
5.5kb
1.5kb
Е
17.
Ген FLORICAULA – уникальный ген,присутствующий в геноме покрытосеменных в
1 копии. Он кодирует белок, содержащий
домены, характерные для белков – активаторов
транскрипции (транскрипционных факторов):
богатый пролином домен на N-конце и кислую
область в середине белка.
Наиболее активная экспрессия гена
FLORICAULA наблюдается в закладывающихся примордиях цветка.
Клонирование гена открывает возможность для
изучения роли кодируемого им белка и мРНК
во взаимодействии клеток
18.
Изучение механизмов регуляцииэкспрессии генов растений с использованием
репортерных (индикаторных) генов
Возможности метода:
1) Выявление факторов (внешних и внутренних),
определяющих
особенности
экспрессии
исследуемого гена
2) Выявление цис-регуляторных элементов гена,
отвечающих на действие внешних и внутренних
факторов
19.
К промотору исследуемого гена присоединяютрепортерный ген
ATG
ТАТА
интрон
ATG
Растительный
ген
Бактериальный
ген-репортер
Слитые гены-репортеры
ATG-растения
ТАТА
Химерный белок
20.
Promoter-reporter fusion:Использование гибридных конструкций, содержащих
репортерный ген, «слитый» с промотором изучаемого гена
Green fluorescent protein (GFP)
- флуоресцентный белок - стабильный,
- позволяет определить локализацию продукта на
клеточном, тканевом уровне
- не дает точной количественной оценки экспрессии
(полуколичественный метод)
pSCR::GFP
ß-glucuronidase (GUS)
- широко используется для изучения активности генов, т.к. позволяет
выявить слабый уровень экспрессии
-позволяет определить локализацию продукта на клеточном,
тканевом уровне,
белок стабильный, накапливается в тканях
- не дает точной количественной оценки экспрессии
(полуколичественный метод)
Luciferase (LUC)
-позволяет определить локализацию продукта на тканевом
уровне,
- дает наиболее точную количественную оценку
- может использоваться для изучения динамических
процессов (белок короткоживущий)
pCO::GUS
21.
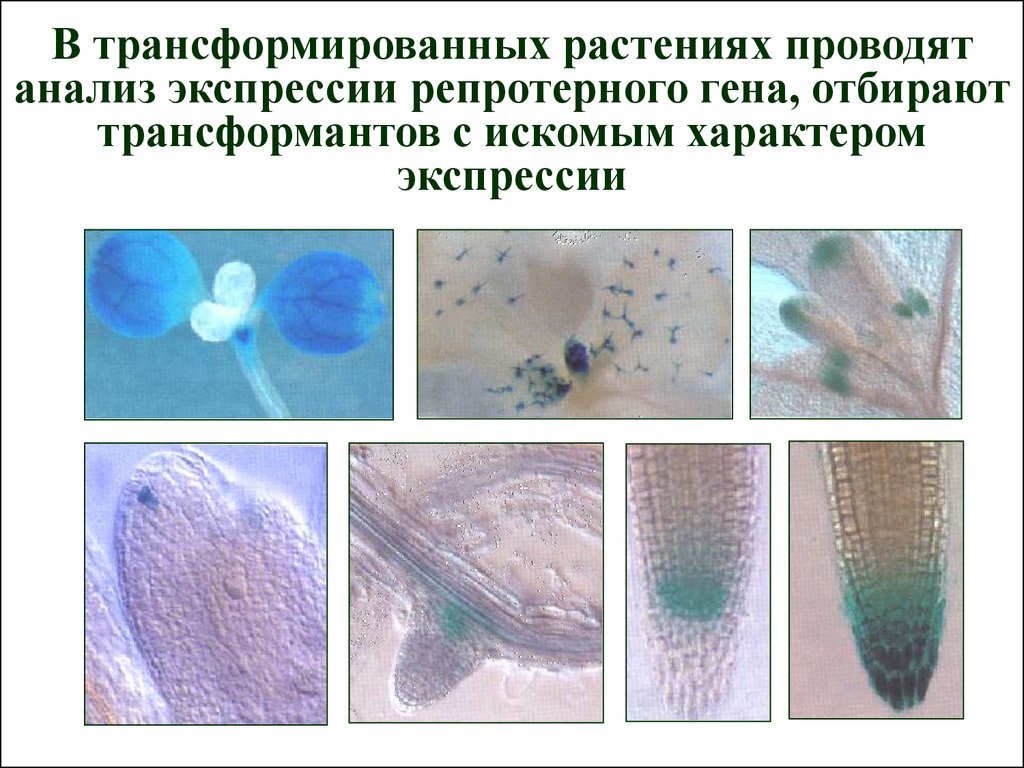
В трансформированных растениях проводятанализ экспрессии репротерного гена, отбирают
трансформантов с искомым характером
экспрессии
22.
Анализ распределение ауксина в растениях с помощьюгенетической конструкции DR5:GUS
DR5 – синтетический ауксин-регулируемый промотор
23.
Анализ активности промоторовключевых регуляторных генов в
меристемах растений
24.
Получение растений со сверхэкспрессиейгена с целью изучения функции гена
25.
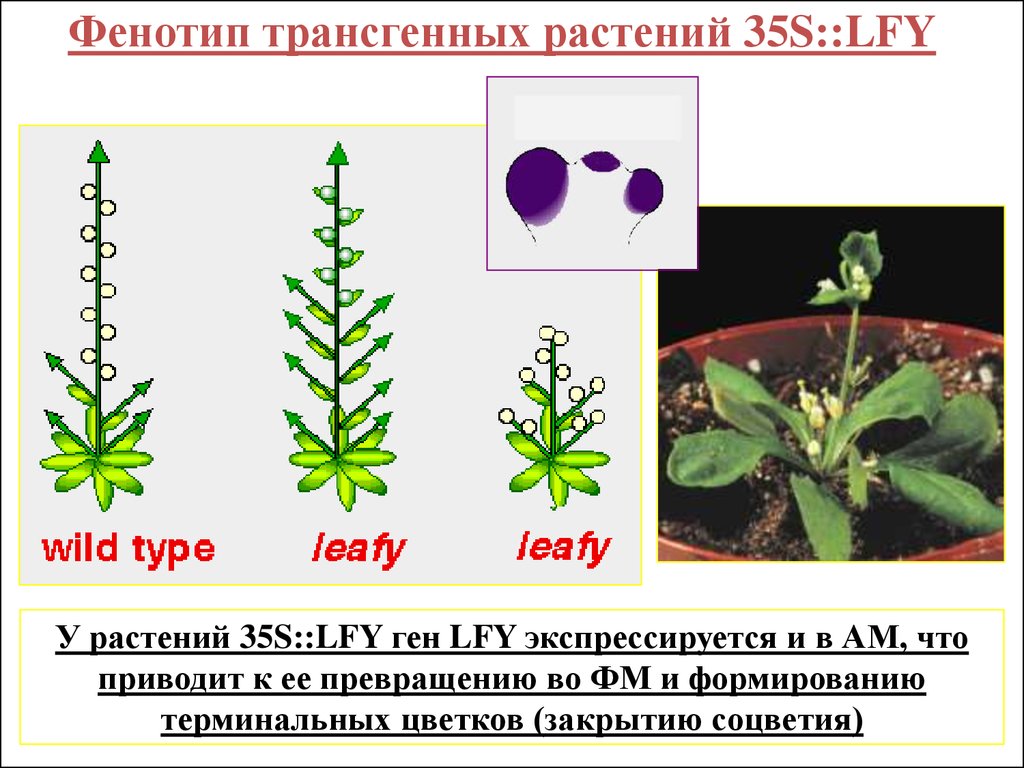
Фенотип трансгенных растений 35S::LFYУ растений 35S::LFY ген LFY экспрессируется и в АМ, что
приводит к ее превращению во ФМ и формированию
терминальных цветков (закрытию соцветия)
26.
Характер экспрессии генов-ортологов LFY/FLO можетопределять тип соцветия (открытое или закрытое)
открытое
закрытое
LFY
Antirrhinum Arabidopsis
табак
томат
27.
Метод позволяет изучить регуляторнуюфункцию любого участка гена
Направленный мутагенез промоторной области гена LFY
позволил обнаружить цис-элемент CAACTGTC, делеция
которого полностью прекращала транскрипцию репортера
A
A GFP:LFY
стоп
ATG
GFP:LFYΔ1
GFP:LFYΔ2
GFP:LFYΔ3
B
ATG
P X
S H
B
ATG
X
S H
B
ATG
P X
B
P X
стоп
ML1 :: YVCVMP : GFP
S H
стоп
GFP
B
стоп
C
ML1::GFP:LFYΔ1 ML1::GFP:LFYΔ2
S H
LFY
D
ML1::GFP:LFYΔ3
28.
Мутант арабидопсиса по гену LFYlfy-26
29.
Основные семейства транскрипционныхфакторов растений
Консервативные для
эукариот:
Уникальные для растений:
ТФ с MADS-доменом
ТФ GRAS
ТФ с гомеодоменом
ТФ AP2-like
ТФ MYB
ТФ с В3-доменом
ТФ bZIP
ТФ с GARP/ARRM-доменом
etc.
etc.
MYB
29
30.
30Гомеодомен-ДНК - комплекс
Структура транскрипционного фактора с ДНК-связывающим гомеодоменом
N
вариабельный уч.
гомеодомен
С
30
31.
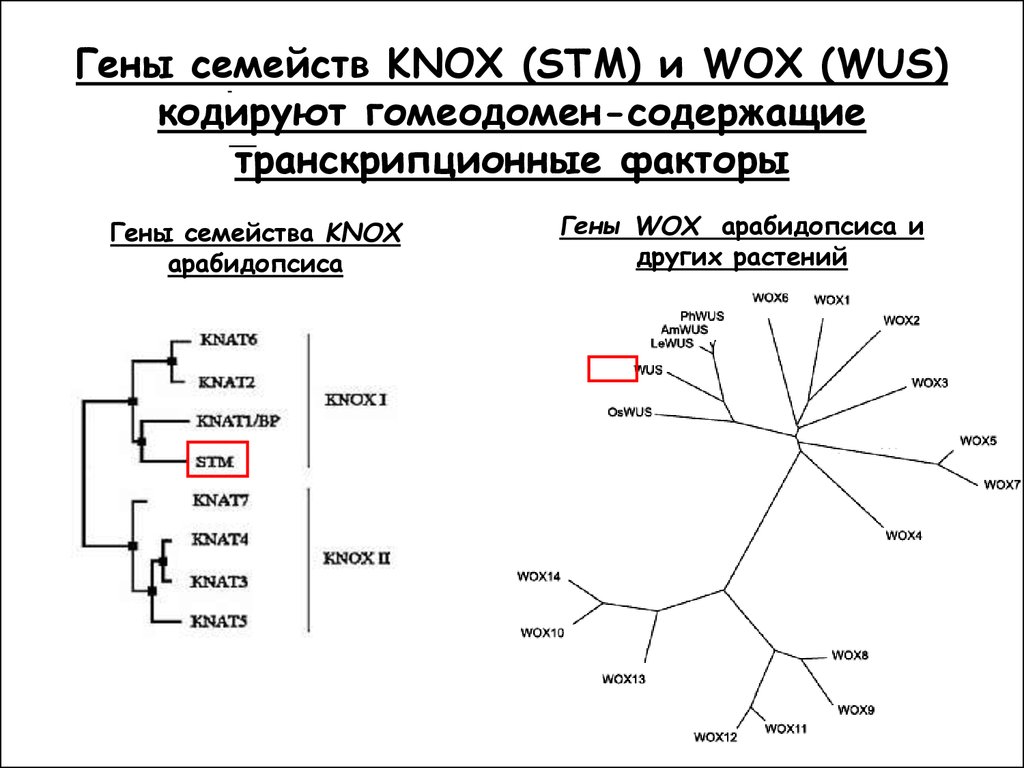
Гены семейств KNOX (STM) и WOX (WUS)кодируют гомеодомен-содержащие
транскрипционные факторы
Гены семейства KNOX
арабидопсиса
Гены WOX арабидопсиса и
других растений
31
31
32.
ТФ с гомеодоменами(семейство TALE)
KNOX
мишени: гены биосинтеза ЦК и ГК
WOX
вышележащие
регуляторы:
система CLAVATA
мишени: гены ARR-A -репрессоры
ответа на ЦК (для WUS)
программы развития:
2). Развитие сложного
1). Развитие ПАМ
листа
программы развития:
1). Идентичность зародыша и
суспензора (WOX2 и WOX8)
stm
WOX8
HD-ZIPIII
WOX2
35S::
KNAT1
мишени: 1). гены ТФ KANADY
(антагонисты HD-ZIPIII) 2). гены PIN
(регулируют транспорт ауксинов)
2). Идентичность ПАМ и КАМ
(WUS и WOX5)
WUS
WT
программы
развития:
WOX5
WT
phb
Адаксиальноабаксиальная
симметрия листа
33.
Ген KNOTTED1 был идентифи-цирован на основедоминантных мутаций kn1, вызывающих отсутствие лигулы
и появление узелков (knots) на листьях, особенно вокруг
жилок. В связи с доминантностью мутаций судить о функции
гена сложно
34.
Зная о важной роли гомеобоксных генов в развитии животных,приступили к клонированию и изучению KNOTTED1-подобных
генов (KNOX-гены knotted1-like homeobox-containing) в
растениях
По гомологии с KN1 клонированы другие гомеобоксные гены
кукурузы (более 13), A.thaliana (7), табака, томатов, сои, риса,
ячменя и др.
Класс II
Кукуруза
KNOX1
Кукуруза
KNOX2
KNOX6
Кукуруза
Кукуруза
KNOX7
B. napus
BNHD1
Другие
Резушка
ATH1
Человек
PNX-1
Helix I
Helix II
turn
Helix III
Экспрессируются
только в АМ побега
Класс I
Кукуруза
KN1
Рис
OSH1
Кукуруза
RS1
Кукуруза
KNOX4
Резушка
KNAT1
Кукуруза
KNOX8
Кукуруза
KNOX3
Соя
SBH1
Резушка
KNAT2
Кукуруза KNOX10
Кукуруза
LG3
Кукуруза
KNOX5
Кукуруза KNOX11
35.
Сверхэкспрессия гена KNAT1 у арабидопсисаНа листьях возникали
дополнительные
меристематические очаги, из
которых могли возникать
почки и побеги
Листья A. thaliana 35S::KNAT1
превращаются из простых в
пальчаторассеченные, хотя фенотип
сильно варьировал (Chuck, Linkoln,
Hake, 1996)
36.
Сверхэкспрессия гена KNAT в листьяхтоматов
дикий тип
35S::KNAT
37.
KNOTTED-подобные гены – возможныеучастники морфологической эволюции
растений
38.
Обнаружение связи между уровнемэкспрессии KNOX-генов и усложнением
структуры листа позволило предположить,
что эти гены могли участвовать в
формировании разных типов листа
у видов с простым листом (арабидопсис, табак,
кукуруза, рис) KNOX-гены экспрессируются только в АМ,
но не в примордиях листьев;
у
томата,
имеющего
сложный
лист,
эти
гены
экспрессируются и в примордиях
листьев
39.
Структура белков, содержащих MADS-доменыN
C
57а/к
Ceratopteris (CRMl)
Antirrhinum (DEFICIENS)
Arabidopsis (AGL15) (Plants)
Consensus
MCM1 – регулятор транскрипции дрожжей
AG – гомеозисный ген арабидопсиса (класс
С)
DEF –гомеозисный ген львиного зева (класс
В)
SRF – регулятор транскрипции человека
39
40.
ТФ с MADS доменомSOC1, FLC , CAL
вышележащие регуляторы:
1). ТФ FT 2). Белки FCA и FY
2). Polycomb-комплекс VRN
мишени: ген ТФ LFY
программы развития: развитие
флоральной меристемы
AP1, AP3, PI, AG, SEP1,2,3
вышележащие регуляторы: ТФ LFY
мишени: ???
программы развития: развитие органов цветка
PHERES
вышележащие
регуляторы:
Polycomb-комплекс FIS
мишени: ???
программы
развития:
развитие зародыша и
эндосперма
41.
Исследования трансгенных растений с конститутивнойэкспрессией гомеозисных генов также доказывают верность
АВС-модели
35S::AP3
35S::PI
В
С
A
Л
35S::AP3
35S::PI
35S::AG
Л Т Т
В
С
Т
Т Т
Т
42.
Мутации в АВС- генах приводят к формирова-ниюцветков разной структуры за счет изменения характера
экспрессии этих генов (изменения доменов экспрессии в
результате изменения уровня экспрессии, нарушения
взаимодействий с другими регуляторными белками)
Можно предполагать, что именно изменения
характера экспрессии консервативных генов ортологов
АВС лежат в основе всего разнообразия форм цветка
покрытосеменных
В
Л
A
Л
Т
С
П
Цветки с двойным
венчиком
(лилейные)
Цветки с двойной
чашечкой
В
Ч
A
С
Ч
Т
П
43.
Использование методов«генетической хирургии» для
изучения взаимодействия клеток и
тканей в процессах развития
44.
Day, Galgoci, Irish, 1995.Генетическое удаление лепестков и
тычинок для выяснения роли клеточных
взаимодействий в развитии цветка
Модели, описывающие процесс возникновения органов
цветка:
«Последовательная» модель
Wardlow, 1957.
Порядок возникновения органов
цветка отражает
последовательность
индуктивных сигналов (развитие
органов каждого последующего
круга зависит от сигналов,
поступающих из предыдущих
кругов
45.
«Пространственная» модель Holder, 1979.Под влиянием позиционных сигналов
происходит разметка ФМ, после которой идет
независимая дифференцировка органов цветка
46.
В основе «генетической хирургии» лежитметод трансформации растений химерными
генами следующей конструкции:
1. Кодирующая часть гена – последовательность,
кодирующая
продукт,
вызывающий
гибель
растительных клеток и не способный перемещаться в
другие клетки:
а) ген Corynebacterium diphtheriae, кодирующий
дифтерийный
токсин
DT-A-белок,
который
катализирует
АДФ-рибозилирование
фактора
элонгации. Это ведет к ингибированию белкового
синтеза и гибели клеток. В состав DT-A-белка не
входит сигнальный пептид, белок не является
секреторным, поэтому вызывает гибель только тех
клеток, где ген экспрессируется;
47.
b. гены, кодирующие рибонуклеазы: Т1 рибонуклеазуAspergillus oryzae и барназу Bacillus amyloliquefaciens,
вызывающие внутриклеточную деградацию РНК;
c. ген pehA из Burkholderia caryophilla PG2982 –
бактерии, метаболизирующей глицерол-глифосат до
глифосата
Глицерил-глифосат
Гидролиз
Глицерол + глифосат
фосфонат-моноэстераза
Глифосат – гербицид,
ингибирующий синтез
ароматических аминокислот
путем связывания с EPSP.
Токсичен для растений, E. сoli
и др.
O
O
O-
P
CH2
NH
CH2
C
O-
Глифосат
(N-[фосфонометил]глицин)
O-
48.
Целью работы Day, Galgoci, Irish (1995) былапроверка существующих моделей с использованием
метода генетической хирургии
Объекты: Arabidopsis thaliana и Nicotiana tabacum
1.7 т.п.н.
5’
3’
AP3
DT-A
NOS
1.7 т.п.н.
5’
3’
AP3
uidA
NOS
49.
Этапы работы1. Слитые гены включали в Т-ДНК область
(фрагмент Ti-плазмиды,который передается
растению) бинарного вектора pBIADN и
клонировали в E.coli
Слитый ген
ori
NPTII – ген устойчивости к Km
-прямые концевые повторы,
фланкирующие Т-ДНК
50.
РЕЗУЛЬТАТЫ:1. У резушки получено 5 трансгенных растений, в
геноме которых присутствовали от 1 до 5 копий
трансгенов (Саузерн-блоттинг)
2. Все трансгенные растения имели одинаковый
фенотип:органы 2 и 3 кругов (лепестки и
тычинки) отсутствовали, а чашелистики и
пестики имели нормальную морфологию.
Следовательно, разметка кругов начинается на
ранних стадиях до экспрессии трансгена во
ФМ, т.е. верна пространственная модель
51.
Использование трансформации дляизучения роли гормонов в развитиии
растений
52.
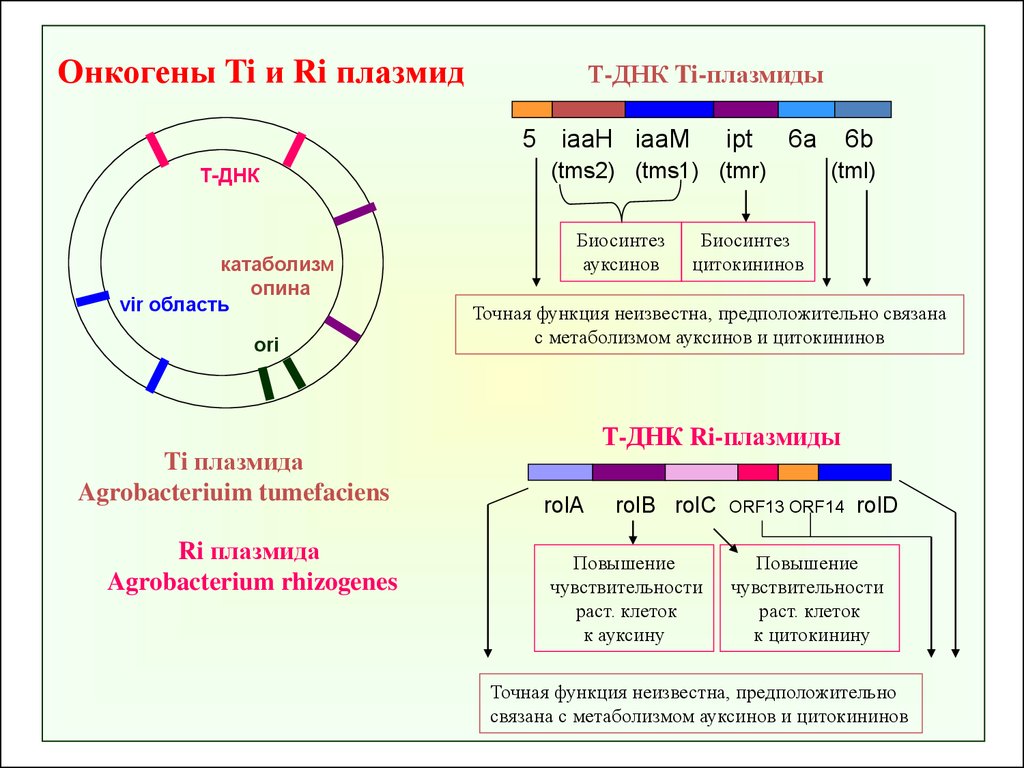
Онкогены Ti и Ri плазмидТ-ДНК Ti-плазмиды
5 iaaH iaaM
Т-ДНК
катаболизм
опина
vir область
ori
Ti плазмида
Agrobacteriuim tumefaciens
Ri плазмида
Agrobacterium rhizogenes
ipt
6a
(tms2) (tms1) (tmr)
Биосинтез
ауксинов
6b
(tml)
Биосинтез
цитокининов
Точная функция неизвестна, предположительно связана
с метаболизмом ауксинов и цитокининов
Т-ДНК Ri-плазмиды
rolA
rolB rolC
Повышение
чувствительности
раст. клеток
к ауксину
ORF13 ORF14
rolD
Повышение
чувствительности
раст. клеток
к цитокинину
Точная функция неизвестна, предположительно
связана с метаболизмом ауксинов и цитокининов
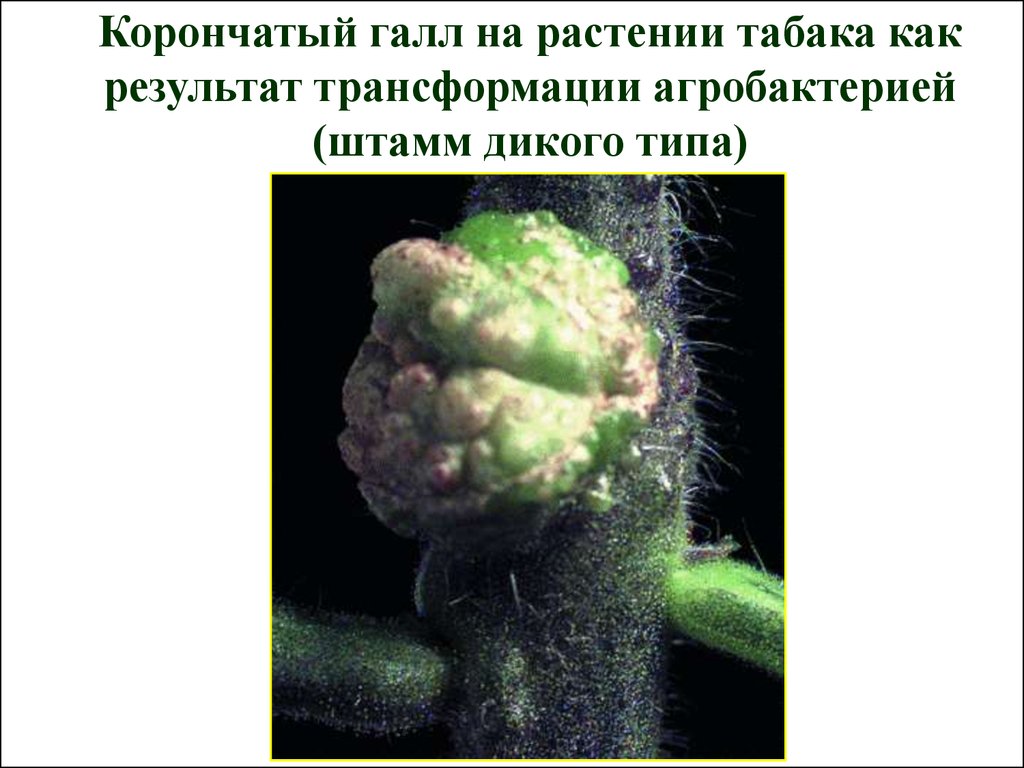
53.
Корончатый галл на растении табака какрезультат трансформации агробактерией
(штамм дикого типа)
54.
Трансгенные рстенияс геном tmr
55.
Ген tmr в трансгенных растениях,экспрессирующийся в плодах (промотор 2А11)
56.
Опухолеобразование у редиса – генетический признакГибрид Опухоль + Опухоль - 2 (1:3)
18х10*
9
15
2,00
18х16*
6
19
0,01
18х19*
67
171
1,26
18х21*
11
50
1,41
6х10*
14
46
0,09
8х16*
12
24
1,33
57.
Фенокопии опухолеобразования у трансгенныхрастений с геном ipt
на корнеплоде интактных
растений
ipt
nptII
у растений
in vitro на среде
с БАП
58. Прямая генетика: фенотип ген Обратная генетика: ген фенотип
Прямая и обратная генетика в изучениифункции гена
Прямая генетика:
фенотип
ген
Обратная генетика:
ген
фенотип
59. - гомологичная рекомбинация/замещение гена -замещение гена: альтернативные подходы (Zn-fingers nucleases, TALEN) - РНК-интерференция/ генный сайленсинг
Подходы, используемые обратной генетикой:- гомологичная рекомбинация/замещение гена
-замещение гена: альтернативные подходы (Zn-fingers nucleases,
TALEN)
- РНК-интерференция/ генный сайленсинг
- T-ДНК-инсерционный мутагенез/T-DNA tagging
- TILLING (Target Induced Local Leisons IN Genomes)
60.
TALE: TAL (transcription activator-like) effectorsЭффекторы TALE –
регуляторные белки,
секретируются бактериями
Xanthomonas (патогены
растений) с помощью
механизма секреции III типа.
TALE – оказывают влияние на
экспрессию генов растенияхозяина, тем самым
обуславливая
восприимчивость или
устойчивость к заболеванию
Kay et al.
AvrBs3 – связывается с UPA-box,
активируют гены-мишени – в т.ч. Bs3,
запускающий реакцию СВЧ
61.
Основные типы замолкания гена (сайленсинга)пост-транскрипционный сайленсинг
(регуляция экспресии гена на уровне
трансляции)
PTGS - Post-transcriptional gene silencing
Специфическое
разрезание мРНК (А)
транскрипционный сайленсинг
(регуляция экспрессии гена на
уровне транскрипции)
TGS - Transcriptional gene silencing
Репрессия трансляции (В)
Метилирование гистонов,
гетерохроматинизация (С)
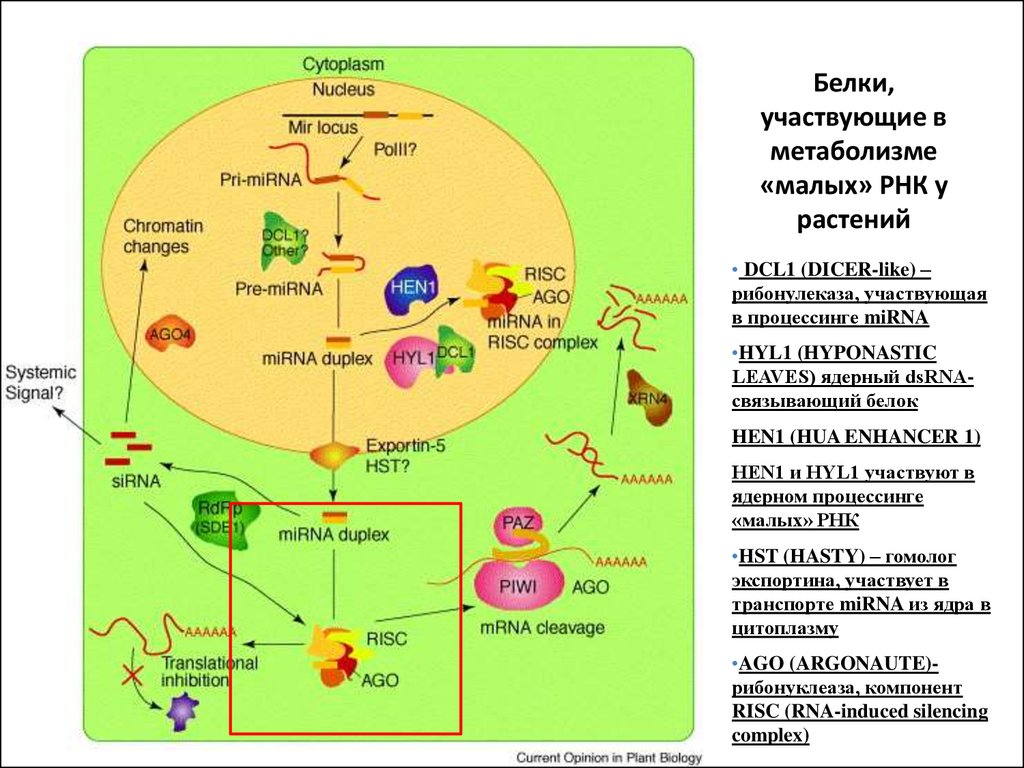
62. Белки, участвующие в метаболизме «малых» РНК у растений
• DCL1 (DICER-like) –рибонулеказа, участвующая
в процессинге miRNA
•HYL1 (HYPONASTIC
LEAVES) ядерный dsRNAсвязывающий белок
HEN1 (HUA ENHANCER 1)
HEN1 и HYL1 участвуют в
ядерном процессинге
«малых» РНК
•HST (HASTY) – гомолог
экспортина, участвует в
транспорте miRNA из ядра в
цитоплазму
•AGO (ARGONAUTE)рибонуклеаза, компонент
RISC (RNA-induced silencing
complex)
63.
Пример: выключение (сайленсинг) гена «икс»Конструкция, используемая
для трансформации
промотор
конститутивный
(35S)
Интроны
икс
регулируемый
(индуцибельный)
ски
dsRNA
DICER-подобная рибонуклеаза
RISC –
RNA-induced
silencing complex
деградация транскрипта,
репрессия трансляции
mRNA
64. Регулируемое замолкание гена PDS (PHYTOENE DEASATURASE)
PDSPHYTOENEDEASATURASE
Нет ИНДУКТОРА
pHELLSTOP
PDS
intron
b-глюкоронидаза
35S
PDS
op
RB
RB
LhGR
GUS
Nos
LB
LB
NPTII
оператор
Неактивный ТФ
LhGR – транскрипционный фактор (ТФ)
HSP90
Добавление ИНДУКТОРА: + DEX дексаметазон (стероидный лиганд)
Экспрессия
b-глюкоронидазы
(репортерного гена)
Экспрессия
PDS- ингибирующей конструкции
PDS
Сайленсинг
гена PDS
RB
RB
intron
35S
PDS
op
Активный ТФ
LhGR
GUS
DEX
Nos
LhGR
NPTII
LB
LB
65. Регулируемое замолкание (индуцибельный сайленсинг) гена PDS (PHYTOENE DEASATURASE)
No DexDex
66.
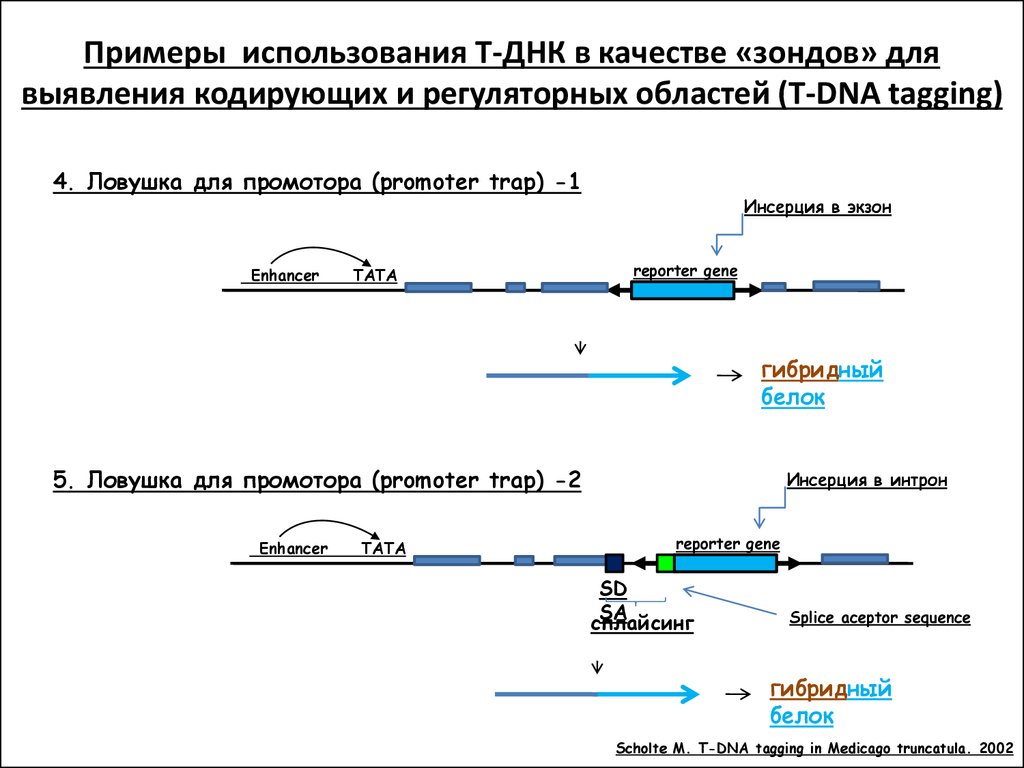
Примеры использования Т-ДНК в качестве «зондов» длявыявления кодирующих и регуляторных областей (Т-DNA tagging)
1. Геномная ДНК дикого типа
Enhancer
TATA
2. Activation tag (активирующая последовательность)
En En En En
Enhancer
TATA
3. Enhancer trap (ловушка для энхансера)
Enhancer
TATA
TATA
reporter gene
Scholte M. T-DNA tagging in Medicago truncatula. 2002
67.
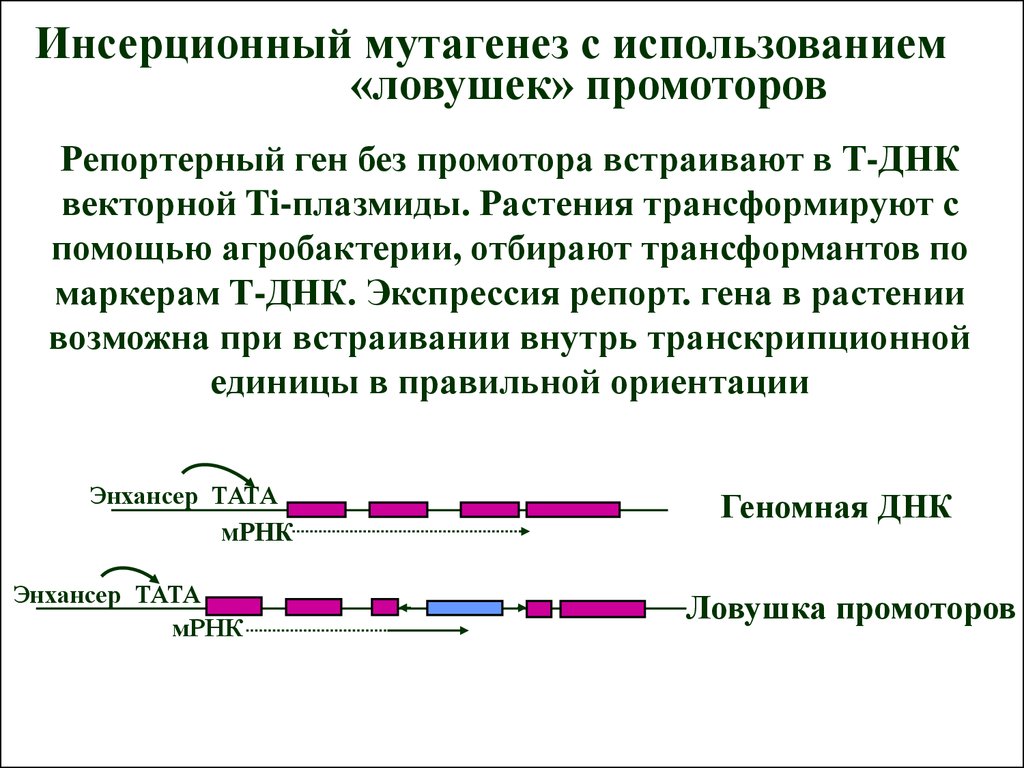
Инсерционный мутагенез с использованием«ловушек» промоторов
Репортерный ген без промотора встраивают в Т-ДНК
векторной Ti-плазмиды. Растения трансформируют с
помощью агробактерии, отбирают трансформантов по
маркерам Т-ДНК. Экспрессия репорт. гена в растении
возможна при встраивании внутрь транскрипционной
единицы в правильной ориентации
Энхансер ТАТА
мРНК
Энхансер ТАТА
мРНК
Геномная ДНК
Ловушка промоторов
68.
Примеры использования Т-ДНК в качестве «зондов» длявыявления кодирующих и регуляторных областей (Т-DNA tagging)
4. Ловушка для промотора (promoter trap) -1
Enhancer
TATA
Инсерция в экзон
reporter gene
гибридный
белок
5. Ловушка для промотора (promoter trap) -2
Enhancer
TATA
Инсерция в интрон
reporter gene
SD
SA
сплайсинг
Splice aceptor sequence
гибридный
белок
Scholte M. T-DNA tagging in Medicago truncatula. 2002
69.
Примерыиспользования Т-ДНК в
качестве «зондов» для
выявления
тканеспецифичных
энхансеров
Принцип работы
«ловушки для
энхансеров»
GAL4:GFP
70. Методы редактирования генома растений
71. Модификации генома: разновидности
Изменение случайногоместа
в геноме
Редактирование
генома
72. Редактирование генома
Инактивация конкретноголокуса
Замена одного локуса на
другой с помощью
гомологичной
рекомбинации (HR)
Этот путь затруднён у
Чтобы инактивировать
растений.
конкретный локус, надо
Введение DSB также
осуществить в нём разрыв
существенно увеличивает
двунитевой ДНК (DSB)
вероятность гомологичной
рекомбинации
73. Программируемые нуклеазы
• ZFN (Zink-Finger Nucleases)• TALENs (Transcription Activator-Like Effector Nucleases)
• CRISPR-Cas система (Clustered regularly Interspaced
Palindromic Repeats- CRISPR associated protein)
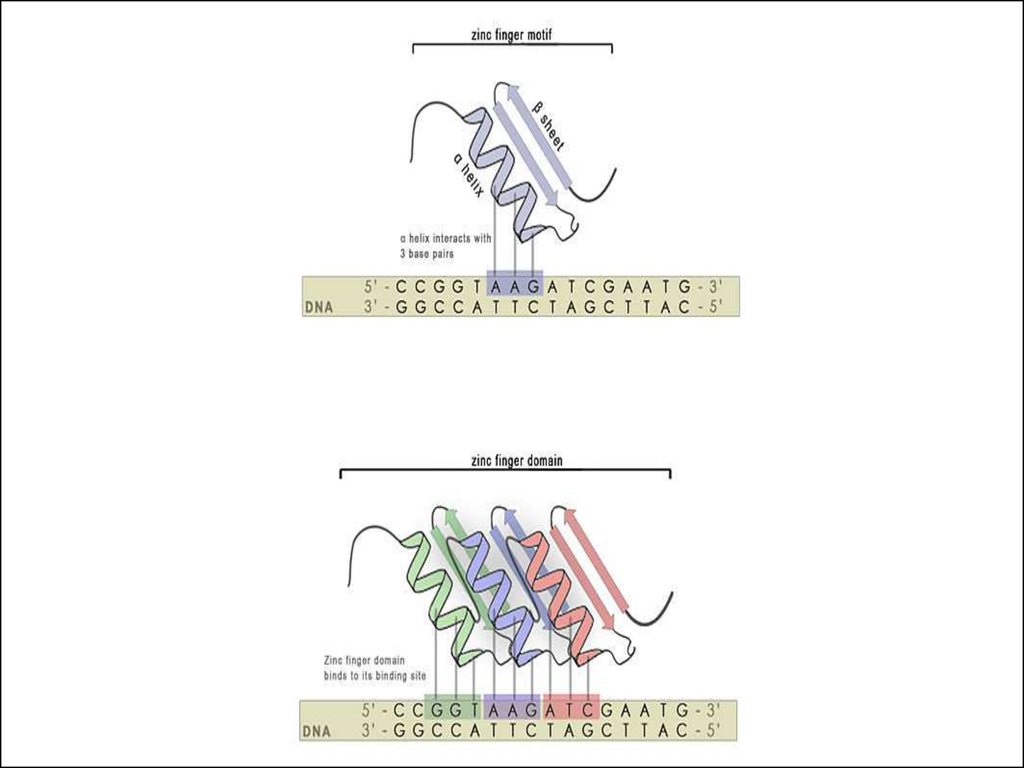
74. Нуклеазы с цинковыми пальцами
В качестве ДНКсвязывающегодомена используется
универсальный домен
типа «Цинковые
пальцы»
75.
76. Как программируют ZFN?
• Каждый цинковый палецраспознаёт 3 (4) нуклеотида.
Обычно используют 3-6 пальцев.
• Можно подбирать цинковые
пальцы под последовательности
• Обычно хорошо режутся не все
последовательности, а только GNN.
(т.е., например, 9-нуклеотидная
последовательность должна быть
GNNGNNGNN)
• В среднем хорошая специфичная
последовательность для разрезания
попадается один раз на 100 нп.
Wolfe et al., 1999
77.
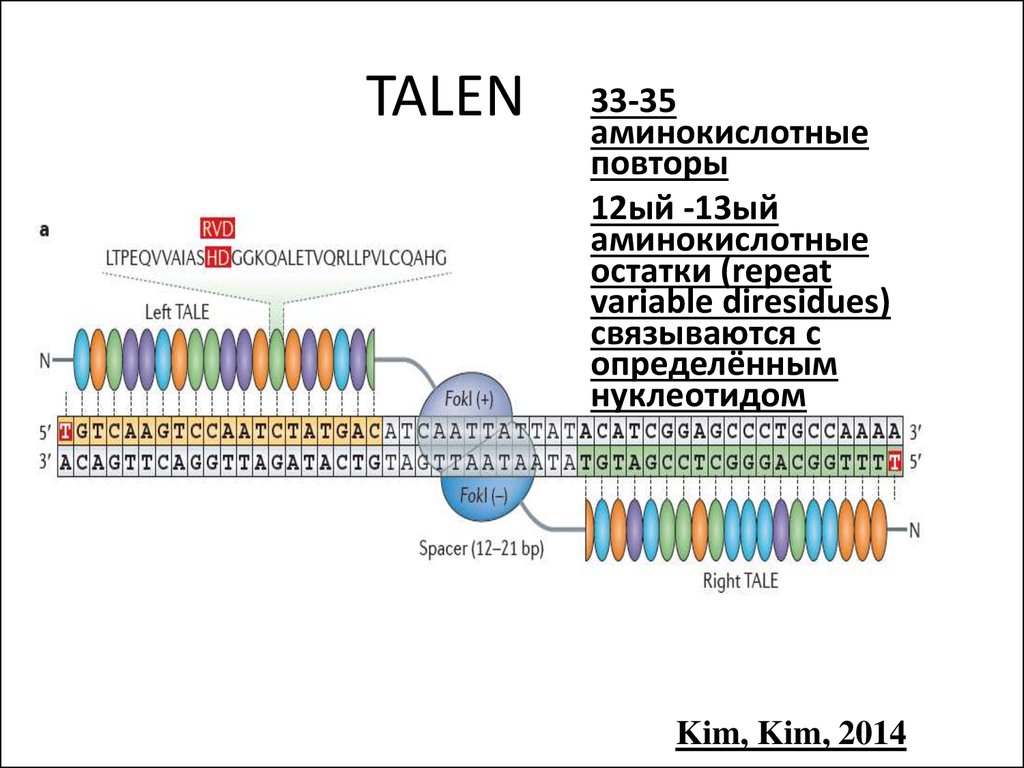
TALEN (Transcription Activator-LikeEffectors Nucleases)
78. TALEN
33-35аминокислотные
повторы
12ый -13ый
аминокислотные
остатки (repeat
variable diresidues)
связываются с
определённым
нуклеотидом
Kim, Kim, 2014
79. Применение TALEN
Потеря функциигена LOX3
(кодирует
липоксигеназу,
создающую
перекись
водорода)
увеличивает сроки
хранения семян
Этот ген
инактивировали с
помощью TALEN
Рис дикого
типа
Рис lox3
После обработки для
ускоренного старения
Ma et al., 2015
80. Система CRISPR/Cas9 (Clustered regularly Interspaced Palindromic Repeats- CRISPR associated protein)
81. Система CRISPR/Cas9
• В 2005 обнаружили, что спейсеры гомологичныпоследовательностям фагов и конъюгативных
плазмид (Mojica et al., 2005)
• Streptococcus thermophilus: Устойчивость к
фагам коррелировала с составом спейсеров
(Barrangou et al., 2007)
• Гены cas тоже необходимы для устойчивости
(Barrangou et al., 2007)
82. Работа системы CRISPR-Cas в бактериях
Horvath,2010
Barrangou,
83.
Čermákal., 2015
et
84. Защита от вирусов с помощью CRISPR-Cas9
Ji et al., 201585.
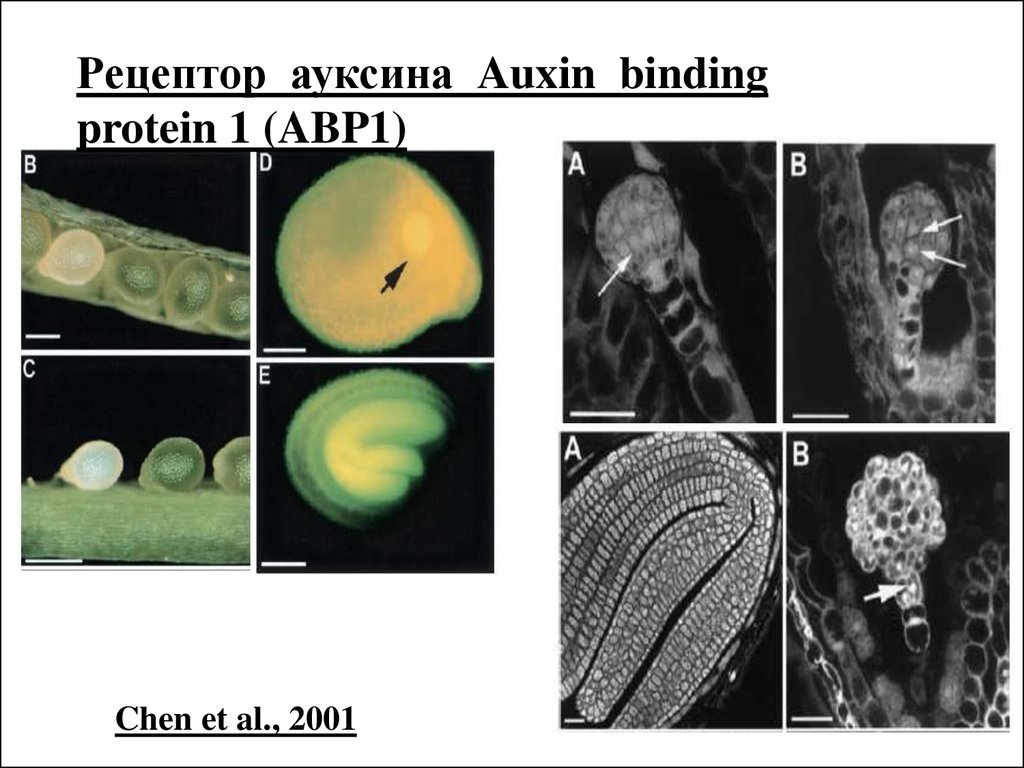
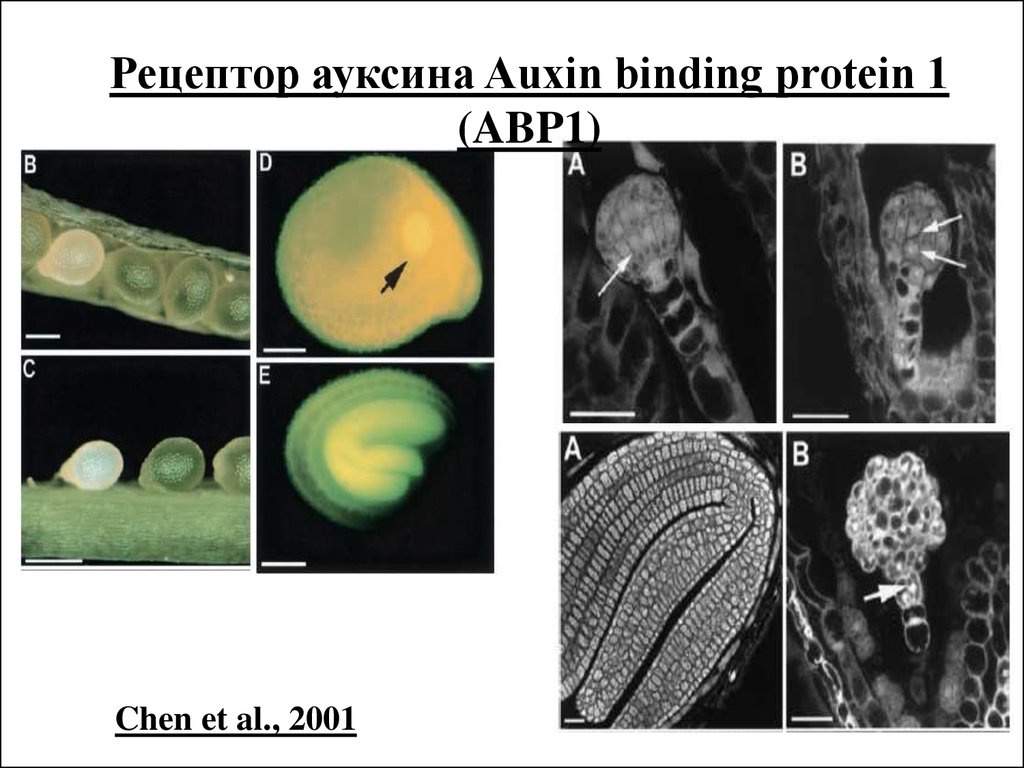
Рецептор ауксина Auxin bindingprotein 1 (ABP1)
Chen et al., 2001
86.
Gao et al.,87.
Рецептор ауксина Auxin binding protein 1(ABP1)
Chen et al., 2001






































































 Биология
Биология








